点击蓝字 关注我们
从机理到应用:几何深度学习解密光控反硝化微生物组

iMeta主页:http://www.imeta.science
研究论文
● 原文链接DOI: https://doi.org/10.1002/imt2.162
● 2024年1月6日,清华大学刘锐平团队在iMeta在线联合发表了题为 “From mechanism to application: Decryptinglight‐regulated denitrifying microbiome throughgeometric deep learning” 的文章。
● 本研究展示了图神经网络(GNNs)结合光遗传学在调控反硝化和加速微生物组新知识发现的潜力,从而赋能更深入的研究和多重应用。
● 第一作者:廖洋
● 通讯作者:刘锐平(rpliu@tsinghua.edu.cn)
● 合作作者:赵婧、边继踊、张子威、徐斯琪、秦一鉴、苗时雨、李瑞、张梦、朱文武、刘会娟、曲久辉
● 主要单位:清华大学水质与水生态研究中心、清华大学计算机科学与技术系、北京航空航天大学电子信息工程学院
亮 点
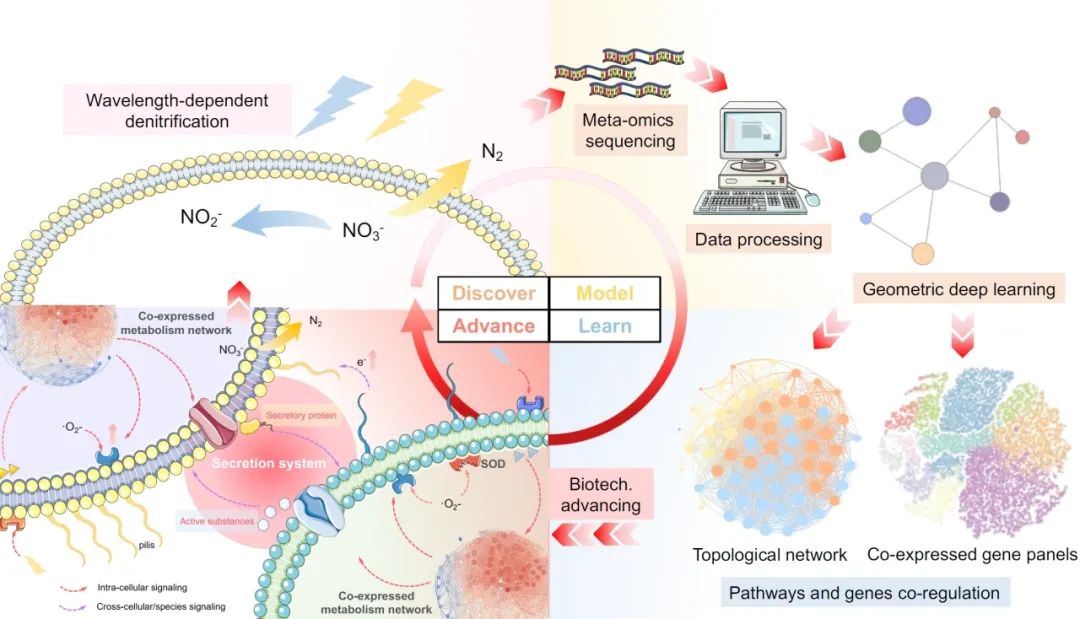
● 基于图神经网络(GNNs)且融入生物领域知识的计算框架,在识别共表达基因面板、解密波长依赖性反硝化上表现出优异的性能;
● 湿实验室验证了GNNs揭秘的知识,包括波长特异的分泌系统和硝酸盐-超氧自由基共调控现象,并可用于硝酸盐去除和资源回收;
● 本研究开发了共表达基因面板和拓扑网络工具包,从而实现加速新科学知识的发现和多功能生物技术的开发。
摘 要
对反硝化微菌群的调控对可持续的工业生物技术和生态氮循环至关重要。宏组学提供了微生物群落的全面遗传图谱,然而面对高度复杂的菌群及其宏组学数据集,精准解密生物数据和进一步应用仍然充满挑战。本研究将光遗传学和几何深度学习相结合,展示了反硝化菌群智能解密并提出调控策略的“发现-建模-学习-迭代(DMLA)”循环范式。图神经网络(GNNs)在整合生物学知识和鉴定共表达基因组方面表现出优异的性能,可用于预测未知表型、阐明分子生物学机制和推进生物技术。通过DMLA循环,揭秘了具备波长特异响应的分泌系统以及硝酸盐-超氧化物的共调控,实现了将胞外蛋白产量提高83.8%、硝酸盐去除率提高99.9%。该研究展示了GNNs结合光遗传学在调控反硝化和加速微生物组新知识发现的潜力,从而赋能更深入的研究和多重应用。
视频解读
Bilibili:https://www.bilibili.com/video/BV1P6421u7NE/
Youtube:https://youtu.be/lQZyxLIj-QQ
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
反硝化菌群对维持生态系统氮循环至关重要,主要包括反硝化将硝酸盐和亚硝酸盐还原为氮气、以及将硝酸盐转化为氨氮和有机氮。近年来地球氮边界引起广泛关注,据估计已达到生态高风险区。此外,反硝化菌群在社会经济发展中也发挥着关键作用,如食品生产、能源、废水处理和资源回收。例如,由于硝酸盐对人类和水生生物的毒性,反硝化脱氮被广泛用于硝酸盐去除,这在工业生产中占据了相当大的成本。因此,人们提出了各种方法来调节优化反硝化过程,包括添加导电材料、施加磁场和光照、适当的生物反应器设计和操作等。在这些策略中,光学技术因其特点极具优势而备受青睐,包括低成本、环境友好、太阳能转化为化学能、选择性和可精确控制微生物。这是因为光可以作为光遗传学开关的信号来调节细胞活动,如光敏启动子、离子通道、生物泵和蛋白质构象变化。最近,我们成功地利用光波长双向调节反硝化污泥,用于多种高效低成本的硝酸盐去除过程。
对于所有的调控策略,宏组学已经成为提供微生物组遗传信息的有效方法,包括物种、基因、蛋白质、代谢途径、物种等高维数据。然而,解密生物大数据需要高超的技能和高度专业的生物知识。此外,搜索生物功能十分费力、且其选择往往是主观的,导致很多研究提出的机理很难在湿实验室和放大实验中得到验证。此外,传统的方法通常侧重于单个基因或酶,难以捕捉在多个层面上系统性相关的动态生物网络,如遗传、代谢和细胞层面。而环境菌群更是具有高度复杂和跨物种相互作用的特点,因此其解析尤其充满挑战。
深度学习在表征生物大数据、学习遗传拓扑和共表达原理方面表现出优异的性能。其中,多使用多层感知(MLP)、卷积神经网络(CNN)和长短期记忆(LSTM)。然而,这些方法无法详细说明生物系统的多级拓扑信息,也仅限于欧几里得数据集。最近,几何深度学习因其在非欧几里得数据集中的强大表示能力而受到极大关注,其中图神经网络(GNN)是利用异构信息和复杂拓扑关系最具代表性的方法。
本研究展示了采用几何深度学习来解密环境菌群的宏组学数据集。在实验室中发现光调控反硝化作用后,我们通过DMLA(发现-建模-学习-迭代)循环加深我们对光遗传学机制的理解并推进其应用。在建模阶段,我们利用GNNs和Deep Graph Infomax(DGI,一种用于图结构数据集的无监督深度学习算法)将基因表达和亚细胞信息整合以识别基因面板。在学习阶段开发的工具包的指导下,我们扩展了光学生物技术的应用,包括增加细胞外蛋白质产量(高83.3%)和回收光诱导的生物活性物质。此外,超氧化物的信号传导作用使硝酸盐去除率提高了99.9%。迭代阶段解释了内在机理,不同的自催化效应表明分泌系统在波长依赖的光控氮代谢中发挥着重要作用。代谢通路富集和拓扑分析表明,黄光集中了代谢流以合成更多的蛋白质,如菌毛和分子伴侣。相反,蓝光分散了代谢流从而能分泌罕见的生物活性物质。具有不同频率的光子、光诱导的第二信使和超氧化物可以作为促进物质交换和集体细胞代谢的信号。除了反硝化菌群,我们还将应用扩展到其他微生物组和实际工程菌群。我们的工作为微生物组解密提供了一个框架,并展示了GNN赋能的光学生物科技在调控生物氮代谢中的潜力。
结 果
光控生物代谢的发现与图神经网络建模宏组学
阳光抑制对环境微生物群普遍存在,阻碍了废水中硝酸盐的去除。在湿实验室中我们发现单波长光对活性污泥的影响各不相同(图2A)。我们采用峰值在456.2 nm的LED蓝光和峰值在589.4 nm LED黄光(图S1)来调控水生反硝化微生物组。总的来说,蓝光和黄光分解了太阳光的抑制作用。蓝光对微生物代谢具有抑制作用,在26小时时实现部分反硝化(PD),亚硝酸盐积累率(NAR)为69.4%,远高于黑暗(33.6%)。稳定的亚硝酸盐积累有利于部分脱氮-厌氧氨氧化(PD/A),与传统脱氮相比,这是一种更经济的含氮废水处理方法。相反,黄光促进了硝酸盐和亚硝酸盐的去除。有趣的是,与黑暗相比,黄光也促进了49.5%的碳源摄入,但生物量合成没有显著增加,这表明微生物群利用乙酸盐进行其他代谢。相比之下,与黑暗相比,蓝光减少了27.8%的生物量积累,但碳源摄入没有显著减少,这也意味着代谢通量从生物量合成中转移。细菌活力染色显示,与黑暗和紫外组相比,蓝光组和黄光组的代谢均被激活(图S2),进一步支持代谢重定向。
为了揭示微生物对光波长的转录反应,我们在光脱氮后进行了宏转录组学研究,所有样本共56991个非冗余基因。首先,我们进行数据预处理,获得25886个有效的差异表达基因(DEGs)。DESeq分析、降维和对基因表达模式的分层聚类(图S3)表明蓝光比黄光触发了更显著的转录活动变化,分散了代谢留(文本S1)。这是因为大量的基因表达可响应蓝光,例如编码光感受器、启动子和增强子的基因。由于鲜有基因可响应黄光,因此黄光对较小的基因集表现出更高的选择性。此外, tSNE降维分析中DEG的重叠部分也证明了遗传共表达。
鉴于先前基于知识的数据库分类未能获得共同表达的基因面板(图S4),我们在图结构数据集上采用几何深度学习来构建上下文定制模型(图2B)。与广泛使用的单细胞数据集相比,宏组学数据集的特点是微生物相互作用以及频繁的细胞外和细胞内物质变化,因此在环境数据噪声的情况下,很难简单地通过线性回归或传统的机器学习来解密。因此,我们通过图卷积网络(GCNs)整合了生物知识,以帮助模型在无监督的环境微生物群监管网络中学习,释放出自然代码库的巨大潜力。经过数据处理,我们获得了有效的DEG,其亚细胞信息和表达信息被工程化为图结构数据集。表达信息表征了细胞内调控网络,而亚细胞信息(包括信号肽和跨膜结构域)代表了细胞间的相互作用。我们使用DGI算法来无监督地学习节点嵌入,然后对其进行聚类以获得基因面板。

图 2. 通过几何深度学习建模湿实验发现的工作流概述
(A)微生物组的光调节氮代谢、碳代谢和生物质合成的湿实验室发现。*: p值<0.05,**: p值<0.01,ns:不显著。红色箭头突出了文中所述比较。乙酸盐消耗和生物量的显著性分析结果取于光控脱氮结束时(24小时);(B) 通过GNN鉴定共表达基因组的工作流程。在数据预处理中,首先通过DESeq分析获得差异表达基因(DEGs),然后过滤低表达基因以获得有效的DEGs。降维分析中的重叠部分表示蓝光和黄光数据集共享的DEGs。在构建图模型时,我们用1和0来表征亚细胞信息,以表示单个基因是否编码信号肽并具有跨膜结构域。对有效DEG的表达水平进行对数归一化以表征基因表达信息。在模型训练中,我们采用图卷积网络(GCN),并利用Deep Graph Infomax(DGI)算法进行无监督学习以获得节点嵌入。

总结矢量。随后对嵌入的基因节点进行聚类,获得共表达基因面板用于机理解析、表型预测和生物技术开发。
几何深度学习在基因面板识别中取得了卓越的效果
共表达基因组数量的确定需要依赖于上下文生物学知识。案例中光波长双向调节硝酸盐转化意味着其与光信号传导相关的基因(即光转导)共表达,类似的光响应基因表达模式进一步证实了这点(图S6A)。相比之下,中枢代谢途径(如氮代谢)涉及多种功能,呈现不同的表达模式,因此不共表达(图S6B)。因此,我们比较了光转导基因的聚类分配(图S7),并定义了7个共表达基因组(文本S2)。
同一基因组(即簇)中的基因在对蓝光和黄光的反应中受到基因共调控。为了从数学上评估遗传潜在空间中的基因协同调节识别能力,我们将节点嵌入的聚类结果投影到2D潜在空间上,以及其他常用方法在没有几何深度学习的情况下的降维结果(图3A,B)。层次聚类(HC)表现出强烈的偏向性,倾向于将大多数基因聚类为2个聚类,这表明在生物共表达捕获方面的性能较差。与HC相比,K-means和DGI在聚类共表达基因方面都表现良好。然而,K-means未能像2D分布所建议的那样整合异质信息。通过DGI的无监督预训练基于基因表达和亚细胞信息学习了清晰的基因面板(图3A,B中的Subcellular DGI)。相反,当仅对异质信息应用K-means时,一些基因未能聚集在一起并分散在潜在空间周围(图3A,B中的Subcellular K-means),表明其未能捕获光遗传学基因共调控。聚类间相似性指标剪影系数指数(SCI)以更高的分数进一步证实了这一结果(文本S2)。

图3. 模型聚类能力评估
(A-B)对蓝光(A)和黄光(B)数据集无监督学习结果的2D投影和聚类能力评估;通过二维tSNE空间中的聚类可视化进行定性评估,通过聚类间相似性指标Silhouette Coefficient Index(SCI)进行定量评估;‘Subcellular’表示整合了亚细胞信息的数据集;(C-D)与蓝光(C)和黄光(D)下的光转导相关基因的基因面板分配,纵坐标表示基因数,“_sub”表示整合了亚细胞信息;(E)不同聚类方法基于先验生物知识光响应途径的功能分配得分(FAS)。
关于生物意义的聚类分配准确性,无论聚类数量如何,DGI模型都优于K-means(图S7和图3C,D)。DGI倾向于将光转导基因聚类成1-2个类。相反,K-means则分散在多个聚类中的基因,这意味着DGI成功地整合了亚细胞信息并捕获了遗传共表达。为了定量评估生物功能匹配性,我们定义了功能分配评分(FAS)。基于先前的知识,我们比较了与光密切相关的途径的FAS,包括氧化应激和光遗传学开关(图3E)。通常,具有亚细胞信息的DGI具有更高的FAS,表明DGI优于K-means,亚细胞信息整合有助于识别生物功能(文本S2)。
通过中枢和信号基因面板中的不同代谢途径预测表型
通过DGI获得的簇就是共表达的基因面板,可用于解密遗传机制。我们开发了基因面板工具包以解密自然代码库赋能内在机制和生物技术的开发。在搜索注释数据库后,可以观察到大多数光转导基因被分配到同一基因组,蓝色光的簇5和黄色光的簇1(图4A,B)。相反,其他聚类方法(图2A,B)会将这些基因错误地分配到不同的聚类中,这进一步证实了我们模型的有效性。硝酸盐和亚硝酸盐相关基因(即PD基因)大多分配到蓝光的簇7、簇4,以及黄光的簇6、簇3、簇4。我们通过富集分析对这些聚类进行了比较(图S9)。我们采用外显子模型的每千碱基片段/百万映射片段(FPKM)值来量化基因表达水平。蓝光的簇4的基因包含更多的反硝化基因,并且具有高表达水平和低倍数变化的特征,黄光的簇3也是如此。这些簇为中枢基因面板(HGPs)。相反,受到的簇光转导基因与HGPs不同,表现出相对高的倍数变化和低表达。鉴于光转导的信号传导作用,这些簇被定义为信号基因面板(SGPs)。

图4. 解密中枢基因面板(HGPs)和信号基因面板(SGPs)以预测表型,并在湿实验室中得到验证
(A-B)蓝光(A)和黄光(B)的功能簇和显著差异的光响应反硝化基因的空间分布;(C-D)蓝光(C)和黄光(D)中枢基因面板的通路富集分析,表达量最高的途径及其所属的KEGG BRITE用相应的颜色和粗体突出显示,气泡大小分别表示在蓝光或黄光下的平均基因表达水平(FPKM)。以深色组为对照,计算变化倍数;(E) 不同光照条件下分层胞外聚合物(EPS)的蛋白质浓度。堆叠柱上细白线分成的三份表示三次重复。S-EPS:可溶性EPS;LB-EPS:松散结合的EPS;TB-EPS:紧密结合的EPS;(F) 自催化实验,将在暗、蓝光和黄光条件下的EPS加入到反硝化系统中。
HGP对应于微生物群的集体行为(即表型),可在实验室中进行表征并用于开发新的生物技术。黄光和蓝光的HGPs中都有衰老这一代谢途径(图4C,D),衰老是一种2级KEGG Brite,有助于细胞适应遗传和环境刺激并延长寿命(文本S3)。长寿调节途径是主要的衰老途径,其特征是具有高活性的氧化活性,会产生大量的活性氧(ROS),表明光控反硝化与ROS代谢共同表达。光诱导的ROS产生的表型得到了湿实验室验证(图S10),所有组的总ROS水平在硝酸盐还原阶段增加,在后期降低,与亚硝酸盐浓度呈相似趋势。此外,蓝光和黄光都促进了总ROS的产生,这是由于光照微生物群的光化学刺激。
信号转导是蓝光HGPs中另一个明显的Brite,包括FoxO信号传导和MAPK信号传导途径,这意味着蓝光照射下信号传导的主导作用。碳水化合物代谢和其他次生代谢产物的生物合成主导了代谢通量,尤其是肌醇磷酸代谢(490.85 FPKM),几乎是第二代谢通量的两倍。肌醇磷酸代谢是协调生长因子信号传导、能量稳态与营养吸收和利用的重要枢纽,这意味着微生物组产生的较高水平的信号物质调节硝酸盐的吸收和转化,有可能促进生存和竞争(文本S3)。相反,黄光的HGP由多种蛋白质合成途径主导(图4D),包括与遗传信息处理、代谢、信号传导和细胞过程等相关的蛋白质。
蓝光和黄光之间HGPs和SGPs的总体表达水平的比较揭示了代谢通量的重定向。黄光的HGP显著上调且远高于蓝光,表明黄光照射下,代谢通量主要转向HGP合成蛋白。相反,蓝光的SGP的平均表达水平(15.70 FPKM)远高于黄光的(3.66 FPKM)(表S4),这解释了图1中蓝光抑制脱氮和乙酸盐摄取。蓝光下的代谢通量被重定向到SGP,用于重要代谢产物的合成,如次级代谢产物和聚糖生物合成、辅因子和维生素的代谢(图S11)。这些代谢物是有价值的生物产品,具有多种生物功能,如能量代谢、细胞间信号传导、细胞抵抗和保护,以维持基本的细胞功能,在环境刺激下生存(文本S4)。值得注意的是,分泌系统在蓝光的SGP中显著表达(图S11)。而黄光中分泌系统被分配给了HGP(图4D),表明分泌系统在蓝光和黄光照射下的作用是不同的。总之,蓝光触发了生物活性物质的分泌,如次级代谢产物、辅因子和维生素,而黄光有助于蛋白质合成和分泌的增加。
为了验证分泌系统差异,我们在光脱氮后提取了微生物群的胞外聚合物(EPS)。如预测:黄光的总蛋白质浓度最高,与暗对照相比增加了83.8%(图4E),对应于图4D中与蛋白质合成相关的高表达途径,展现了黄光在以硝酸盐废水为底物的生物法蛋白质生产中具有潜力。此外,蓝光增加了紧密结合的EPS的比例,对应于胞外多糖的生物合成和半乳糖代谢的显著上调(图S11A),这可以促进生物膜的形成,以通过胞外多糖类(一种胞外碳水化合物聚合物)保护细胞。提取EPS作为生物催化剂证明了蓝光和黄光下不同的分泌物质,与黑暗组相比,提取的EPS可以提高了16.6%的硝酸盐去除(图4F),这与上述共表达模型一致,即蓝光下的微生物群分泌更多的生物活性物质以在光化学氧化应激下生存。
通过拓扑网络的代表性基因调节微生物组
在发现微生物组的未知潜力后,例如使用硝酸盐作为生物生产的底物,需要精确的调控策略来加强生物反应。传统的方法通常针对单个基因或途径,称为生物标志物。但这种方法忽略了生物的系统性(系统生物学),往往表现不佳。因此,我们将网络拓扑和代表性基因结合起来,构建用于拓扑网络以指导精确的基因调控策略。
为了获得可信的基因表示,我们在基因面板内进行模块化分类,以获得子网络和相应的代表性基因(数据集S5和S6),表达量最高的三个基因为代表性基因。黄光HGP中分子伴侣携带了很大一部分具有高表达水平的代表性基因(图S12),与途径富集分析中的活化蛋白合成(图4D)和湿实验室结果(图4E)一致,这进一步证明了黄光下,硝酸盐利用和蛋白合成共表达。蓝光中PD基因被分配到3类和0类(图5A、B和数据集S1)。编码亚硝酸盐还原酶(NirK)和硝酸盐/亚硝酸盐转运蛋白(NarK1)的基因可以由第3类代表性基因代表,包括编码肌肌醇-1-磷酸合成酶(MIPS)、一氧化氮还原酶亚基B(NorB)和异柠檬酸脱氢酶(IDH)。这些代表性基因涉及信号转导、氮代谢和能量生产(文本S5),与反硝化呼吸链的途径富集分析和生物学先验知识一致。此外,硝酸还原酶(NarG)属于0类,可由编码超氧化物歧化酶(SOD)和4-羟基四氢二磷胆碱合酶(DapA)的代表性基因表示,这两种酶都是抗氧化系统中的关键酶,主要参与超氧化物清除。与先前研究中SOD在确定微生物网络中的结构域作用一致。

图5. 利用基因拓扑网络的代表性基因制定硝酸盐还原的调控策略
(A) 蓝光HGP的基因拓扑网络。关于基因节点和标志基因的拓扑信息的细节总结在数据S1、S5中,加粗的红色字体突出显示了最高度表达的代表性基因。黑体突出显示了PD基因;(B) 蓝光HGP代表性基因的表达水平。背景颜色突出了关键反硝化基因所在模块类,不同背景颜色代表不同的模块类;(C) 第一阶段:硝酸盐还原。第二阶段:亚硝酸盐还原。第三阶段:无机氮耗尽;(D) 不同初始硝酸盐浓度下超级自由基生成水平;(E) 超级自由基投加实验。在时间点a时添加硝酸盐和超级自由基,时间点b用于计算超级自由基对硝酸盐去除效率的影响,对照组和添加超级自由基组均在黑暗条件下进行;(F) 添加超级自由基对硝酸盐去除率的影响。
如拓扑网络模型所示,NarG介导的硝酸盐还原与编码超氧化物相关酶的代表性基因共表达。因此,我们测定了光反硝化过程中不同阶段的超氧化物水平(图5C),包括硝酸盐还原(第1阶段)、亚硝酸盐还原(第2阶段)和氮耗竭(第3阶段)。结果表明,不同光照条件下超氧化物水平的变化与图2A中的硝酸盐去除性能一致,即超氧化物水平越高,硝酸盐去除活性越高。这些初步证实了拓扑网络模型预测的硝酸盐超氧化物共调控。梯度氮实验和硝酸盐存在实验(图5D和图S13)进一步巩固了硝酸盐还原和超氧化物产生之间的共表达关系。
总ROS(图S10A)和超氧化物产生(图5C)在不同光照条件下的不同趋势表明,超氧化物在光控硝酸盐代谢中发挥着关键作用。我们在黑暗、蓝光和黄光下进行了猝灭实验,以研究典型ROS的贡献,包括羟基自由基(·OH)、单线态氧(1O2)和超氧化物(·O2-)(图S14B)。这些结果进一步证明了超氧化物在所有组的硝酸盐去除中的主要作用,特别是对于超氧化物达到99.1%猝灭率的黄光。基于超氧化物和硝酸盐还原之间的共表达关系,我们开发了一种酶促超氧化物产生方法来促进硝酸盐的去除(图S14C,D)。可以观察到,在时间点a添加超氧化物后,试验组的硝酸盐去除率提高(图5E),并在时间点b实现了比对照组高99.9%的硝酸盐清除效率(图5F)。
光控反硝化的机理与潜力
除了表型预测和新生物技术的调控策略外,建模结果还使机制方案重建成为可能。在光反硝化的情况下,这些包括波长依赖性反硝化的分子生物学机制(文本S6)、硝酸盐超氧化物共调节(文本S7)和波长发散分泌系统(文本S8)。
总体而言,分泌系统是跨物种相互作用的核心(图1)。蓝光光感受器无处不在,并已在广泛的生物平台中实现,解释了蓝光在光反硝化中引发的代谢通量分散。合成了更多样化的代谢产物,通常是活性物质,如辅助因子和维生素。其中一些分泌分子对微生物群在光化学胁迫下维持体内平衡至关重要。这些代谢物是潜在的高价值资源,这为利用硝酸盐作为底物来节省高价值化学品生产成本的生物过程提供了线索。有趣的是,黄光具有显著的激活效果,而基于黄光的光遗传学开关鲜有报道,这可能是由于跨物种的相互作用。黄光触发的中心代谢通量主要用于蛋白质合成,尤其是菌毛相关蛋白质,这暗示着菌毛在加速种间电子转移以进行的集体功能代谢中起到了作用,例如黄光中枢基因面板中富集的萜类和聚酮代谢(图S11),这种新颖的光诱集体效应在生物生产、开发合成生物学模块和加深对生物环境响应机理认知方面具有巨大潜力。
讨 论
解锁自然代码库以赋能健康生态系统、清洁能源和更可持续的未来,过去几年也见证了这类基于自然的生物技术的繁荣。尽管合成生物学的设计-构建-学习-测试(DBLT)循环在加速生物技术进步方面表现出巨大的潜力,但大多数努力都集中在大肠杆菌等模型菌株上,而且仅限于提高效率和产量。而天然微生物组的解密一直是瓶颈。我们在这里展示的DMLA循环在通过光遗传学释放反硝化微生物群的力量方面表现出巨大的潜力(图6)。

图6. DMLA循环工作流解锁基于自然的技术迭代
图中 “发现”阶段描述了适用范围和理论基础;“建模”阶段阐述了技术核心需求,包括几何深度学习模型和生物数据集;“学习”阶段展示了应用软件套装可以提供的服务;最后一个阶段“迭代”展示了通过DMLA循环促进新生物技术的多种潜在应用。
在“发现”阶段,环境扰动是激活遗传过程以捕获动态调控网络所必需的。在这里,我们使用基于光遗传学的遗传扰动来触发通过光遗传学开关的信号传输因此,这些方法也可以应用于除反硝化菌的宏转录组学之外的其他微生物组和宏组学。更多的案例,包括同时固定CO2的反硝化微生物群和实用工程微生物群,以及使用指南可参考https://github.com/YoungeLiao/DMLA。光遗传学诱导的动态调控网络变化包含在输入数据集中,包括表达矩阵、数据库注释和组条件。
在“建模”阶段,生物拓扑原理即系统生物学将被模型自动捕获。几何深度学习在表征复杂拓扑关系和集成异构信息方面具有卓越的能力。此外,与其他机器学习方法相比,该框架可以克服数据噪声、小样本量和标记瓶颈。生物数据集(包括表达水平和亚细胞注释)被设计为节点特征,并在2D潜在空间中的基因表达距离被设计为边缘。通过无监督预训练整合异构知识以获得嵌入,然后对嵌入进行聚类以获得共表达的基因面板。
在“学习”阶段,应用软件套装释放自然代码库的潜力。基于基因面板,富集的途径可以揭示未知的表型。以标志性基因为特征的生物网络是阐明生物学机制和制定调控策略的有力工具。
基于上述过程,新的生物技术可以用于驯化和调控稀有微生物,提供多方面的应用,包括综合机理报告、新化学物质和代谢产物的发现、开发用于生物生产的新型酶、合成生物学个性化细胞工厂等。
先前的研究大多集中在预测单细胞数据中的基因调控网络,而天然遗传密码库在可持续生物技术方面蕴藏着巨大的潜力,仍然是待挖掘的自然宝藏。我们的研究展示了如何利用GNN来发现新的微生物能力并制定对自然微生物组的调控策略,为利用自然解决方案应对全球挑战开辟了一个领域,如氮污染缓解、二氧化碳捕获和利用等。尽管如此,要充分利用DMLA循环来释放自然潜力,在计算和生物都仍存在挑战。在计算方面,算力和建模过程是两大瓶颈。宏组学通常包括数百万基因,与生物医学和临床数据集相比,这一数字高出数百倍甚至更多。此外,传统的方法通常需要手动选择合适的模型架构和超参数,这既费力又昂贵。最近提出的自动机器学习(AutoML)和生成模型可能是有前景的方法。在生物学方面,有限的机理认知和缺乏基因注释是实现有效DMLA循环的挑战,这需要对生态学原理、系统生物学和量子生物学进行更深入的认识和湿实验室验证,如光捕获蛋白中的量子相干、群体感应和厌氧消化中的直接种间电子转移,从而更好建立与生物专家领域知识相适配的模型。此外,除了光波长策略之外,还需要更多的调控策略和相应的数据集来丰富模型,使其更加智能、精准和通用,从而能够解密自然原理,加速生物技术的发展。
方 法
光控平台、菌群和光控反硝化操作
光控制平台由锡箔屏蔽,以避免干扰外部光线。LED光带被构建在光控制平台的外壁中,以为平台中间的反应器提供周围的光场。光控制平台的底部是磁力搅拌器,以确保反应的充分混合。将LED灯珠无覆盖地嵌入灯带上,灯珠间距为50mm,灯带宽度为8mm,额定电压为4-6V,额定功率为3-5W/m,最终空间光场的光强度为2.0±0.5mW cm-2。通过使用不同的LED灯珠作为光源来控制光波长,通过灯带的长度来控制光强度,并且通过灯带灯珠在光控平台内壁上的对称分布来保证光场的均匀分布。通过光谱仪(OceanInsight,Maya2000Pro,USA)表征LED的光谱(图S1)。
数据预处理和数据集探索性分析
通过DESeq分析获得了对蓝光和黄光反应的差异表达基因,log2倍变化>1和p<0.05的基因被视为DEG。在R中对包括2D-PCA、3D-PCA和tSNE在内的这些基因进行降维分析。在tSNE分级中,首先使用对数归一化数据(见方程1),最大迭代为1000,θ为0.4,perplexity为20,verbose为false。在层次聚类(HC)中还进行了对数归一化和缩放。
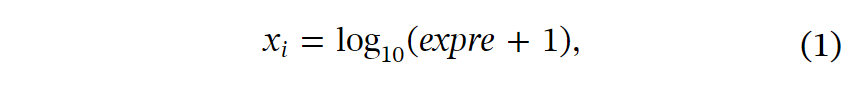
其中xi是对数归一化基因表达水平,expre是通过上述FPKM量化的基因表达水平。
为了通过多模式子数据集解读微生物组的转录组学反应,包括细胞内和细胞间相互作用的信息至关重要。因此,将基因表达信息和亚细胞位置信息作为互补的生物领域知识输入到我们的模型中。基因表达主要反映细胞内调控信号传导,亚细胞定位反映细胞间相互作用。为了更好地描述拓扑相互作用,采用几何深度学习并通过无监督学习整合基因表达信息和亚细胞位置信息。为了缓解建模中潜在的生物数据噪声,过滤平均表达<1的低表达DEG,以获得有效的DEG。考虑到去噪,我们将有效的DEG分为两个数据集,即蓝光DEG(25277个基因)和黄光DEG(4366个基因)进行建模。
图的构建
根据上述数据预处理,构建的图数据分别具有25277个节点和4366个节点,用于蓝光和黄光,其中每个节点对应于一个基因,并具有8个节点特征,包括6列表示表达信息和2列表示亚细胞信息。
对于特征工程,用log10对FPKM中表示的基于光波长的表达矩阵进行归一化,以获得节点特征。亚细胞注释,包括信号肽和跨膜结构域注释,被工程化为节点特征。使用1和0来表示肽和跨膜蛋白编码基因是否存在。该设计基于复杂的环境微生物群相互作用的可用性。具有不同亚细胞位置的蛋白质的基因具有不同的相互作用模式。例如,分泌蛋白,即具有信号肽但没有跨膜结构域的蛋白,可能具有跨物种影响,而细胞内蛋白的基因主要用于基本的细胞代谢。最后的节点特征,表示为X,是基因表达和亚细胞定位特征的串联。
利用预处理的节点特征,我们构建了边缘来模拟细胞间的相互作用。我们采用tSNE降维并计算投影二维平面中节点之间的欧氏距离。在距离小于预设阈值的节点之间建立边,通过调整阈值使图的平均程度为5,即每个基因与预期的其他5个基因相连,连接到每个节点的边都是无向的。对边的边构建的生物理解是,无论物理空间距离如何,通过边缘连接的节点都具有相似的基因表达模式,即由于蛋白质的启动子调控机制或空间结构,序列中相距遥远的两个基因可以共表达或能够相互作用。
图神经网络和无监督聚类
基于构建的图,我们应用GNN来学习作为基因表示的节点嵌入,并实现进一步的分析。具体而言,我们采用了最具代表性的图卷积网络(GCN)。一层GCN的计算如下:



其中I是单位矩阵,是控制自循环强度的超参数,在我们的实验中设置为0.8。使用几层GCN,节点可以与其邻居交换信息,从而学习细胞间的相互作用。
考虑到节点标签信息的缺乏,我们采用了DGI算法、一种用于训练GNN的自监督算法,并基于改进的GNN来学习节点嵌入。具体而言,DGI的目标制定如下:


在我们的实验中,我们通过随机置换节点特征来扰动图,采用均值池作为读出函数,并将鉴别器设置为双线性函数。在训练并获得节点嵌入H后,我们使用主成分分析(PCA)降低节点嵌入的维数,并使用经典的基于向量的聚类算法对单元进行分组。我们根据湿式实验中的脱氮性能设置了簇的数量,表示为,选择典型的聚类数,包括n=24(eggNOG分类数)、10和7(KEGG 1级途径数),以比较它们对光转导基因的辨别能力。最后,我们使用n=7,这是用于后续分析的表现最好的聚类数。
模型评估
我们对基因面板识别能力进行了定性和定量评估,并对其生物学意义进行了验证。利用层次聚类和K-means这两种常用的聚类方法作为基准来评估DGI模型的聚类和信息集成能力。
为了进行定性评估,我们通过tSNE将基因聚类结果投影到2D空间上,并在表达矩阵上比较了层次聚类、K-means和DGI方法,然后进一步比较了有亚细胞信息和无亚细胞信息的K-means与DGI性能。我们利用轮廓系数指数(SCI)来定量评估不同方法的聚类能力。SCI用于评估聚类间的相似性。我们使用蓝光和黄光组的DEG表达矩阵来表示样本,并使用指定的基因簇数来表示标签。SCI是根据这些表示进行计算的。
鉴于复杂的环境微生物群中包含的丰富信息,我们基于文献综述和其他先前知识提取了对光反应的高信用途径,包括光转导、光感测途径;过氧化物酶体和寿命调节途径——多种环境应激相关途径;细胞色素P450和外源性物质通过含有蓝光受体的细胞色素P45 0途径的代谢。功能注释,包括Swiss Prot和KEGG数据库,也包括在内,用于富集分析和代谢网络重建。
为了验证其生物学功能,我们首先描绘了光转导的表达模式和簇分配。受光反应通路之间相似表达模式的启发,我们开发了FAS作为一种指标,以定量评估聚类结果与上下文生物学知识之间的一致性。FASw是分配给代谢通路w的基因的FAS,定义如下:

其中,n是目标功能的基因分配给的总簇数。所有簇都按比例降序排列。k是表示正确分配的簇的数量的超参数。考虑到属于同一基因组的基因对靶向功能有积极或消极的影响,我们假设具有两个最高基因比率的簇是正簇基因,即集合k=2,而其余的是负簇基因。变量ri是簇i与某一通路w的所有基因的基因比率,可以计算为:
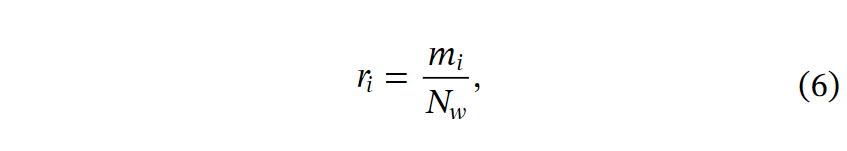


其中Mg是数据集的总基因计数。
光控反硝化基因的空间分布以及代谢通路富集分析
为了阐明光诱导反应的机制,我们提取了氮代谢和光转导基因所受的主要簇(图S8),即蓝光的簇7、簇4和黄光的簇6、簇3、簇4。我们提取了与硝酸盐和亚硝酸盐代谢相关的高表达基因(图S6B),以代表PD活性的关键基因。具体而言,硝酸盐和亚硝酸盐相关基因,即PD基因,是通过基于Swiss-Prot描述的关键词提取的。我们选择Swiss Prot数据库是因为它的注释比其他数据库更完整。考虑到低表达基因对微生物群集体行为的影响可以忽略不计,我们筛选出平均表达小于1的低表达基因。在Swiss-Prot中搜索并过滤低表达基因后,我们将蓝光和黄光的合格基因合并以获得PD基因集。
我们通过tSNE预测了这些功能基因在潜在空间中的表达空间分布,以描绘整体表达模式。为了确定功能基因组中的基本途径,我们将这些基因注释为KEGG 3级途径,并分别总结了对蓝光和黄光的反应表达模式。为了确定与微生物集体行为预测表型相对应的主要途径,有必要筛选低表达和不显著的途径。对于蓝光,p值<0.01、倍数变化<0.05或>2和表达水平<10 FPKM的途径被选为显著的光反硝化途径。同样对于黄光,最终获得了满足过滤要求的最高有效路径。用黑暗组作为对照计算折叠变化。
活性氧物种(ROS)的检测与分析
1,3-二苯基异苯并呋喃(DPBF)是一种活性氧荧光探针,通过量化DPBF的消耗量来评估总活性氧的产生。简言之,将DPBF溶解在75%乙醇中,并将2.5mM DPBF添加到96孔板中。使用超纯水来控制总分析体积。添加细胞样品后,通过微孔板读取器(Thermo Fisher Scientific,TENCAN Spark)以10 s的间隔连续监测UV-vis吸收光谱30分钟。基于UV-vis吸光光谱(图S14A),选择Abs410来监测总ROS产生。为了推导不同光照下总ROS产生的动力学常数,利用具有足够底物的时间点(Abs410>0.85)来计算DPBF消耗率,即总ROS生成率。数学方程如下:

其中kROSt是在时间t的总ROS产生速率,At和A0和是在时间t和初始时间在410nm的吸光度。OD600用于表示用于归一化的细胞密度。103被用于将计算结果调整到用于比较的合适范围。
胞外聚合物的提取与自催化实验
光脱氮后,按照先前研究中报道的方法提取EPS,并进行适当的修饰[55]。简言之,将细胞悬浮液充分振荡,并在4℃的温度下以4000g离心15分钟。收集上清液作为可溶性EPS(S-EPS)。此外,将残余细胞重悬于5%NaCl溶液中,并通过涡流混合器(Scientific Industries,vortex Genie2)搅拌3分钟,然后在60℃水浴下搅拌3分钟并再次搅拌3分钟。然后将混合物在4000g和4℃下离心15分钟,将获得的上清液收集为松散结合的EPS(LB-EPS)。为了获得紧密结合的EPS(TB-EPS),将剩余的污泥重悬在5%NaCl溶液中,如上所述快速搅拌3分钟,然后在60℃水浴30分钟,然后再次搅拌3分钟。之后,将混合物在10000g和4℃下离心15分钟,收集上清液作为TB-EPS。将等量的S-EPS、LB-EPS和TB-EPS混合在一起作为混合EPS(M-EPS)。利用从不同光照组中清除的M-EPS作为生物催化剂,在黑暗条件下促进模式反硝化菌Paraccocus反硝化菌(P.反硝化菌)的脱氮。在反硝化开始时加入等量的M-EPS,并在约24小时后定量硝酸盐去除效率。
基因拓扑网络的构建与代表性基因识别
我们构建了枢纽和信号基因面板的基因拓扑网络。对于蓝光,分别提取分配到簇4和簇5的基因作为中枢和信号基因面板。对于黄色光,相应地为簇3和簇1。还提取了分配给光转导的基因并将其整合到拓扑网络中。我们在Gephi 0.10.1中构建了网络。采用Pearson相关系数获得相关矩阵。为了获得高信用边缘,对相关系数<0.9和p值>0.05的边缘进行蓝光滤波。通过Gephi 0.10.1的内置算法,得到了度、加权度、模块类、偏心度、贴近度中心度、调和贴近度、介数中心度和聚类系数。结果可在数据集S1-S4中找到。利用暴露于光照的样品的平均表达水平来区分代表性基因。模块化类中排名前三的基因被定义为该类的代表性基因。
超氧自由基的检测与分析
超氧化物的细胞外产生通过MCLA(一种化学发光探针)进行评估。通过微孔板读数器(Thermo Fisher Scientific,TENCAN Spark)监测由MCLA与氧的超氧化物或单线态激发态的反应引起的化学发光30分钟。为每个样品设置一个以SOD作为超氧化物清除剂的额外对照组。加入MCLA和SOD后,立即加入注射器从光脱氮反应器中提取的细胞悬浮液,并在微板中进行监测。利用动态稳定点的化学发光差异测定超氧化物水平。为了减轻细胞密度的影响,我们将细胞密度调整到相似的水平,并用OD600标准化了化学发光差异。
生物法添加超氧自由基
我们采用温和的生物方法在反硝化过程中添加超氧化物。首先,设置三个试验组和对照组,在不添加超氧化物的黑暗条件下进行脱氮,以确保这两组具有相似的脱氮能力。在去除大部分硝酸盐后,同时补充超氧化物和硝酸盐,并持续监测硝酸盐浓度。反硝化系统中补充的超氧化物是由黄嘌呤氧化酶催化黄嘌呤氧化产生的。我们通过超氧化物检测实验(图S14C)和非生物硝酸盐超氧化物实验(图S1 4D)验证了这些方法。所有试剂,包括黄嘌呤、黄嘌呤氧化酶、WST-1均购自Dojindo Laboratories。在超氧化物检测实验中,超氧化物是按照指示通过黄嘌呤氧化产生的。没有将任何单元格添加到所有组中。仅在不添加SOD(10kU)的超氧化物基团处才能检测到约450nm处的峰值吸光度,这证明了该方法产生超氧化物和SOD作为超氧化物清除剂的可行性。大约一天后收集非生物实验的样品以测定硝酸盐浓度。很明显,硝酸盐不能被超氧化物去除(图S14D)。
代码和数据可用性
所有测序数据均保存在中国国家生物信息中心的数据库中,编号PRJCA017836 (https://ngdc.cncb.ac.cn/search/?dbId=&q=PRJCA017836)和国家生物技术信息中心的生物项目PRJNA984758 (https://www.ncbi.nlm.nih.gov/bioproject/PRJNA984758). 与研究相关的其他数据和代码在文章和/或支撑中。该软件包代码已在GitHub上发布(https://github.com/YoungeLiao/DMLA). 所有的补充材料(文本、图、表、中文翻译版本或视频)也可从线上获取。
引文格式:
Yang Liao, Jing Zhao, Jiyong Bian, Ziwei Zhang, Siqi Xu, Yijian Qin, Shiyu Miao, Rui Li, Ruiping Liu, Meng Zhang, Wenwu Zhu, Huijuan Liu, Jiuhui Qu. 2024. From mechanism to application: Decrypting light‐regulated denitrifying microbiome through geometric deep learning. iMeta e162. https://doi.org/10.1002/imt2.162
作者简介

廖洋(第一作者)
● 清华大学环境科学与工程博士。
● 研究方向为菌群光遗传与人工智能解析宏组学,相关学术成果已发表于iMeta、Water Research、ACS ES&T Engineering等国际学术期刊。

刘锐平(通讯作者)
● 清华大学教授,博士生导师。
● 研究方向为污染控制与资源能源转化,在Environ Sci Technol、Water Res、iMeta等期刊发表学术论文180余篇,主持国家重点研发计划项目、“863”重点项目、国家自然科学基金重大项目课题、优青、杰青及中科院重点部署项目课题等30余项。
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、50万用户的社交媒体宣传等。2022年2月正式创刊发行!目前期刊已经被ESCI、Scopus等数据库收录。
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science

































 2万+
2万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








