快速阅读目录
前面的几节中,我们介绍了GCTA计算G矩阵,和单性状遗传力的计算,它本质上就是GBLUP的估计,但是速度快很多。本节我们介绍,两性状遗传力和遗传相关的计算。
1. GCTA计算两性状遗传相关常用参数
1.1 --reml-bivar(必须)
这部分,是使用reml的方法进行估计方差组分。默认的是AI算法,可以使用EM算法。
1.2 --reml-alg 指定迭代方法(非必须)
–reml-alg 0 # AI算法,默认算法
–reml-alg 1 # EM算法
1.3 --grm(必须)
指定GRM矩阵
–grm # 接二进制文件GRM的前缀
–grm-bin # 同上
–grm-gz # 接文本的GRM文件前缀
推荐使用二进制的文件,因为速度快,暂用空间少。
1.4 --covar(非必须)
这是接因子协变量的,第一列和第二列分别是FID和IID,后面接因子协变量,比如场年季

1.5 --qcovar(非必须)
接的是数字协变量,比如PCA,比如初生重等

1.6 --pheno(必须)
接的是表型数据,格式也是plink的格式,第一列FID,第二列IID,第三列是表型数据(缺失用NA表示),第四列是表型数据

2. 数据准备
2.1 表型数据
三列,第一列是FID,第二列IID,第三列是表型数据y,没有行头,空格隔开。
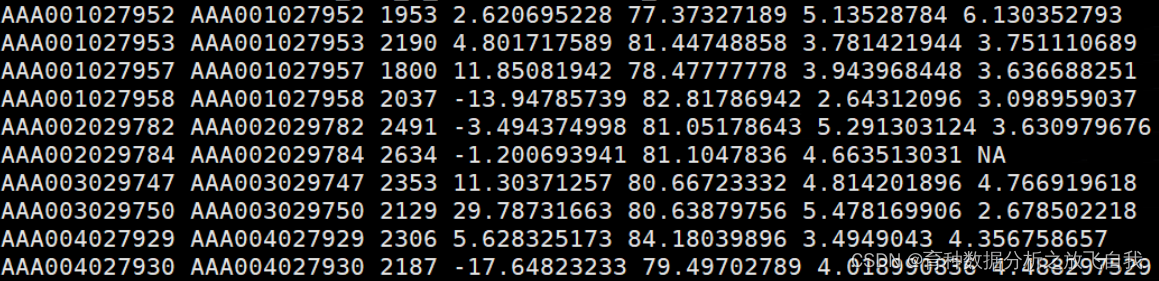
2.2 基因型数据
plink的二进制文件

2.3 协变量
这里,示例数据中,没有提供协变量信息。如果提供,可以按照第一列是FID,第二列是IID,其它是协变量的方法整理数据。协变量分为数字协变量和因子协变量,要分开整理。
3. 构建GRM矩阵
使用Van的方法
这里,用Van的方法,类似我们GBLUP估计所用的矩阵构建形式。
gcta64 --bfile ../test --make-grm --make-grm-alg 1 --out g2

结果文件:

4. 两性状估算遗传力和标准误
这里,已经构建好了GRM矩阵,指定表型数据,进行遗传力的估计
gcta64 --reml-bivar 1 2 --pheno ../nn_pheno.txt --grm ../grm/g1 --threads 30 --out re1

结果可以看出:
- Vg11:加性方差组分为:0.51
- Vg22:加性方差组分为:0.31
- Vg12:加性协方差为:-0.089
- Ve11:残差方差组分为:0.56
- Ve22:残差方差组分为:1.58
- Ve12:残差协方差组分为:-0.012
- Vp11:表型方差组分(Vg+Ve),为1.076
- Vptr2:表型方差组分(Vg+Ve),为1.89
第一个性状,遗传力为0.47,标准误是0.02
第二个性状,遗传力为0.16,标准误为0.03
遗传相关为:-0.22,标准误为0.094
使用ASReml作为对比
方差组分和遗传力结果:
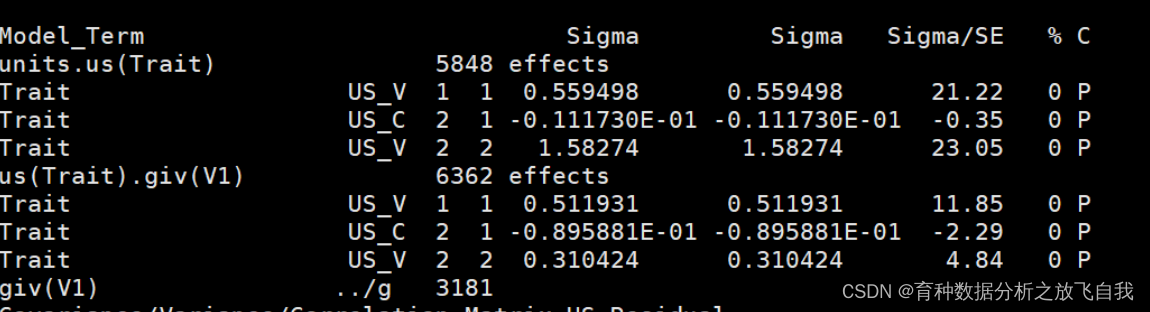
可以看出,结果完全一样。
5. GCTA多性状不支持的参数
多性状GCTA不支持的部分:
By default, GCTA will take the first two traits in the phenotype file for analysis. The phenotype file is specified by the option --pheno as described in univariate REML analysis. All the options for univariate REML analysis are still valid here except --mpheno, --gxe, --prevalence, --reml-lrt, --reml-no-lrt and --blup-snp. All the input files are in the same format as in univariate REML analysis.
- –mpheno
- –gxe
- –reml-lrt
- –reml-no-lrt
- –blup-snp
6. GCTA的优势和不足
GCTA只能分析两性状的GBLUP遗传评估,计算遗传力和遗传相关,速度很快。
相对于ASReml软件,缺点如下:
- 不支持固定因子缺失
- 只能是两个性状,3个,以及3个以上不支持
- 不支持多个随机因子
- 只能计算遗传相关,不能计算表型相关及标准误
欢迎关注我的公众号:
育种数据分析之放飞自我。主要分享R语言,Python,育种数据分析,生物统计,数量遗传学,混合线性模型,GWAS和GS相关的知识。
GCTA 系列教程
GCTA学习2 | 软件下载安装–windows和Linux
GCTA学习3 | GCTA的两篇NG:fast-LMM和fast-GLMM
























 4420
4420

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








