在做分子对接前,需要对蛋白质和分子进行预处理。对蛋白质进行:去水,去配体,加氢,处理;小分子需要加氢,做能量优化。
关于数据库:DUDE数据库上有蛋白质受体和配套的阳性、阴性配体。从DUDE数据库上下载的蛋白质和PDB数据库上下载的有所差异,但是对接使用的是蛋白质口袋,外圈的其实影响不大,DUDE上下载的受体打开看感觉基本已经处理好了,可以直接用于对接,以防万一可以用pymol打开看一眼。DUDE上下载的小分子文件也是准备好的,可以直接用。
1.pymol下载
1)pymol下载参考:
(18条消息) PyMol安装教程(Windows10+Python2.7)_blhbay的博客-CSDN博客_pymol安装
安装指南 — PyMOL中文教程 2022.09 文档 (chenzhaoqiang.com)
2)报错
这里忘记截屏了,安装遇到的错误是numba和numpy版本不匹配,可以以numba的版本为基础,查看需要的python版本,如果电脑的python版本过高(我就是过高了),可以conda创建一个python版本低的环境,再下载相应的whl进行安装。
2.蛋白质预处理
DUDE数据库蛋白质还是很有限的,只有102个,大部分时候还是从PDB上下载蛋白质结构,处理过后再拿去对接。
1)下载蛋白
pdb主页网址:RCSB PDB: Homepage
点进主页搜索蛋白质编号,我这里搜索1w0g为例: 1)主页搜索1w0g;2)下载PDB格式文件

3.pymol预处理蛋白质
1)去水:
pymol打开蛋白质pdb文件,点击Action-remove waters,进行去水处理

2)加氢:
点击Action-hydrogens-add

3)去配体:
点击右下角S,窗口上面会显示蛋白质序列,一般配体出现在序列最前端或是最后端
这里的HEM是蛋白质和小分子反应的催化剂,需要保留,,MYT是天然结晶的配体小分子。

点击MYT,右边列表会出现sele,指被选中分子(或原子),
这个时候可以点击file- Export Molecule,先把配体小分子单独保存下来,这里我把小分子文件重新命名为crystal_ligand,存为mol2格式 


保存好配体小分子后,仍然选中MYT,点击右侧sele的Action-remove atoms。删除掉配体后保存蛋白质文件,保存为pdb格式。(保存过程和保存小分子一样) 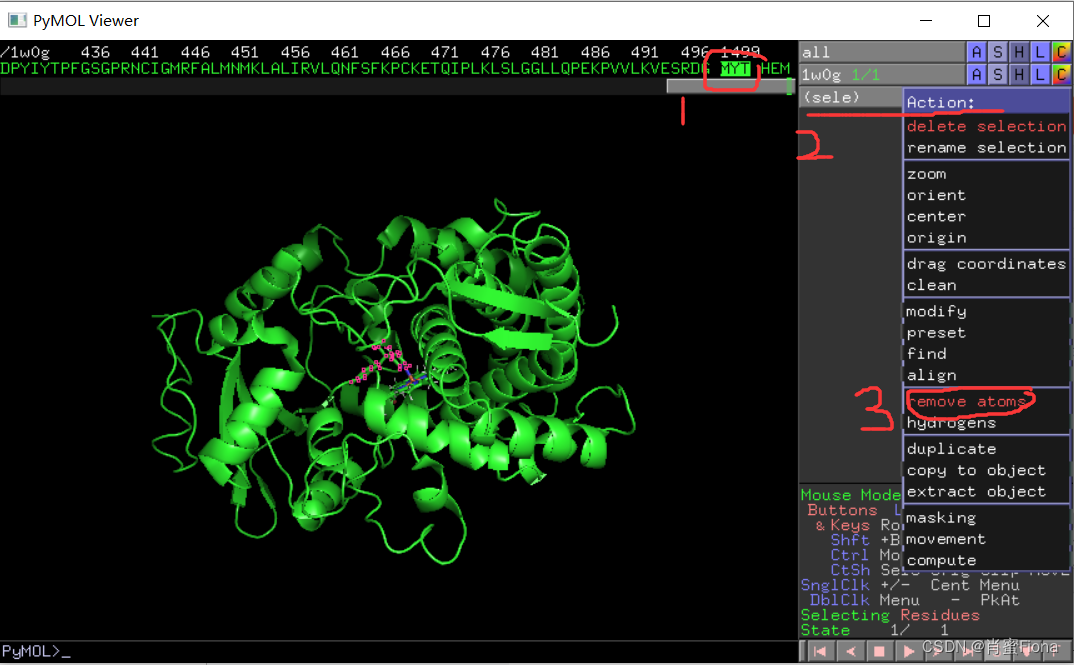
4)从多聚体蛋白中拆分出单体
对接一般是使用蛋白质单体,有时候从PDB下载的蛋白质是多聚体,这时候需要从多聚体蛋白质中拆分出单体,点击右下角Selecting 左边的英文单词(一般打开是Residues,点击会有变化),让它变成Chains,这时候点击窗口中央多聚体蛋白质的其中一个单体,可以看到单体中的所有原子都被选中了,点击右侧sele的Action-remove atoms,就可以删除多聚体中的其中一个单体了。

























 7671
7671

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








