一、介绍
在进行生物学实验或者生物信息的学习中,都会听说KEGG富集分析,而且该方法在高通量测序分析中已然成为数据分析中必不可少的一环。
这种分析方法依托的是由 Kanehisa实验室 在1995年开发的KEGG数据库,全称为 Kyoto Encyclopedia of Genes and Genomes(京都基因与基因组百科全书)。它拥有多个子数据库,包含基因组,生化反应,生化物质,疾病与药物,以及最常用PATHWAY通路信息。
接下来进入KEGG官网:https://www.kegg.jp ,它的主页主要由以下几部分构成:

二、KEGG的数据库构成
KEGG子库中存储的信息是生物系统的计算机表示形式,由基因和蛋白质(基因组信息)和化学物质(化学信息)的分子部件组成,这些部件的相互作用,反应和关系构成调控网络图(系统信息),除此之外,还包含疾病和药物信息(健康信息)。具体的分类及数据库如下:

三、KEGG PATHWAY 数据库
在所有子数据库中最重要也是最常用的就是KEGG PATHWAY,它包括大量由科研人员根据已有研究文献,通过手动绘制的KEGG通路图,代表着代谢过程,环境信息过程,细胞过程,生物系统,人类疾病和药物开发。
每个通路都由一个五位数字标识,后跟以下任意一个:map,ko,ec,rn和三字母或四字母生物代码,它们分别代表五种通路类型:
- map编号:代表reference pathway,根据已有的知识绘制的、概括的、详尽的具有一般参考意义的代谢图。 一个点同时表示一个基因,这个基因编码的酶或这个酶参加的反应
- org编号:物种特异性通路,这里就是将K编号基因(直系同源基因,后面会介绍)换为每个物种中对应的基因
- ko编号:KO通路中的点表示直系同源基因
- ec编号:EC通路中的点表示相关的酶
- rn编号:化学反应通路中的点只表示该点参与的某个反应、反应物及反应类型
在了解每种通路之前我们先学会在KEGG中切换每种通路类型
地址:https://www.kegg.jp/kegg-bin/show_pathway?org_name=map&mapno=00020&mapscale=&show_description=hide
使用过程中切换各种通路类型,比如进入TCA循环 ,可以通过左上角下路菜单来切换:

接下来,我们详细介绍每种通路:
1. 参考通路图 (map)
这里以 TCA循环 的通路图为例,进入参考通路图(Reference pathway)。这是原始版本的通路,也是后续几种通路图的"模板"。每个白框可以代表直系同源基因,酶,反应,也可以点击链接至KO,ENZYME和REACTION详细信息。

上述的形状,箭头,线段代表如下意义:

2. 物种特异性通路 (org)
我们选择人的物种名Homo sapiens (human),点击Go。可以看到与Reference pathway 图(map00020)不同的是有物种特异性基因被标注为绿色,而且通路编号为hsa00020
访问链接: https://www.kegg.jp/kegg-bin/show_pathway?org_name=hsa&mapno=00020&mapscale=&show_description=hide
当然,如果直接访问hsa00020的链接也可以进入该通路: https://www.kegg.jp/kegg-bin/show_pathway?hsa00020

点击绿色基因,会进入Gene详细信息

3. 直系同源物通路 (ko)
蓝色框超链接到从原始版本中选择的KO条目

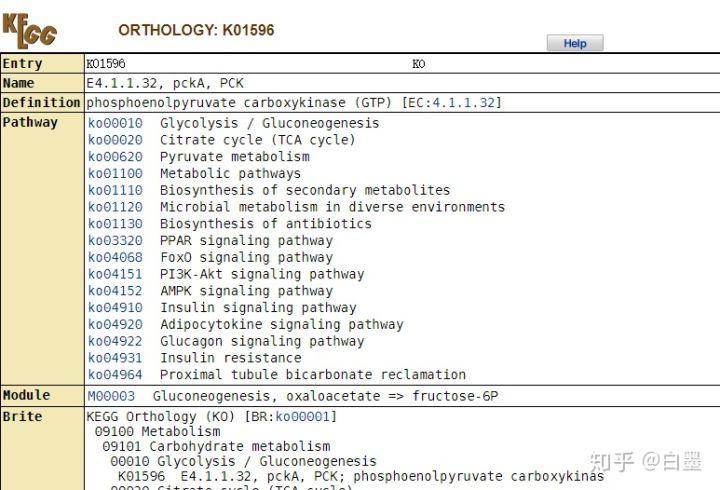
进入PCK的直系同源基因信息
4. 酶通路 (ec)
蓝色框超链接到从原始版本中选择的ENZYME条目

进入ENZYME

5. 反应通路 (reaction)
蓝色框超链接到从原始版本中选择的反应条目,

点击后进入对应的反应信息界面,如下图

四、KEGG ORTHOLOGY(KO)数据库
KEGG ORTHOLOGY (KO)数据库是构建Pathway和Module的基础,相当于KEGG数据库构建的基石,因此理解KO数据库的构成对于使用及了解KEGG至关重要。
然而,这种通用方法不足以理解由物种内基因和基因组的变异所引起的更详细的特征,特别是对于理解与人类基因和基因组的疾病相关的变异而言。后来他们开发了 KEGG NETKERK,该数据库不仅涉及基因变异,而且包括病毒和其他因素的网络变异方面的疾病和药物知识。
KEGG的开发者根据不同生物之间基因和基因组的保守和变异,引入直系同源物(KO)的概念,使得KEGG通路图,BRITE层次结构和KEGG模块的参考数据集可以广泛应用于任何细胞生物。

概念
1.KO号:表示不分物种的通路,相当于所有物种的这一通路的并集,比如ko00020代表的 TCA 循环 (下图所示),下图的每个圆角矩形也代表着一个KO通路。

2.K号:表示基因,每个号代表的是所有物种的一个同源基因,比如上图中的K01596代表的是 PCK。

进入K01596的详细页面,我们会看到它代表的是一个基因列表,这些基因具有一个功能却来自于不同的物种。

3.C号:表示化合物


























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










