
点击关注,桓峰基因
桓峰基因公众号推出单细胞系列教程,有需要生信分析的老师可以联系我们!单细胞系列分析教程整理如下:
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【14】单细胞调节网络推理和聚类 (SCENIC)
今天来说说单细胞转录组数据推断调节网络和聚类,学会这些分析结果,距离发文章就只差样本的选择了,有创新性的样本将成为文章的亮点,并不是分析内容了!
前 言
原理
GRN (gene regulatory network)基因调控网络包括TF (transcription factor转录因子)、cofactor(共调因子)与其调节的target gene组成,决定了某个状态下的细胞的转录状态。
SCENIC流程包括三步骤:
(1)使用GENIE3或GRNBoost(Gradient Boosting)基于共表达推断转录因子与候选靶基因之间的共表达模块。
(2)由于GENIE3模型只是基于共表达,会存在很多假阳性和间接靶标,为了识别直接结合靶标(direct-binding targets),使用RcisTarget对每个共表达模块进行顺式调控基序(cis-regulatory motif)分析。进行TF-motif富集分析,识别直接靶标。(仅保留具有正确的上游调节子且显著富集的motif modules,并对它们进行修剪以除去缺乏motif支持的间接靶标。)这些处理后的每个TF及其潜在的直接targets gene被称作一个调节子(regulon);
(3)使用AUCell算法对每个细胞的每个regulon活性进行打分。对于一个regulon来说,比较细胞间的AUCell得分可以鉴定出哪种细胞有显著更高的subnetwork活性。结果生成一个二进制的regulon活性矩阵(binarized activity matrix),这将确定Regulon在哪些细胞中处于"打开"状态。
这三部分都在桓峰基因公众号上分别介绍过:
RNA 26. SCI文章中基于转录组数据的基因调控网络推断 (GENIE3)
RNA 27 SCI文章中转录因子结合motif富集到调控网络 (RcisTarget)
SCS【13】单细胞转录组之识别细胞对"基因集"的响应 (AUCell)
今天将利用SCENIC 进行整合,对处理单细胞数据非常方便!!

<https://scenic.aertslab.org/
软件安装
软件包安装时要注意一下需要提前安装好 devtools包,方便调取 install_github
if (!requireNamespace("devtools", quietly = TRUE))
install.packages("devtools")
library(devtools)
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::version()
# If your bioconductor version is previous to 3.9, see the section bellow
## Required
BiocManager::install(c("AUCell", "RcisTarget"))
BiocManager::install(c("GENIE3")) # Optional. Can be replaced by GRNBoost
## Optional (but highly recommended):
# To score the network on cells (i.e. run AUCell):
BiocManager::install(c("zoo", "mixtools", "rbokeh"))
# For various visualizations and perform t-SNEs:
BiocManager::install(c("DT", "NMF", "pheatmap", "R2HTML", "Rtsne"))
# To support paralell execution (not available in Windows):
BiocManager::install(c("doMC", "doRNG"))
install.packages("doMC", repos="http://R-Forge.R-project.org")
# To export/visualize in http://scope.aertslab.org
devtools::install_github("aertslab/SCopeLoomR", build_vignettes = TRUE)软件包加载:
# Suppress loading messages when building the HTML
library(SCENIC)
library(AUCell)
library(RcisTarget)
library(SCopeLoomR)
library(KernSmooth)
library(BiocParallel)
library(ggplot2)
library(data.table)
library(grid)
library(ComplexHeatmap)数据准备
我们需要准备三个文件:
表达矩阵(Expression matrix),SCENIC的输入是一个单细胞RNA-seq表达式矩阵(以基因为"行名")
配套数据库:运行单细胞转录因子分析之SCENIC流程还需要下载配套数据库,不同物种不一样,在查看自己的物种,按需下载:
https://resources.aertslab.org/cistarget/
对于本教程,提供了一个玩具样例,只有200个细胞和<1000个基因来自小鼠大脑:
loomPath <- system.file(package = "SCENIC", "examples/mouseBrain_toy.loom")Open the loom file and load the expression matrix (and cell annotation if available)
library(SCopeLoomR)
loom <- open_loom(loomPath)
exprMat <- get_dgem(loom)
cellInfo <- get_cell_annotation(loom)
close_loom(loom)
dim(exprMat)
## [1] 862 200实例操作
这是用于运行SCENIC工作流的主要命令的概述。下面几节将解释这些命令:
1.初始化设置
### Initialize settings
library(SCENIC)
scenicOptions <- initializeScenic(org = "mgi", dbDir = "cisTarget_databases", nCores = 1)
# scenicOptions@inputDatasetInfo$cellInfo <- 'int/cellInfo.Rds'
saveRDS(scenicOptions, file = "int/scenicOptions.Rds")2.构建基因共表达网络
### Co-expression network
genesKept <- geneFiltering(exprMat, scenicOptions)
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.0 17.0 48.0 146.8 137.0 5868.0
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.00 10.00 25.00 39.01 60.00 180.00
exprMat_filtered <- exprMat[genesKept, ]
runCorrelation(exprMat_filtered, scenicOptions)
exprMat_filtered_log <- log2(exprMat_filtered + 1)
runGenie3(exprMat_filtered_log, scenicOptions)3.构建和评分GRN
### Build and score the GRN
library(doParallel)
library(foreach)
exprMat_log <- log2(exprMat + 1)
scenicOptions@settings$dbs <- scenicOptions@settings$dbs["10kb"] # Toy run settings
scenicOptions <- runSCENIC_1_coexNetwork2modules(scenicOptions)
## [,1]
## nTFs 8
## nTargets 770
## nGeneSets 47
## nLinks 17298
scenicOptions <- runSCENIC_2_createRegulons(scenicOptions, coexMethod = c("top5perTarget")) # Toy run settings
## [,1]
## top5perTarget 8
## [1] 22058
scenicOptions <- runSCENIC_3_scoreCells(scenicOptions, exprMat_log)
## min 1% 5% 10% 50% 100%
## 46.00 58.99 77.00 84.80 154.00 342.004.可选:二进制化活动
# Optional: Binarize activity
aucellApp <- plotTsne_AUCellApp(scenicOptions, exprMat_log)
# savedSelections <- shiny::runApp(aucellApp) newThresholds <-
# savedSelections$thresholds scenicOptions@fileNames$int['aucell_thresholds',1]
# <- 'int/newThresholds.Rds' saveRDS(newThresholds,
# file=getIntName(scenicOptions, 'aucell_thresholds'))
scenicOptions <- runSCENIC_4_aucell_binarize(scenicOptions)
tsneAUC(scenicOptions, aucType = "AUC") # choose settings
## [1] "int/tSNE_AUC_50pcs_30perpl.Rds"
# Export: saveRDS(cellInfo, file=getDatasetInfo(scenicOptions, 'cellInfo')) #
# Temporary, to add to loom export2loom(scenicOptions, exprMat) To save the
# current status, or any changes in settings, save the object again:
# saveRDS(scenicOptions, file='int/scenicOptions.Rds')5.探索输出
### Exploring output Check files in folder 'output' Browse the output .loom
### file @ http://scope.aertslab.org output/Step2_MotifEnrichment_preview.html
### in detail/subset:
motifEnrichment_selfMotifs_wGenes <- loadInt(scenicOptions, "motifEnrichment_selfMotifs_wGenes")
tableSubset <- motifEnrichment_selfMotifs_wGenes[highlightedTFs == "Sox8"]
viewMotifs(tableSubset)# output/Step2_regulonTargetsInfo.tsv in detail:
regulonTargetsInfo <- loadInt(scenicOptions, "regulonTargetsInfo")
tableSubset <- regulonTargetsInfo[TF == "Stat6" & highConfAnnot == TRUE]
viewMotifs(tableSubset)# Cell-type specific regulators (RSS):
regulonAUC <- loadInt(scenicOptions, "aucell_regulonAUC")
rss <- calcRSS(AUC = getAUC(regulonAUC), cellAnnotation = cellInfo[colnames(regulonAUC),
"CellType"], )
rssPlot <- plotRSS(rss)
plotly::ggplotly(rssPlot$plot)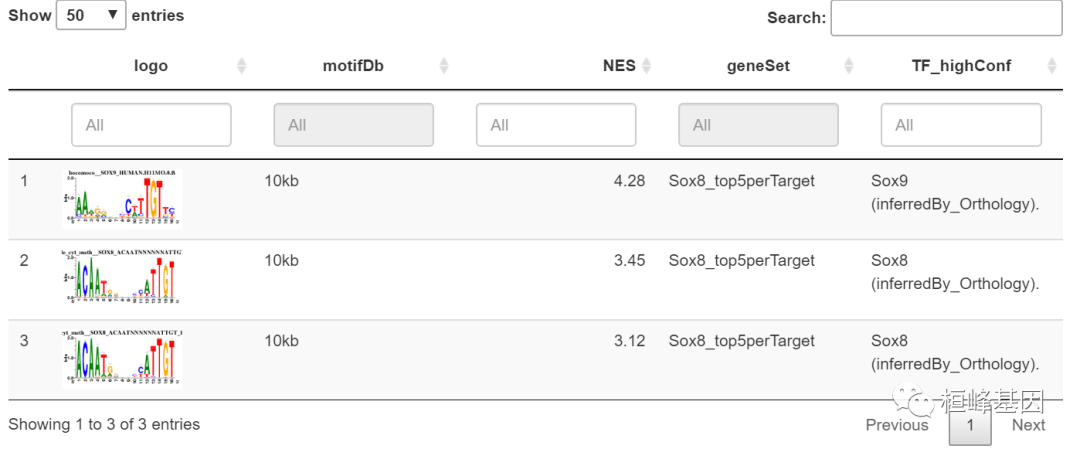


结果可视化
SCENIC通过几个step即可绘制一些默认的图片,可以看下output输出的文件夹:3-4步骤会出现很多结果文件,分别说一下:

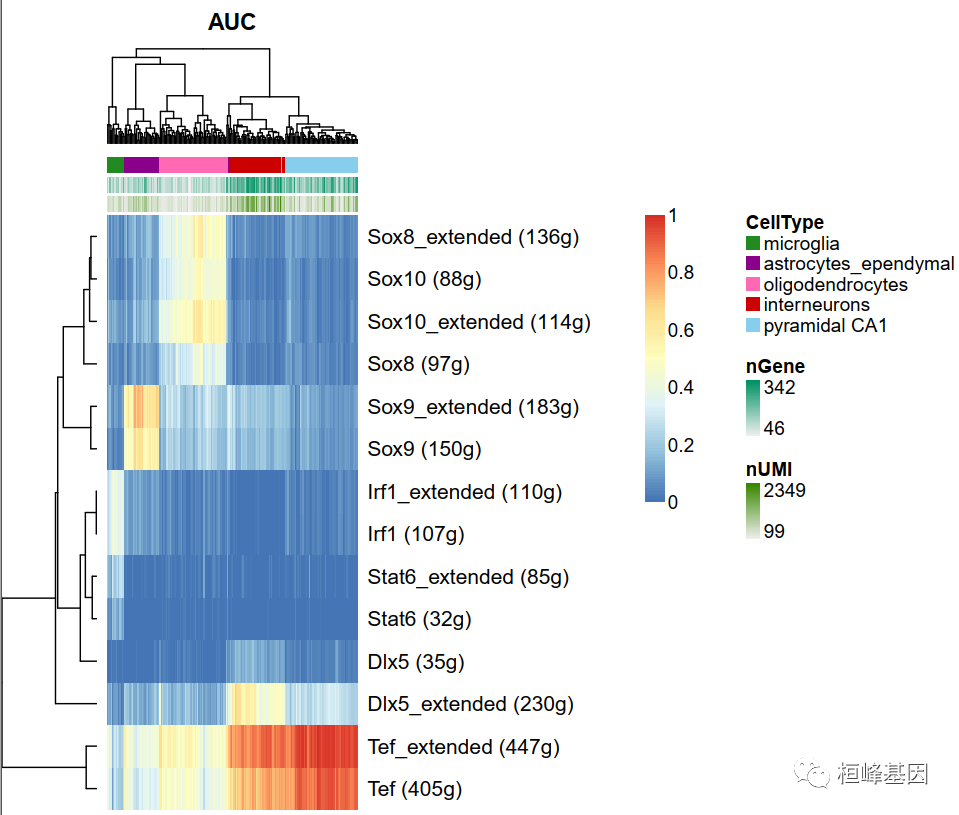
1. Regulon Activity heatmap
step3有所有regulon在细胞的AUCscore热图Step3_RegulonActivity_heatmap.pdf
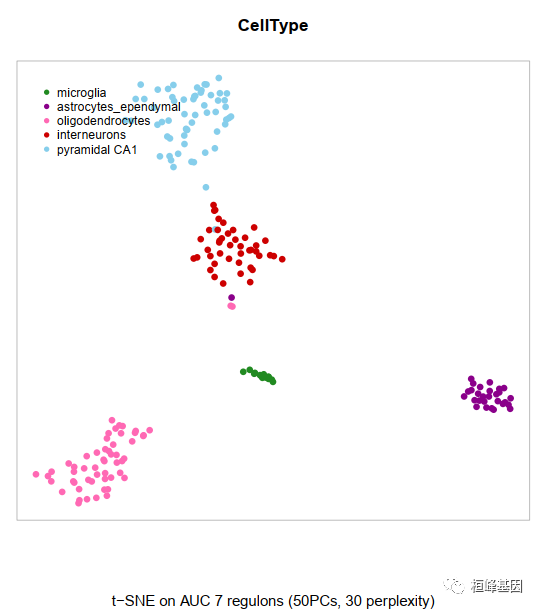
step3有所有regulon在细胞聚类的Step3_RegulonActivity_tSNE_colByCellProps.pdf
step3有所有的基因的筛选结果见文件夹Step3_RegulonActivity_tSNE_colByActivity

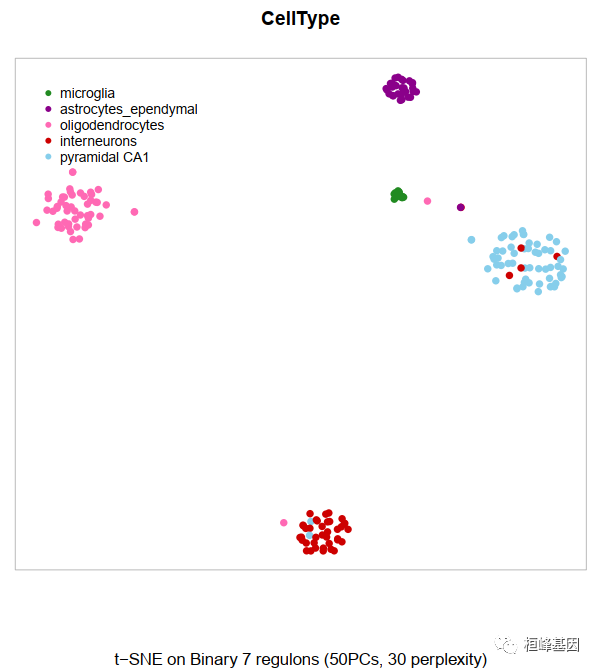
2.Binary Regulon Activity Heatmap
step4则生成多个热图Step4_BinaryRegulonActivity_Heatmap*
a. Step4_BinaryRegulonActivity_Heatmap_all.pdf

b. Step4_BinaryRegulonActivity_Heatmap_corr.pdf

c. Step4_BinaryRegulonActivity_Heatmap_onePercent.pdf

d. Step4_BinaryRegulonActivity_tSNE_colByCellProps.pdf


e. Step4_BoxplotActiveCellsRegulon.pdf

我们这期主要介绍单细胞转录组数据推断调节网络和聚类(SCENIC)。目前单细胞测序的费用也在降低,单细胞系列可算是目前的测序神器,有这方面需求的老师,联系桓峰基因,提供最高端的科研服务!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!

References:
Aibar, Sara, et al. 2017. "SCENIC: Single-Cell Regulatory Network Inference and Clustering." Nature Methods 14 (october): 1083--6.doi:10.1038/nmeth.4463.
Davie, et al. 2018. "A Single-Cell Transcriptome Atlas of the Aging Drosophila Brain." Cell, june. doi:10.1016/j.cell.2018.05.057.
Huynh-Thu, Vân Anh, Alexandre Irrthum, Louis Wehenkel, and Pierre Geurts. 2010. "Inferring Regulatory Networks from Expression Data Using Tree-Based Methods." PloS One 5 (9).doi:10.1371/journal.pone.0012776.
Marbach, et al. 2012. "Wisdom of Crowds for Robust Gene Network Inference." Nature Methods 9 (8): 796--804.doi:10.1038/nmeth.2016.






















 376
376











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








