
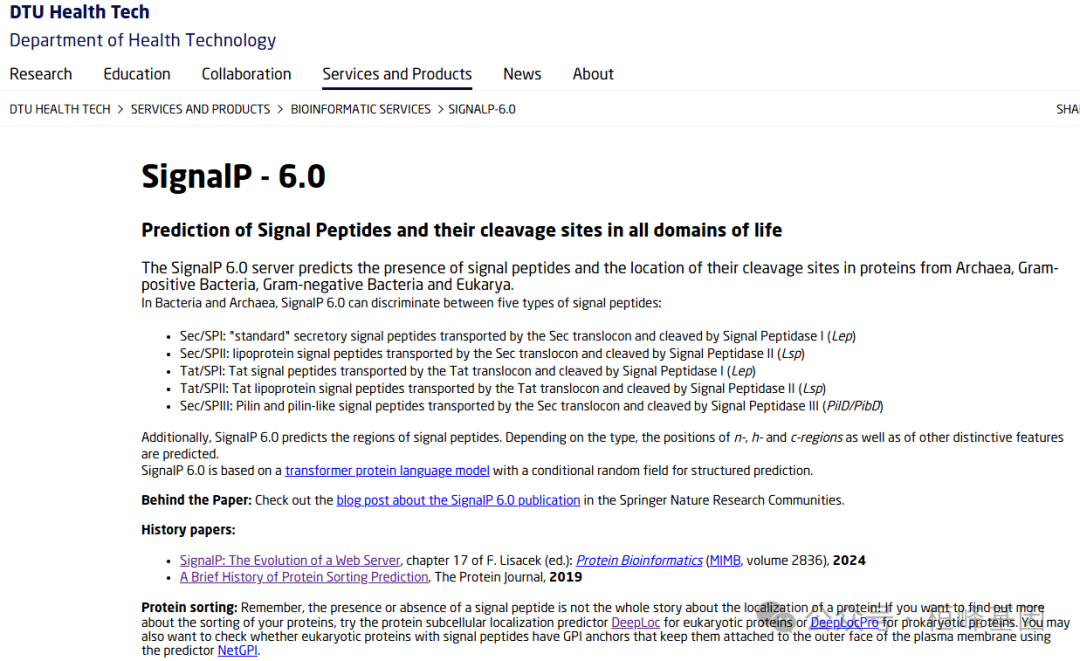
简 介
信号肽(SPs)是一种短的氨基酸序列,在所有生物体中控制蛋白质分泌和易位。SPs可以从序列数据中预测,但现有的算法无法检测到所有已知类型的SPs。我们介绍SignalP6.0,这是一个机器学习模型,可以检测所有五种SP类型,并适用于宏基因组数据。

SignalP 在线分析工作流程:
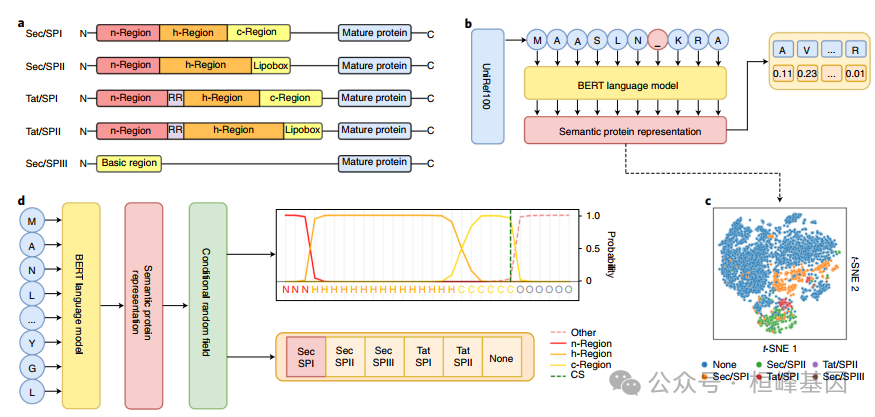
a、五种SP类型的区域结构。
b、蛋白质LM训练程序。
c、预测训练前蛋白质表示的t-分布随机邻居嵌入(t-SNE)投影。
d、signalp6.0架构。
文件准备
这个输入文件只有一个文件可以是蛋白序列文件,例如:
>IPI00000001.2 SWISS-PROT:O95793-1 TREMBL:A8K622;Q59F99 ENSEMBL:ENSP00000360922;ENSP00000379466 REFSEQ:NP_059347 H-INV:HIT000329496 VEGA:OTTHUMP00000031233 Tax_Id=9606 Gene_Symbol=STAU1 Isoform Long of Double-stranded RNA-binding protein Staufen homolog 1
MSQVQVQVQNPSAALSGSQILNKNQSLLSQPLMSIPSTTSSLPSENAGRPIQNSALPSAS
ITSTSAAAESITPTVELNALCMKLGKKPMYKPVDPYSRMQSTYNYNMRGGAYPPRYFYPF
PVPPLLYQVELSVGGQQFNGKGKTRQAAKHDAAAKALRILQNEPLPERLEVNGRESEEEN
LNKSEISQVFEIALKRNLPVNFEVARESGPPHMKNFVTKVSVGEFVGEGEGKSKKISKKN
AAIAVLEELKKLPPLPAVERVKPRIKKKTKPIVKPQTSPEYGQGINPISRLAQIQQAKKE
KEPEYTLLTERGLPRRREFVMQVKVGNHTAEGTGTNKKVAKRNAAENMLEILGFKVPQAQ
PTKPALKSEEKTPIKKPGDGRKVTFFEPGSGDENGTSNKEDEFRMPYLSHQQLPAGILPM
VPEVAQAVGVSQGHHTKDFTRAAPNPAKATVTAMIARELLYGGTSPTAETILKNNISSGH
VPHGPLTRPSEQLDYLSRVQGFQVEYKDFPKNNKNEFVSLINCSSQPPLISHGIGKDVES
CHDMAALNILKLLSELDQQSTEMPRTGNGPMSVCGRC在线分析
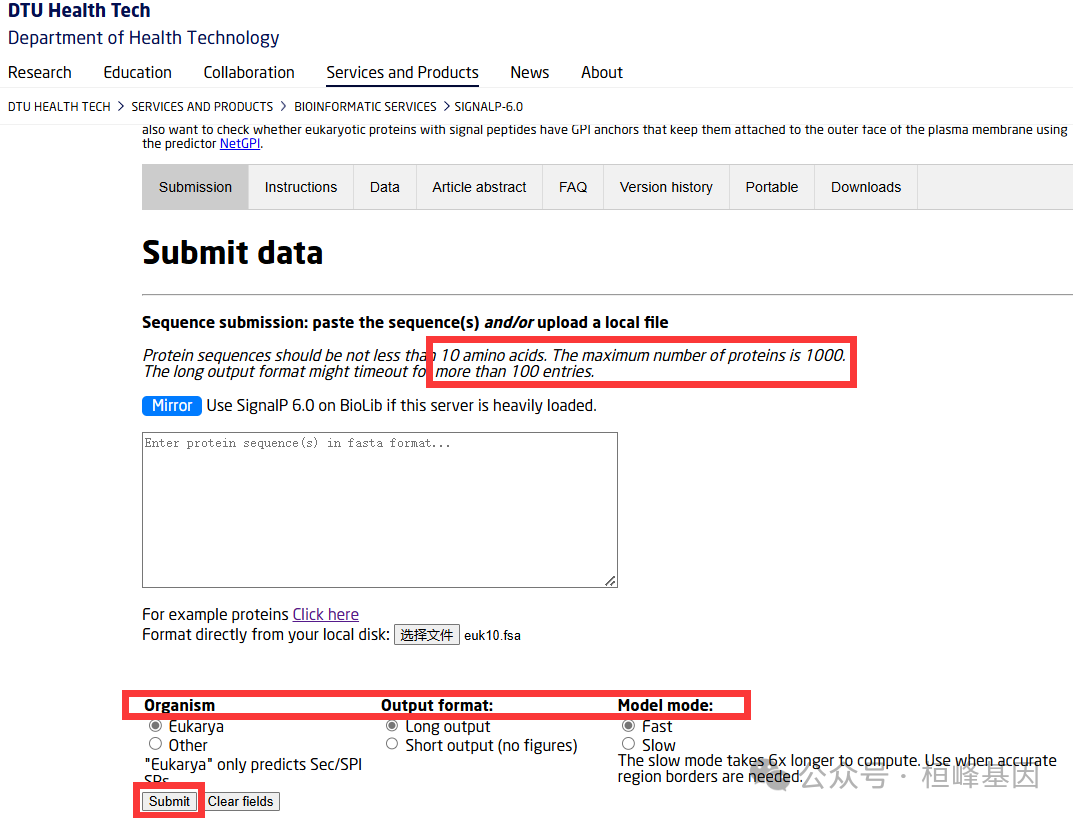
在线网址SignalP,或者SignalP在线使用还是非常简单,序列少可以优先选择在线操作。
线上分析对数据量要求有一定局限性:
蛋白质序列应不少于10个氨基酸。蛋白质的最大数量为1000个。超过100个条目时,长输出格式可能会超时。
SignalP 测试结果:
生成5个文件,可以直接点击文件下载,也可以一起下载压缩包。如下:

最主要的是看一下预测结果文件:
# SignalP-6.0 Organism: Eukarya Timestamp: 20240806060926
# ID Prediction OTHER SP(Sec/SPI) CS Position
IPI00000001.2 OTHER 1.000000 0.000000
IPI00000005.1 OTHER 1.000000 0.000000
IPI00000006.1 OTHER 1.000000 0.000000
IPI00000012.4 OTHER 1.000000 0.000000
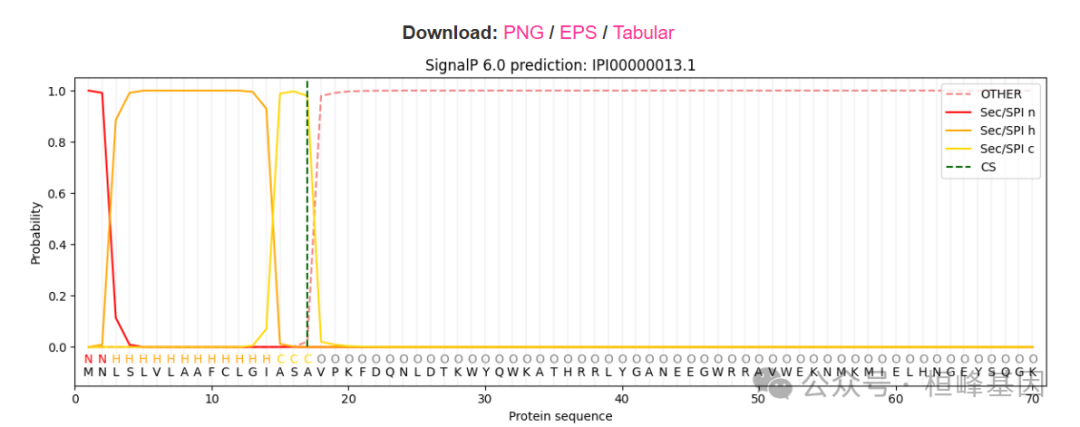
IPI00000013.1 SP 0.000193 0.999768 CS pos: 17-18. Pr: 0.9795
IPI00000015.2 OTHER 1.000000 0.000000
IPI00000017.1 OTHER 0.999749 0.000252
IPI00000020.1 OTHER 1.000000 0.000000
IPI00000021.5 OTHER 0.983094 0.016937
IPI00000023.4 SP 0.311199 0.688786 CS pos: 40-41. Pr: 0.3726Prediction: 预测类型。OTHER 表示没有信号肽, SP (Sec/SPI), LIPO (Sec/SPII), TAT (Tat/SPI), TATLIPO (Tat/SPII), PILIN (Sec/SPIII)后面的这几种都是有信号肽,括号里是用到的切割酶。
CS Position:切割位置。信号肽酶切割的序列位置及其预测概率。
processed_entries.fasta:预测的成熟蛋白质,即去除其信号肽的序列。
output.gff3:GFF3格式的所有预测信号肽的起始和终止位置。
region_output.gff3:GFF3格式的所有预测信号肽区域的起始和终止位置。
output.json:预测结果为JSON格式,以及运行参数和生成的输出文件路径的详细信息。
预测蛋白(Predicted proteins)
本地分析
软件包安装
首先下载软件包,这个 SignalP-6.0 软件包基于python3版本,下载的时候注意一下需要输入带有.edu类后缀的邮箱,否则也下载不了,因为我没有下成功,但是求别人所有了 signalp-5.0b 版本的,目前条件邮箱,安装操作都差不多哈。
tar zxf ~/software/SignalP/signalp-5.0b.tar.gz
echo 'PATH=$PATH:~/signalp-5.0/bin' >> ~/.bashrc
source ~/.bashrc测试安装是否成功
signalp
-batch int
Number of sequences that the tool will run simultaneously. Decrease or increase size depending on your system memory. (default 10000)
-fasta string
Input file in fasta format.
-format string
Output format. 'long' for generating the predictions with plots, 'short' for the predictions without plots. (default "short")
-gff3
Make gff3 file of processed sequences.
-mature
Make fasta file with mature sequence.
-org string
Organism. Archaea: 'arch', Gram-positive: 'gram+', Gram-negative: 'gram-' or Eukarya: 'euk' (default "euk")
-plot string
Plots output format. When long output selected, choose between 'png', 'eps' or 'none' to get just a tabular file. (default "png")
-prefix string
Output files prefix. (default "Input file prefix")
-stdout
Write the prediction summary to the STDOUT.
-tmp string
Specify temporary file directory. (default "System default tmpdir")
-verbose
Verbose output. Specify '-verbose=false' to avoid printing. (default true)
-version
Prints version.
No input file provided.实际操作
1. 参数说明
signalp命令的参数:
-batchdefault: 10000 程序并行化运行的序列条数。程序能多线程运行,速度很快。推荐设置该参数值大于FASTA文件的序列总条数,虽然增加内存消耗,但能加快程序运行。
-orgdefault: euk 设置分析的物种类型。该参数值有4种:arch,古菌;gram+,革兰氏阳性细菌,gram-,革兰氏阴性细菌;euk,真核生物。
-fasta输入FASTA格式的蛋白序列文件。
-prefix设置输出文件前缀。默认在程序运行目录下输出结果文件,和输入文件名的前缀相同,后缀为_summary.signalp5。
-gff3 添加该参数,输出GFF3格式的信号肽预测结果。
-mature 添加该参数,对含有信号肽的蛋白序列,切除信号肽后输出其成熟蛋白序列。可以用于下游的跨膜区分析。
-tmpdefault: /tmp 设置临时文件夹路径。
-formatdefault: short 设置输出格式。该参数值有两个:short,仅输出一个信号肽预测的整合文本结果;long,额外输出每条序列的各位点预测文本结果和图片结果;
-plotdefault: png 设置输出图片结果的类型。当--format参数为long时,该参数生效。该参数值有三个:png;eps;none表示仅得到表格文件。
-version 打印程序版本并退出。
2. 实际操作命令如下:
signalp -batch 30000 -org euk -fasta test/euk10.fsa -gff3 -mature结果解读
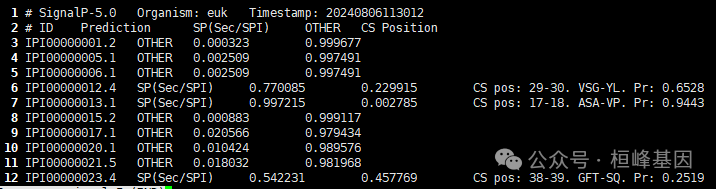
Prediction: 预测类型。OTHER 表示没有信号肽, SP (Sec/SPI), LIPO (Sec/SPII), TAT (Tat/SPI), TATLIPO (Tat/SPII), PILIN (Sec/SPIII)后面的这几种都是有信号肽,括号里是用到的切割酶。
CS Position:切割位置。信号肽酶切割的序列位置及其预测概率。
其他两个文件内容:
euk10_mature.fasta:预测的成熟蛋白质,即去除其信号肽的序列。
euk10.gff3:GFF3格式的所有预测信号肽的起始和终止位置。
Reference
Teufel, F., Almagro Armenteros, J.J., Johansen, A.R. et al. SignalP 6.0 predicts all five types of signal peptides using protein language models. Nat Biotechnol 40, 1023–1025 (2022).
Almagro Armenteros, J.J., Tsirigos, K.D., Sønderby, C.K. et al. SignalP 5.0 improves signal peptide predictions using deep neural networks. Nat Biotechnol 37, 420–423 (2019).
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/






















 1382
1382

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








