欢迎关注”生信修炼手册”!
在GWAS分析结果中,大部分显著的SNP位点都位于非编码区,很难直接挖掘这些位点的调控机制。通常假设与疾病关联的SNP位点通过调控基因表达来发挥作用,而eQTL可以识别SNP与基因间的调控关系,将eQTL和GWAS结果相结合,可以进一步筛选候选基因。
科学家们发明了许多的工具来完成这一任务,根据时间先后顺序,部分软件列表如下
RTC
Sherlock
coloc
eCaviar
enloc
不同软件运用的模型和算法不尽相同,本文先来看一下Sherlock这个软件。
仅根据GWAS的结果来筛选基因时,只能筛选出显著关联的SNP位点所在的基因,这种做法类似cis-eQTL,收到了距离的限制,无法全面挖掘后续基因。Sherlock软件同时利用了cis和trans-eQTL的信息来识别候选基因,利用trans-eQTL突破距离限制,可以识别到更多的候选基因,对应的文章如下
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3644637/
筛选候选基因的基本模型如下
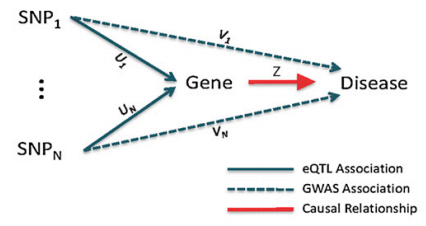
SNP位点在GWAS和eQTL分析中同时显著,这样的SNP位点对应的靶标基因就是潜在的候选基因。以基因为单位,比对gwas和eQTL结果,示意如下
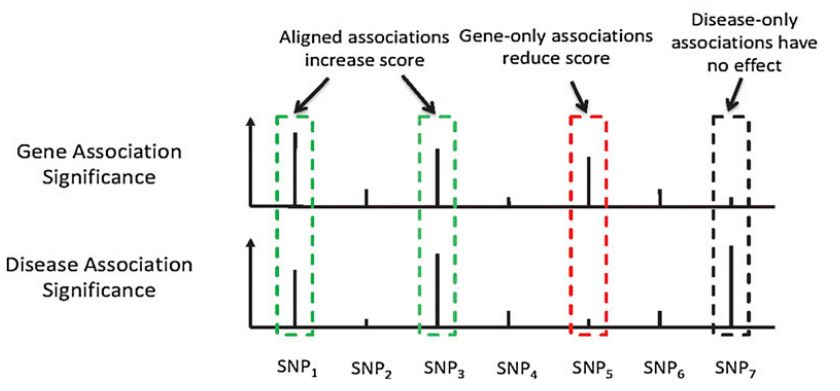
分为了3种情况,绿色部分表示在两个结果中同时显著,红色表示只在eQTL中显著,黑色表示只在gwas中显著。根据SNP位点在两种分析中的一致性,计算一个称之为LBF的值来表征其一致性,绿色位点加分,红色位点减分。
该软件官网如下
http://sherlock.ucsf.edu/index.html
提供了在线服务,eQTL结果利用的是公共数据,只需上传自己的gwas结果即可,内容示意如下








 本文介绍了如何利用GWAS和eQTL分析相结合的方法筛选疾病相关基因,特别是通过Sherlock软件来突破距离限制,识别更多的候选基因。通过对GWAS和eQTL结果的比较,计算一致性得分来确定潜在的疾病基因。Sherlock提供在线服务,用户只需上传GWAS结果,即可进行分析。
本文介绍了如何利用GWAS和eQTL分析相结合的方法筛选疾病相关基因,特别是通过Sherlock软件来突破距离限制,识别更多的候选基因。通过对GWAS和eQTL结果的比较,计算一致性得分来确定潜在的疾病基因。Sherlock提供在线服务,用户只需上传GWAS结果,即可进行分析。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1134
1134

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








