导读
今天有小伙伴问我,筛选完差异基因后,想看自己关注的基因和其他基因的关系,应该怎么做?那当然要先做一下相关性分析了!好,下面让我给大家娓娓道来…
正文:
step1:我们先读取下所需要的数据
library(openxlsx)setwd("E:\\Bioinfo_analysis\\scripts\\corr\\corr_batch") #设置工作路径
fr<-read.xlsx('infile.xlsx',rowNames = T,colNames = T)
View(fr) #查看下数据类型,列名基因名,行名为样本名

Step2:下载安装包
library("ggstatsplot") #加载包的时候失败了,因为之前没有安装这个包
library("BiocManager")
BiocManager::install("ggstatsplot") #安装ggstatsplot
要安装很多依赖包,泡杯咖啡,静静的等着它,差不多花了30分钟才把所有的包安装完,下面开始进行相关性分析,假如我们关注的基因是COX1和ATP6。
Step3:绘图
ggscatterstats(data = fr,
y = COX1,
x = ATP6,
centrality.para = "mean",
margins = "both",
xfill = "red",
yfill = "blue",
marginal.type = "histogram",
title = "Relationship")

图出来的还是很快的,大概5秒钟。图中每个点代表一个样本,两条虚线分别代表两个基因在样本中的平均表达量。看一下结果,P<0.001,r=0.46,说明COX1, ATP6存在正相关关系,P值过关了,不过相关系数r有点低,还没有超过0.5。
到这里结束了么,不。我的小伙伴又问了,我想看一下COX1和其他所有基因的相关性怎么办?你手动一个个画呗。。。。哈哈哈,开个玩笑,这里我们可以用ggcorrplot绘图!
corr.result<-cor(fr,method = 'pearson') #先计算相关性系数
corr.p<-ggcorrplot::cor_pmat(fr) #再计算P值
#开始绘图
ggcorrplot( corr = corr.result, type = 'full')

如果你想看哪些基因间是显著性相关,怎么办,往下看:
ggcorrplot(
corr = corr.result,
type = 'full',
p.mat = corr.p,#P-Value
sig.level = 0.05 #P-Value大于0.05的在图中标记出来)
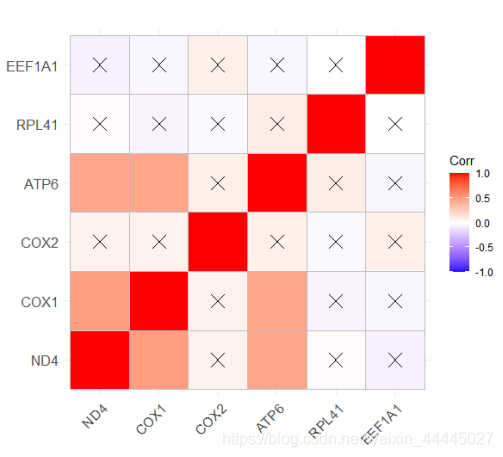
图中标记❌号的即为P值大于0.05的,反之为P值小于0.05。今天就给大家介绍到这里,有什么疑问或者建议,可以下方留言探讨哦!







 本文详细介绍如何使用R语言进行TCGA和GEO数据集的基因相关性分析,包括数据读取、包的安装、相关性绘图及批量分析流程,通过实例演示了如何探究特定基因与其他基因间的相关性。
本文详细介绍如何使用R语言进行TCGA和GEO数据集的基因相关性分析,包括数据读取、包的安装、相关性绘图及批量分析流程,通过实例演示了如何探究特定基因与其他基因间的相关性。
















 135
135

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








