lncRNA能够将miRNA吸附过来,相当于竞争性结合,不让miRNA对mRNA发挥作用,因此形成了lncRNA-miRNA-mRNA的调控网络,简称ceRNA。
在做生信分析时,我们通过数据库筛选了一些调控自己靶基因mRNA的lncRNA,可以使用很多网站预测lncRNA结合的miRNA。
那么问题来了,大部分网站通常都是输入一个lncRNA,预测与其结合miRNA。如果我们筛选到了几十个甚至几百个lncRNA,如何批量预测lncRNA靶向的miRNA。
我向大家推荐一个网站:mircode。
直接点击download:

一般我们下载高度保守的miRNA数据,数据如下:
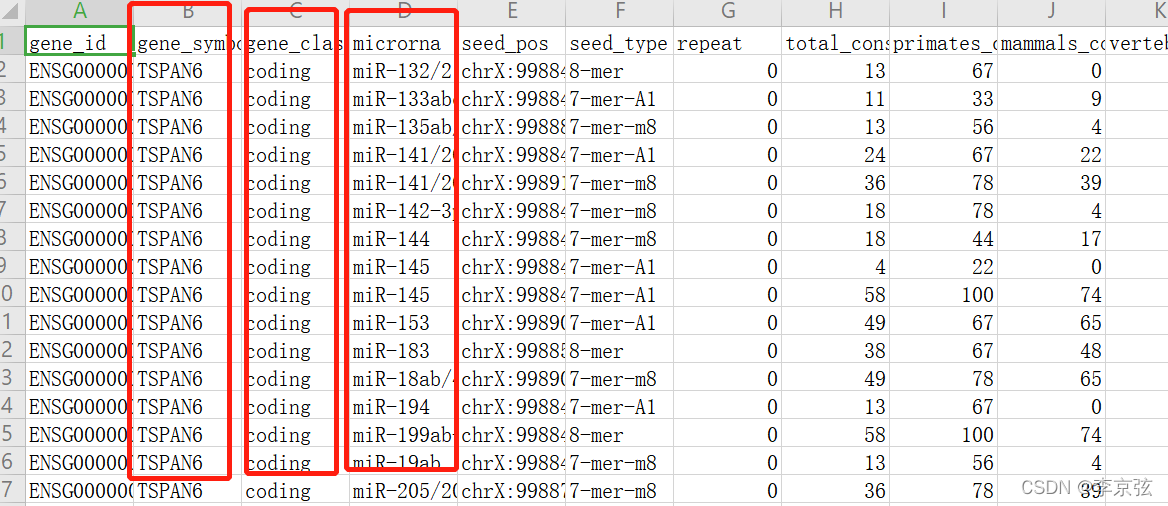
红框的三列是主要的,首先准备好自己的lncRNA文件:

读取文件,然后运行以下代码:
setwd("F:\\lncRNA")## 设置工作路径
dir()
data <- read.csv("lncRNA.csv",header = T,sep = ",")## 读取自己筛选的差异lncRNA
head(data)
miRNA <- read.table("mircode.txt",header = T,sep = "\t")## 读取mircode下载的文件
head(miRNA)
miRNA <- miRNA[miRNA$gene_symbol %in% data$LncRNA,] ## 从miRNA中提取我们筛选的差异lncRNA
dim(miRNA)
head(miRNA)
write.csv(miRNA,"lncRNA结合的miRNA.csv")










 本文介绍了如何利用生信分析工具批量预测大量lncRNA的miRNA结合情况,重点讲解了使用mircode网站下载高度保守miRNA数据,并提供了一套步骤来筛选特定lncRNA并生成结合miRNA的CSV文件。
本文介绍了如何利用生信分析工具批量预测大量lncRNA的miRNA结合情况,重点讲解了使用mircode网站下载高度保守miRNA数据,并提供了一套步骤来筛选特定lncRNA并生成结合miRNA的CSV文件。


















 1894
1894

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










