
写在前面
在与生物相关的研究中,生信分析基本上已经成为了一个绕不开的过程,面对高通量测序的大量数据,我们可能需要在Linux系统中使用专门的生信分析工具完成,这些工具通常学习成本较高,对于大部分研究人员来说很难快速的独立完成。
但是在研究过程中,我们除了需要对大数据进行分析之外,面对少量的数据进行一些统计、识别、比对、功能分析、可视化也是需求量很大的工作内容,这是一些功能单一、操作简单的在线分析工具就成为了更优的选择。
本篇文章将介绍100个在线的生信分析小工具,涵盖了几乎所有可能遇到的分析内容。
这些在线分析工具通常操作比较简单,所以本文只会给出工具的网址及其能够实现的功能,部分工具可能会简单介绍一下工具使用的限制,但是并不会详细介绍这些工具的使用方法。
PS:如果特别想要知道某一个自己搞不懂的工具的用法,可以后台私信我或者加微信私聊,对于询问的多的工具,后面也会写专门的文章进行介绍。
1. PathogenFinder
网址:https://cge.cbs.dtu.dk//services/PathogenFinder/
根据细菌的全基因组预测其对人类的致病性。

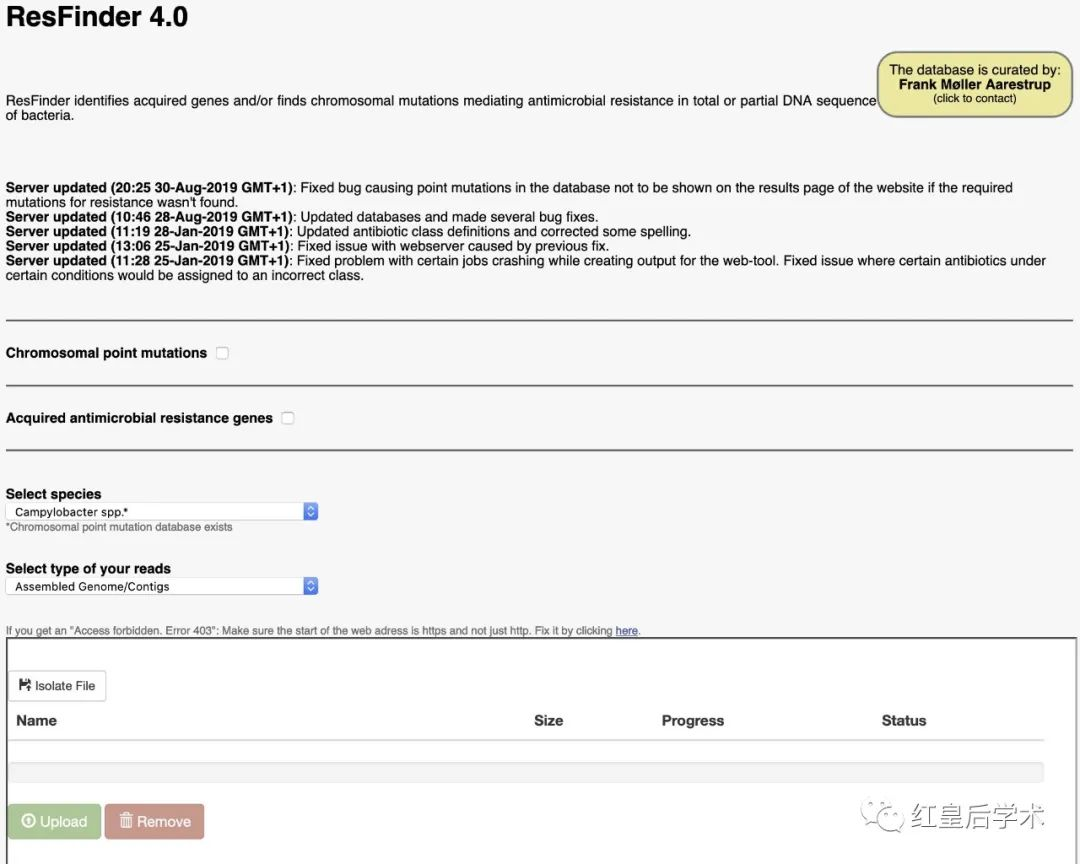
2. ResFinder
网址:https://cge.cbs.dtu.dk//services/ResFinder/
识别细菌全基因组序列中的抗生素抗性基因。
3. DNA/RNA GC Content Calculator
网址:http://www.endmemo.com/bio/gc.php
计算核酸序列的GC含量和序列长度。

4. RAST
网址:https://rast.nmpdr.org/
基于SEED的细菌和古菌全基因组注释工具。

5. USCS Genome Browser
网址:http://genome.ucsc.edu/cgi-bin/hgGateway
基因组数据浏览器。

6. CGView
网址:http://stothard.afns.ualberta.ca/cgview_server/
以环状形式对基因组进行可视化。

7. ATGC:Alignable Tight Genomic Clusters
网址:http://atgc.lbl.gov/atgc/
微生物基因组聚类及系统发育关系数据库。

8. IslandViewer
网址:http://www.pathogenomics.sfu.ca/islandviewer/
整合了IslandPick、IslandPath-DIMOB和SIGI-HMM预测方法的基因岛预测工具。

9. EasyGene
网址:http://www.cbs.dtu.dk/services/EasyGene/
根据核酸序列预测原核生物的基因,有在线分析工具和下载工具两个版本,在线分析工具需要提交序列并且选择模板微生物,以R-value反应提交的序列是否为具有蛋白质编码ORF。

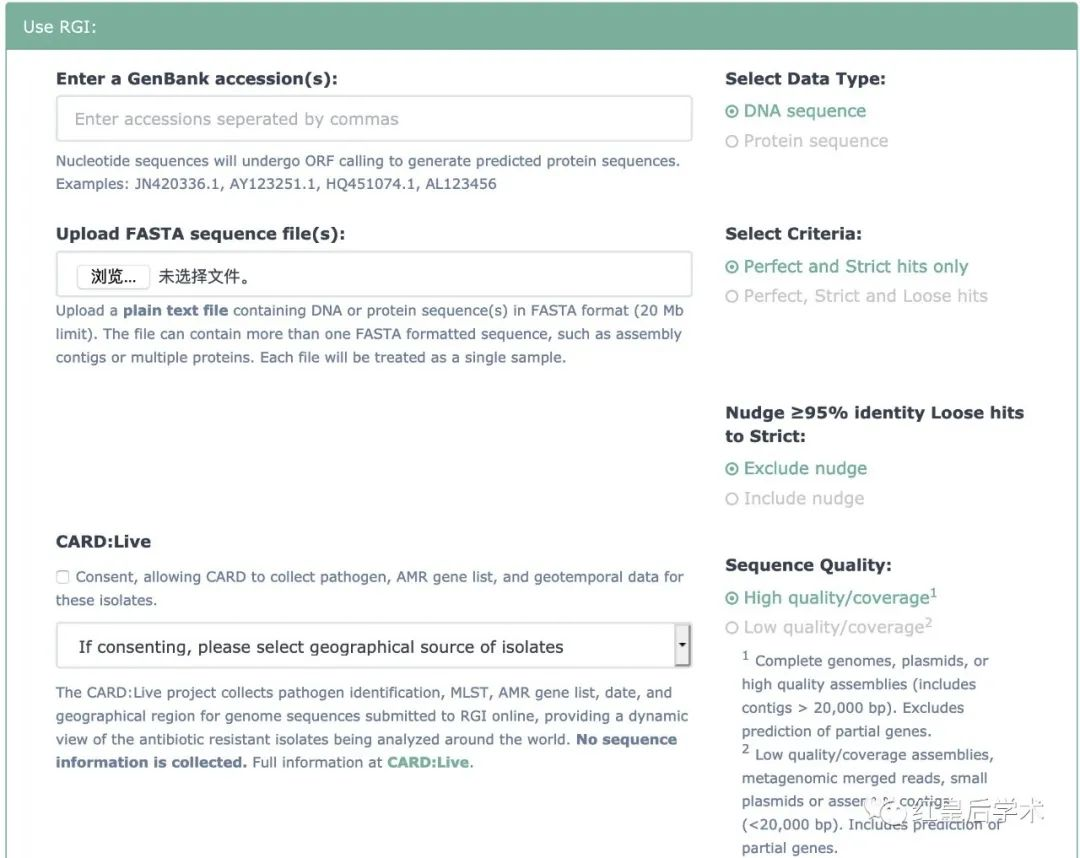
10. Resistance Gene Identifier
网址:https://card.mcmaster.ca/analyze/rgi
基于CARD对DNA序列进行ARGs自动注释的在线分析工具。
11. TestCode
网址:http://bioinformatics.org/sms/testcode.html
在线ORF识别工具。

12. ORFinder
网址:https://www.ncbi.nlm.nih.gov/orffinder/
在线ORF识别工具。

13. REPuter
网址:https://bibiserv.cebitec.uni-bielefeld.de/reputer?id=reputer_view_submission
基因组DNA重复序列识别在线分析工具,最多提交数据量为5Mb。

14. IslandPick
网址:http://www.pathogenomics.sfu.ca/islandviewer2/run_islandpick.php
通过细菌基因组的比较鉴定基因岛并对其进行可视化的在线工具。

15. RNAmmer
网址:http://www.cbs.dtu.dk/services/RNAmmer/
预测核酸序列中的5S/8S、16S/18S和23S/28S核糖体RNA。

16. siDirect
网址:http://sidirect2.rnai.jp/
siRNA在线设计工具。

17. tRNAscan-SE
网址:http://lowelab.ucsc.edu/tRNAscan-SE/
注释序列中的tRNA基因。

18. DINAMelt
网址:http://unafold.rna.albany.edu/?q=DINAMelt
核酸杂交和融解谱图预测工具。

19. Mfold
网址:http://unafold.rna.albany.edu/?q=mfold
DNA和RNA折叠预测工具。

20. RNAStructure
网址:http://rna.urmc.rochester.edu/RNAstructureWeb/
RNA二级结构预测工具。

21. VecScreen
网址:https://www.ncbi.nlm.nih.gov/tools/vecscreen/
去除核酸序列中的载体序列。

22. Prophinder
网址:http://aclame.ulb.ac.be/Tools/Prophinder/
基于ACLAME注释的细菌基因组前噬菌体在线识别工具。

23. SigniSite
网址:http://www.cbs.dtu.dk/services/SigniSite/
分析氨基酸残基中基因型和表型的相关性,通过多序列比对文件预测与表型相关的氨基酸残基的显著性,提交的多序列比对文件不能超过500000个氨基酸残基,工具只能识别20个proteogenic氨基酸和gap,其它氨基酸残基不能识别。

24. LipoP
网址:http://www.cbs.dtu.dk/services/LipoP/
识别革兰氏阴性基因中的脂蛋白及其信号肽,最多可同时提交5000条序列,每条序列的氨基酸个数要超过70同时低于5000。

25. TatP
网址:http://www.cbs.dtu.dk/services/TatP/
预测细菌中的Twin-arginine信号肽的剪切位点,最多提交4000条序列,每条序列不超过5000个氨基酸。

26. NetCorona
网址:http://www.cbs.dtu.dk/services/NetCorona/
预测Coronavirus 3C-like蛋白酶剪切位点,最多提交10条序列,每条序列不超过10000个氨基酸。

27. NetPhosBac
网址:http://www.cbs.dtu.dk/services/NetPhosBac-1.0/
预测细菌中丝氨酸和苏氨酸的磷酸化位点,最多提交2000条序列,每条序列不超过6000个氨基酸。

28. ArchaeaFun
网址:http://www.cbs.dtu.dk/services/ArchaeaFun/
预测古菌序列对应的酶分类,最多同时提交10条序列,每条序列不低于15同时不超过4000个氨基酸。

29. CDART
网址:https://www.ncbi.nlm.nih.gov/Structure/lexington/lexington.cgi
NCBI下属的保守蛋白质结构域在线识别工具。

30. TMHMM
网址:http://www.cbs.dtu.dk/services/TMHMM/
蛋白质跨膜α-螺旋在线预测工具

31. PSORTb
网址:http://www.psort.org/psortb/
细菌蛋白质亚细胞位置在线预测工具,最多一次可以同时预测100个蛋白质。

32. SignalP
网址:http://www.cbs.dtu.dk/services/SignalP/
预测氨基酸序列中的信号肽裂解位点的在线分析工具。

33. I-TASSER
网址:https://zhanglab.ccmb.med.umich.edu/I-TASSER/
同时预测蛋白质3D结构和功能的在线分析工具。

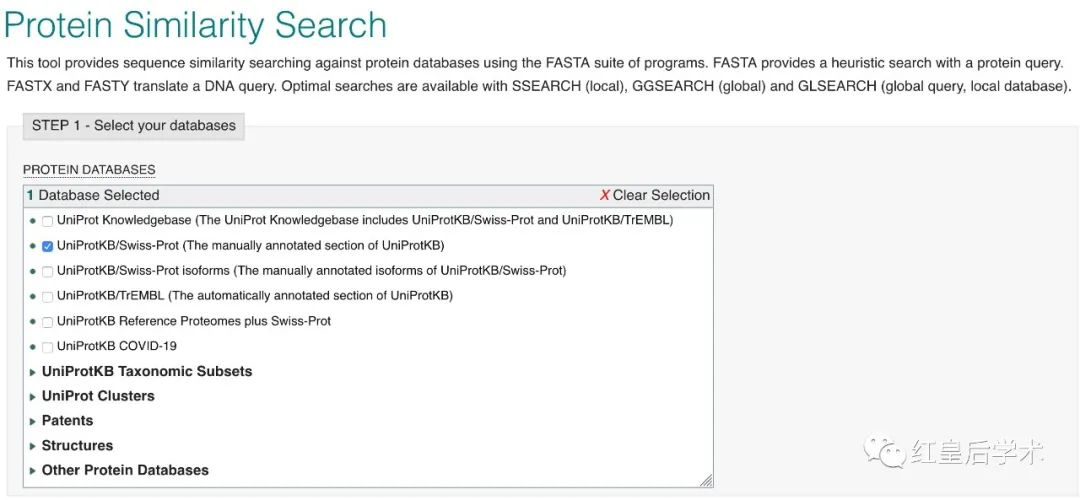
34. FASTA
网址:https://www.ebi.ac.uk/Tools/sss/fasta/
蛋白质序列相似性搜索,类似于BLASTP,参考数据库为Swiss-Prot。
35. NetSurfP
网址:http://www.cbs.dtu.dk/services/NetSurfP/
预测氨基酸序列的表面可接近性和二级结构,表面可接近性以Z-score表示,最多同时提交1500条序列。

36. NetTurnP
网址:http://www.cbs.dtu.dk/services/NetTurnP/
预测蛋白质序列中是否存在β-转角。

37. PSIPRED
网址:http://bioinf.cs.ucl.ac.uk/psipred/
蛋白质二级结构预测工具。
38. Jpred
网址:http://www.compbio.dundee.ac.uk/jpred/index.html
蛋白质二级结构预测工具。

39. SWISS-MODEL
网址:https://swissmodel.expasy.org/
在线蛋白质三维结构同源建模工具。

40. CPHmodels
网址:http://www.cbs.dtu.dk/services/CPHmodels/
在线的蛋白质三维结构同源建模工具,不需要提供模板信息,只需要提供待分析序列,会自动搜索数据库进行建模。

41. Phyre
网址:http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index
蛋白质折叠识别在线工具,根据氨基酸序列识别蛋白质的折叠结构。


42. ENDscript
网址:http://espript.ibcp.fr/ESPript/cgi-bin/ENDscript.cgi
蛋白质一级到四级结构的综合在线分析工具,结果中会用不同的颜色表示氨基酸序列中不同的二级结构单元,每个残基的溶剂可接触性及hydropathy scales,二硫键的位置,与同源蛋白质的多序列比对结果以及保守区域,同源蛋白质中已知的二级结构单元,蛋白质的三维结构其中不同的保守区域应用不同的颜色表示,待分析序列与其它同源蛋白质主链中α-C的差异。

43. SPPIDER
网址:http://sppider.cchmc.org/
蛋白质相互作用位点在线预测工具。

44. InterProSurf
网址:http://curie.utmb.edu/prosurf.html
在线的蛋白质预测工具,预测PDB数据库中的蛋白质选择PDB Complex,预测用户自己的蛋白质选择User Complex。

45. PDBeFold
网址:https://www.ebi.ac.uk/msd-srv/ssm/cgi-bin/ssmserver
蛋白质三维结构比对工具,可以比较两个蛋白质,也可同时比较多个蛋白质,还可以进行蛋白质结构域PDB或SCOP数据库的结构比对。

46. Proteins Plus
网址:https://proteins.plus/
蛋白质活性口袋预测在线分析工具。

47. Active Site Prediction
网址:http://www.bioinformatics.cm-uj.krakow.pl/activesite/
基于Fuzzy-oil-drop模型的蛋白质功能位点识别的在线分析工具。

48. Primer3
网址:http://bioinfo.ut.ee/primer3/
PCR引物在线设计工具

49. Splign
网址:https://www.ncbi.nlm.nih.gov/sutils/splign/splign.cgi?textpage=online&level=form
mRNA与基因组序列比对的在线分析工具。

50. RNAfold
网址:http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi
RNA或DNA二级结构预测工具。

51. APSSP
网址:http://crdd.osdd.net/raghava/apssp/
利用氨基酸序列预测蛋白质的二级结构。

52. Blast2Fasta
网址:http://imed.med.ucm.es/Tools/blast2fasta.html
将BLAST输出格式转换成FASTA格式。

53. BoxShade
网址:https://embnet.vital-it.ch/software/BOX_form.html
多序列比对文件美化工具,可以对MSF和ALN格式文件进行修改,输出格式可以为Postscript/EPS、RTF、XFIG、ASCII、HPGL、PICT。

54. CFSSP
网址:http://www.biogem.org/tool/chou-fasman/
应用Chou & Fasman算法预测氨基酸序列的二级结构。

55. ClustalW
网址:https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_clustalw.html
在线多序列比对工具。

56. COILS
网址:https://embnet.vital-it.ch/software/COILS_form.html
预测蛋白质序列中的无规卷曲。

57. Coiled-Coils prediction
网址:https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_lupas.html
预测蛋白质序列中的无规卷曲。

58. Compute pI/Mw
网址:https://web.expasy.org/compute_pi/
计算蛋白质的等电点和分子质量。

59. ColorSeq
网址:https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_color.html
对蛋白质序列中特定的氨基酸残基进行颜色标注。

60. DAS-TMfilter
网址:http://mendel.imp.ac.at/sat/DAS/DAS.html
预测蛋白质的跨膜结构域。

61. DendroUPGMA
网址:http://genomes.urv.cat/UPGMA/
利用序列差异、相似性矩阵或距离矩阵进行UPGMA聚类分析,对于应用差异的分析输入文件要为fasta格式,对于相似性军阵和距离矩阵分析,输入文件要为CSV格式的矩阵文件。

62. Decrease Redundancy
网址:https://web.expasy.org/decrease_redundancy/
对于一系列序列文件进行去冗余。

63. Dialign
网址:https://bibiserv.cebitec.uni-bielefeld.de/dialign?id=dialign_view_id
氨基酸、DNA、RNA多序列比对工具。

64. DisEMBL
网址:http://dis.embl.de/
预测蛋白质的不规则区域。

65. ESTScan
网址:https://myhits.sib.swiss/cgi-bin/estscan
检测DNA序列中的蛋白质编码区域,同时能够检测和修正导致移码的测序错误,该工具不是一个基因预测工具,而是一个ORF识别工具,但是有别于其他ORF识别工具,该工具可以识别不完成的ORF。

66. FindMod
网址:https://web.expasy.org/findmod/
预测蛋白质中的后转录修饰和单氨基酸取代。

67. GENO3D
网址:https://geno3d-prabi.ibcp.fr/cgi-bin/geno3d_automat.pl?page=/GENO3D/geno3d_home.html
蛋白质三维结构预测工具。

68. GlobPlot
网址:http://globplot.embl.de/
蛋白质domain、globularity、disorder预测工具。

69. GPS
网址:http://gps.biocuckoo.cn/online.php
蛋白质激酶磷酸化位点预测。

70. HAMAP-scan
网址:https://hamap.expasy.org/
蛋白质家族注注释工具。

71. CCTOP
网址:http://cctop.enzim.ttk.mta.hu/?_=/jobs/submit
蛋白质跨膜螺旋区域及其拓扑学预测工具。
72. Isotopident
网址:http://education.expasy.org/student_projects/isotopident/htdocs/
估计多肽、蛋白质、核酸、化学化合物的同位素分布情况。

73. LALIGN
网址:https://embnet.vital-it.ch/software/LALIGN_form.html
两条序列配对比对工具。

74. One to Three
网址:http://bioinformatics.org/sms2/one_to_three.html
将以单字母简写形式表示的氨基酸序列转换为三字母简写格式。

75. Paircoil2
网址:http://cb.csail.mit.edu/cb/paircoil2/paircoil2.html
预测蛋白质序列中平行的coiled coil。

76. PeptideCutter
网址:https://web.expasy.org/peptide_cutter/
根据蛋白质序列预测特定蛋白酶或化学物质可能的裂解位点。

77. Phobius
网址:http://phobius.sbc.su.se/
跨膜结构拓扑和信号肽预测工具。

78. Phylogibbs
网址:https://phylogibbs.unibas.ch/cgi-bin/phylogibbs.pl?part=runit&page=advanced
DNA序列调控位点预测工具。

79. PRATT
网址:https://www.ebi.ac.uk/Tools/pfa/pratt/
发现蛋白质序列中保守patterns的工具。

80. ProSA-web
网址:https://prosa.services.came.sbg.ac.at/prosa.php
蛋白质3D结构模型合理性检测工具。

81. ProtParam
网址:https://web.expasy.org/protparam/
计算蛋白质的分子质量、pl、氨基酸组成、原子组成、灭绝稀疏、半衰期、亲水性等物理化学性质。
82. ProtScale
网址:https://web.expasy.org/protscale/
蛋白质亲水性范围、二级结构构想参数范围计算。
83. RADAR
网址:https://www.ebi.ac.uk/Tools/pfa/radar/
蛋白质序列中重复序列的检测和比对工具。

84. Reverse Translate
网址:http://www.bioinformatics.org/sms2/rev_trans.html
根据蛋白质序列反转录生成DNA序列。

85. SAPS
网址:https://www.ebi.ac.uk/Tools/seqstats/saps/
蛋白质序列组成差异、电荷和氨基酸类型聚类,重复结构的差异,周期性的模体和相同残基之间的不规则间隙统计。

86. STRING
网址:https://string-db.org/
蛋白质相互作用识别。

87. BioVenn
网址:https://www.biovenn.nl/
在线的Venn图绘制工具,一张图中最多可以有三个样品,可以修改字号,名称和颜色。

88. Draw Venn Diagram
网址:http://bioinformatics.psb.ugent.be/webtools/Venn/
在线的Venn图绘制工具,可以绘制三个以上的样品,分为对称和不对称两种模式。

89. Venny
网址:https://bioinfogp.cnb.csic.es/tools/venny/index.html
在线的Venn图绘制工具,最多公式绘制4个样品。

90. jvenn
网址:http://jvenn.toulouse.inra.fr/app/example.html
在线Venn图绘制工具,最多可以绘制6个/组样品。

91. Orphelia
网址:http://orphelia.gobics.de/submission
宏基因组数据蛋白质编码基因在线预测工具。
92. MetaBioME
网址:https://metasystems.riken.jp/metabiome/
基于web的宏基因组和细菌全基因数据中新型的商业可利用酶预测工具。

93. WEGO
网址:http://wego.genomics.org.cn/
基于web的在线宏基因组数据GO富集及绘图工具。

94. iPath
网址:https://pathways.embl.de/
基于web的交互式KEGG代谢路径可视化、分析和自构建在线工具。

95. Anti-SMASH
网址:https://antismash.secondarymetabolites.org/#!/start
细菌次级代谢路径在线分析工具,次级代谢产物包括抗生素、激素、生物碱、毒素、维生素等。

96. CAP3
网址:http://doua.prabi.fr/software/cap3
用于序列拼接的在线工具,上传序列不超过50kb。

97. PathPred
网址:https://www.genome.jp/tools/pathpred/
细菌外源污染物代谢路径在线预测工具。

98. PhyML
网址:http://www.atgc-montpellier.fr/phyml/
应用最大拟然法进行系统发育分析的在线工具。

99. iToL
网址:https://itol.embl.de/index.shtml
系统发育树在线美化工具。

100. Mapbox
网址:https://www.mapbox.com/
可以通过添加图层的方式在各种风格的地图上绘制自定义地图的在线工具。

猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
文献阅读 热心肠 SemanticScholar Geenmedical
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读























 520
520

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








