通过查阅文章找到marker gene后手动注释分群,如何检验分群正确与否?

这个图是通过Seurat标准流程降维后得到的muscle组织分群图
然后查阅文章得到muscle组织不同细胞的markergene
genes_VEC = c("Fabp4", "Cdh5", "Cav1") #vascular endothelial cells
genes_FC = c("Ddr2","Tcf21", "Col3a1", "Col1a2", "Col1a1") #fibroblasts
genes_AC = c("Nppa", "Myl7", "Sln") #"Nppa", "Myl7" not found #atrial cardiomyocytes
genes_EC = c("Npr3", "Pecam1") #endocardial cells
genes_IC = c("C1qa", "H2-Eb1") #immune cells
genes_SMSC= c("Myf5", "Myod1","Sox9","Acta2","Chodl") #skeletal muscle satellite cell
genes_MSC= c("Pdgfra","Chad") #mesenchymal stem cell
genes_endothelialC=c("Atxn1")
genes_TmB = c('Ptprc')
genes_macrophage=c("Itgam")
genes_T=c("Cd3g","Cd4")
genes_B=c("Cd19")
genes_others=c("Acan")
genes_all = c(genes_VEC, genes_FC, genes_AC,genes_EC,genes_IC,genes_SMSC,genes_MSC,genes_endothelialC,genes_TmB,genes_macrophage,genes_T,genes_B,genes_others)
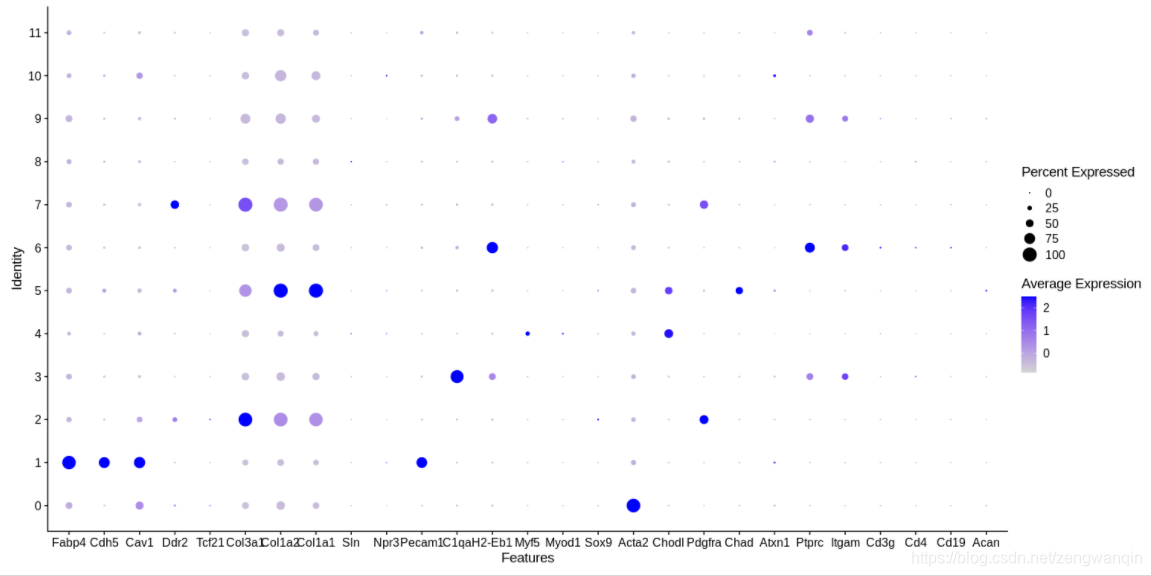
以下三幅图片都是想表达不同的markergene的表达情况:
FeaturePlot(tiss, genes_all, pt.size = 1,ncol = 4)

DotPlot(tiss, features = genes_all)
VlnPlot(tiss, genes_all, ncol = 4)

有了这三幅图,我们可以对这些muscle细胞进行一个手动的注释,得到下面的细胞分群图:
#annotaion
tiss@meta.data$free_annotation <- plyr::mapvalues(from = c(1,2,5,7,8,10,11,3,6,9,0,4),
to = c("vascular endothelial cells", rep("fibroblasts",6), rep("immune cells", 3),rep("skeletal muscle satellite cells", 2)),
x = tiss@meta.data$seurat_clusters)
TSNEPlot(object = tiss, group.by = "free_annotation")

但是这个分群图正确与否应该怎么判断呢?
检验分群正确与否方法
- Findmarkers 函数
- singleR
- Seruat4.0
一、Findmarkers 函数
首先需要加载所需的R包和数据集
library(Seurat)
tiss<- #数据集加载
以下为自己数据示例
levels(tiss)
markers_df <- FindMarkers(object = tiss, ident.1 = 1, min.pct = 0.25)#一号群
markers_df #Fabp4", "Cdh5", "Cav1"文章中提到的
print(x = head(markers_df))
markers_genes = rownames(head(x = markers_df, n = 5))
VlnPlot(object = tiss, features =markers_genes,log =T )
FeaturePlot(object = tiss, features=markers_genes )


这样我们可以得到每一个分群的markergene,可以大概看一下这些markergene与文献中提到的有没有重合的。这个只能当作一种检验方法吧。
二、SingleR
singleR自带7个数据库文件,需要联网才能下载,其中5个是人类数据,2个是小鼠的数据:
-
BlueprintEncodeData Labels
-
HumanPrimaryCellAtlasData Labels
-
DatabaseImmuneCellExpressionData Labels
-
NovershternHematopoieticData Labels
-
MonacoImmuneData Labels
-
ImmGenData Labels
-
MouseRNAseqData Labels
本文以MouseRNAseqData 数据集为例:
首先加载参考数据集
# load ref
library("SingleR")
cg <- MouseRNAseqData()
cg
以下为加载测试数据集的过程:
#load test
library("openxlsx")
library("ggplot2")
library("Matrix")
mypwd <- "/mnt/raid64/Mouse_iso_atlas/analysis/SingleCell/NGS_ONT_analysis/01.Seurat/"
library("Seurat", lib.loc = "/home/zhangdan/R/x86_64-pc-linux-gnu-library/4.0")
source(paste0(mypwd, "00_data_ingest/02_tissue_analysis_rmd/boilerplate.R"))
tissue_of_interest = 'muscle'
process_tissue = function(tiss, scale){
tiss <- NormalizeData(object = tiss, scale.factor = scale)
tiss <- FindVariableFeatures(object = tiss, do.plot = TRUE, x.high.cutoff = Inf, y.cutoff = 0.5)
tiss <- ScaleData(object = tiss)
tiss <- RunPCA(object = tiss, do.print = FALSE)
}
load_tissue_singleron = function(tissue_of_interest){
singleron_metadata_filename = paste0(mypwd, "00_data_ingest/00_singleron_raw_data/metadata_singleron.csv")
singleron_metadata <- read.csv(singleron_metadata_filename, sep=",", header = TRUE)
tissue_metadata <- singleron_metadata[singleron_metadata$Tissue == tissue_of_interest, ]
subfolder = tissue_metadata$Sample
raw.data <- Read10X(data.dir = paste0(mypwd, "00_data_ingest/00_singleron_raw_data/singleron/", subfolder[1]))
# Create the Seurat object with all the data
tiss <- CreateSeuratObject(counts = raw.data, project = tissue_of_interest)
tiss[["percent.mt"]] <- PercentageFeatureSet(tiss, pattern = "^mt-")
tiss[["percent.ribo"]] <- PercentageFeatureSet(tiss, pattern = "^Rp[sl][[:digit:]]")
tiss@meta.data[,'free_annotation'] <- NA
tiss[["Tissue"]] <- tissue_metadata[1,]$Tissue
tiss[["Sex"]] <- tissue_metadata[1,]$Sex
tiss[["Strain"]] <- tissue_metadata[1,]$Strain
tiss[["mouse.id"]] <- tissue_metadata[1,]$mouse.id
VlnPlot(tiss, features = c("nFeature_RNA", "nCount_RNA", "percent.mt","percent.ribo"), ncol = 2)
#tiss <- subset(tiss, subset = nFeature_RNA > 200 & nFeature_RNA < 6000 & percent.mt < 5)
tiss <- process_tissue(tiss, 1e4)
return(tiss)
}
tiss = load_tissue_singleron(tissue_of_interest)
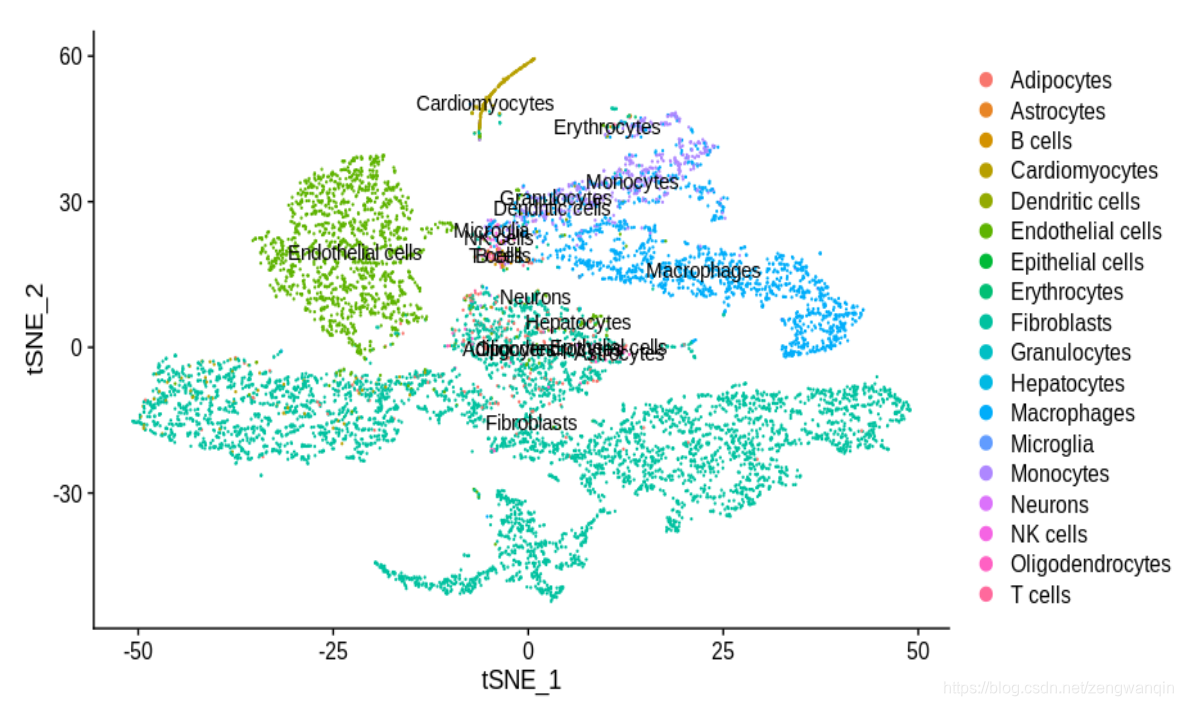
测试数据集通过SingleR比对到参考数据集上:
data <- tiss@assays$RNA@data
pred.hesc <- SingleR(test = data, ref = cg, assay.type.test=1,
labels = cg$label.main)
pred.hesc
#比对结果,会显示有哪些细胞及其细胞数量
# Summarizing the distribution:
table(pred.hesc$labels)

plotScoreHeatmap(pred.hesc)

tiss@meta.data$lables <- pred.hesc$labels
DimPlot(tiss, group.by="lables", label = T)
三、Seurat4.0
还在学习中 后续补上







 本文介绍了一种基于Seurat工具的手动注释肌肉组织细胞分群的方法,并提供了三种检验分群正确性的策略:利用Findmarkers函数寻找标志基因、使用SingleR进行细胞类型预测以及应用Seurat 4.0进行进一步分析。
本文介绍了一种基于Seurat工具的手动注释肌肉组织细胞分群的方法,并提供了三种检验分群正确性的策略:利用Findmarkers函数寻找标志基因、使用SingleR进行细胞类型预测以及应用Seurat 4.0进行进一步分析。
















 1920
1920

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








