scDist通过分析不同状态下细胞的距离来找到差异最大的细胞亚群(见下图的A),然后再分析每一个细胞亚群的PCA通过线性的混合模型并结合最终的系数去预估不同干预方式下细胞群之间的距离。
Augur是通过对每一个细胞进行AUC评分并排序最终找到扰动最佳的细胞群,开发者认为Augur忽略了样本间的异质性,可能会导致由于样本本身的异质性而导致差异的出现。
笔者在回溯Augur代码的时候发现开发者其实已在函数中设置了矫正参数(可以用于批次矫正/样本矫正等),但开发者认为Augur并不能很好的把样本误差更抹去(可以读一读参考资料中的scDist的文章),即使使用了harmony去矫正之后也无法处理的很完美。
但笔者认为我们并不应该去重点关注样本间的差异,因为样本间的差异就应该是存在的,要以一个整体去看待纳入分析的样本,因此不同干预方式去处理了样本之后出现的扰动差异是样本本身的异质性导致的,但是也有可能是真实存在的扰动结果。
因此笔者认为scDist可以作为Augur的辅助分析,来验证Augur的结果。
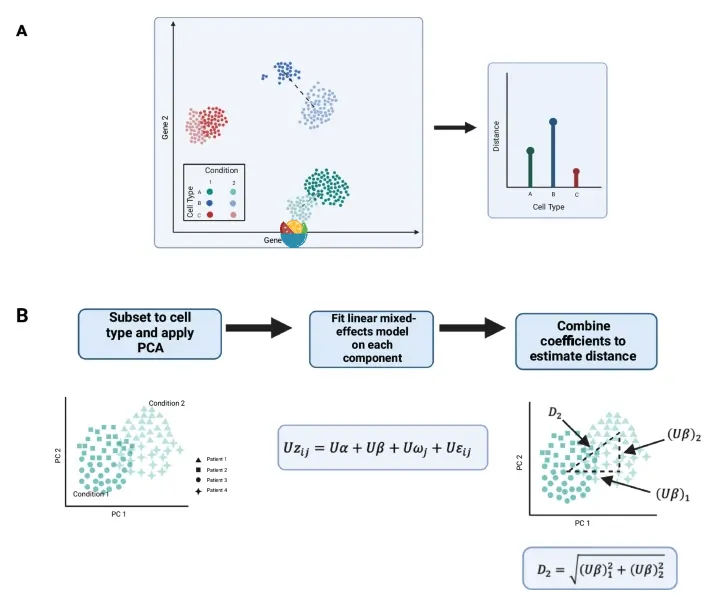
分析流程
1.导入
rm(list = ls())
V5_path = "/Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/seurat5/"
.libPaths(V5_path)
.libPaths()
library(stringr)
library(Seurat)
library(scDist)
library(dplyr)
library(patchwork)
library(viridis)
library(qs)
library(BiocParallel)
register(MulticoreParam(workers = 4, progressbar = TRUE))
load("scRNA.Rdata")
2.数据预处理
# check
table(scRNA$orig.ident)
table(scRNA$celltype)
dat <- scRNA@meta.data
# 增加一下批次信息,转换成数字
dat$batch <- ifelse(grepl("sample2|sample4|sample6", dat$orig.ident), "1", "2")
# 增加一下分组信息,这里是随意编造的
dat$group <- ifelse(grepl("sample[1-3]", dat$orig.ident), "con", "treat")
scRNA@meta.data <- dat
3.scDist分析
dat <- GetAssayData(scRNA,layer = "counts") #其实是需要normalize data,但这样也行
out <- scDist(dat,scRNA@meta.data,
fixed.effects = "group",
random.effects="batch",
clusters="celltype")
qsave(out,"out.qs")
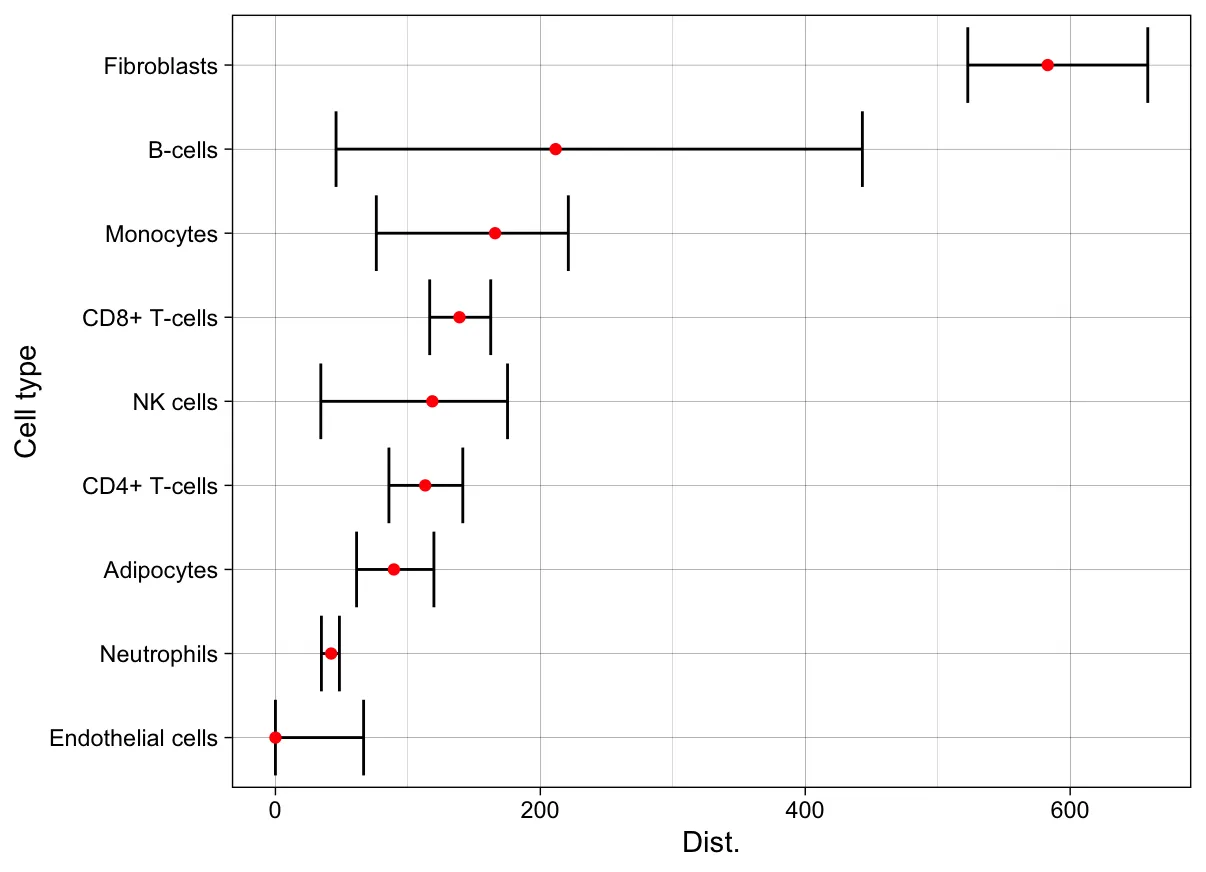
out$results
# Dist. 95% CI (low) 95% CI (upper) p.val
# CD4+ T-cells 113.06663 85.67429 141.45175 0.0000099999
# Fibroblasts 583.03755 522.70365 658.58584 0.0000099999
# B-cells 211.53939 45.76088 443.12152 0.7932920671
# CD8+ T-cells 138.91180 116.46147 162.53086 0.0000099999
# Neutrophils 42.07108 34.69947 48.30078 0.0000099999
# Monocytes 165.83546 76.16444 221.11992 0.0000099999
# Adipocytes 89.43945 61.25896 119.64719 0.0000099999
# NK cells 118.44792 34.25347 175.22504 0.0000099999
# Endothelial cells 0.00000 0.00000 66.57318 0.2874671253
4.可视化
DistPlot(out)
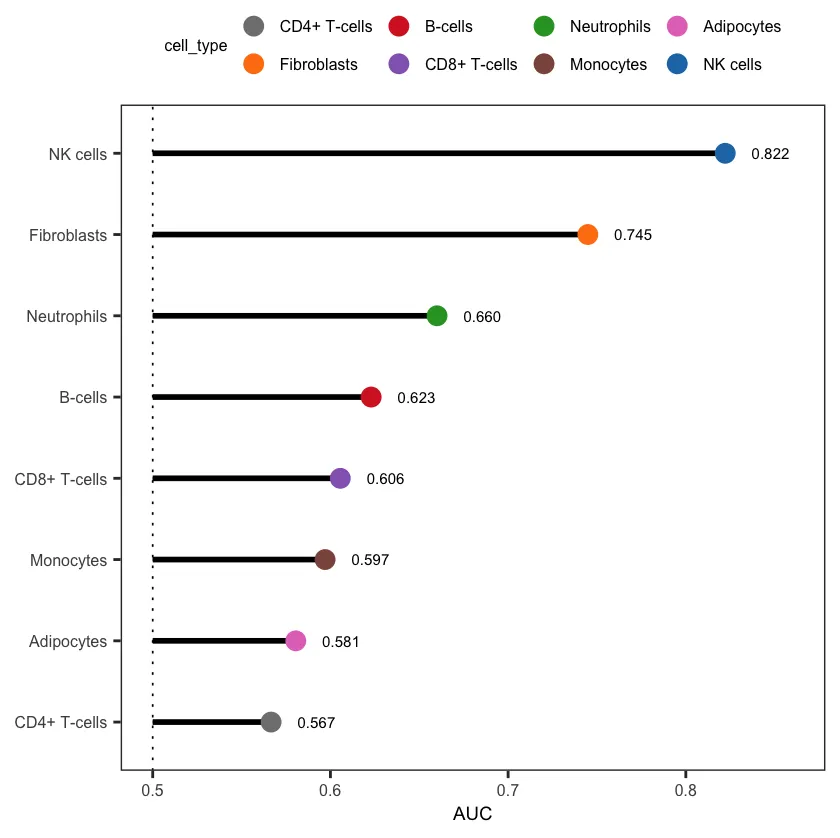
scDist分析结果,细胞的排序和Augur的AUC分数排序存在较大的不同。
Augur分析结果
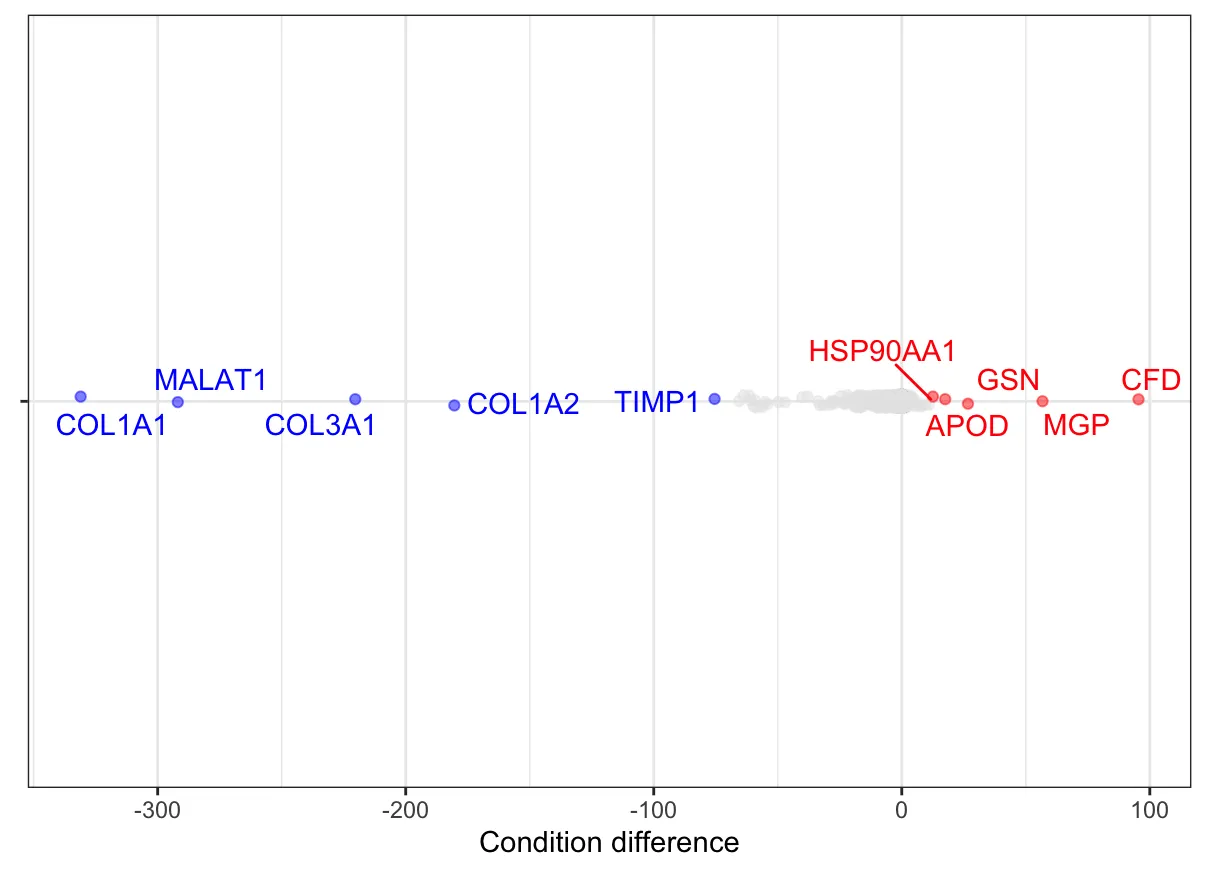
distGenes(out, cluster = "Fibroblasts")
展示一下差异基因
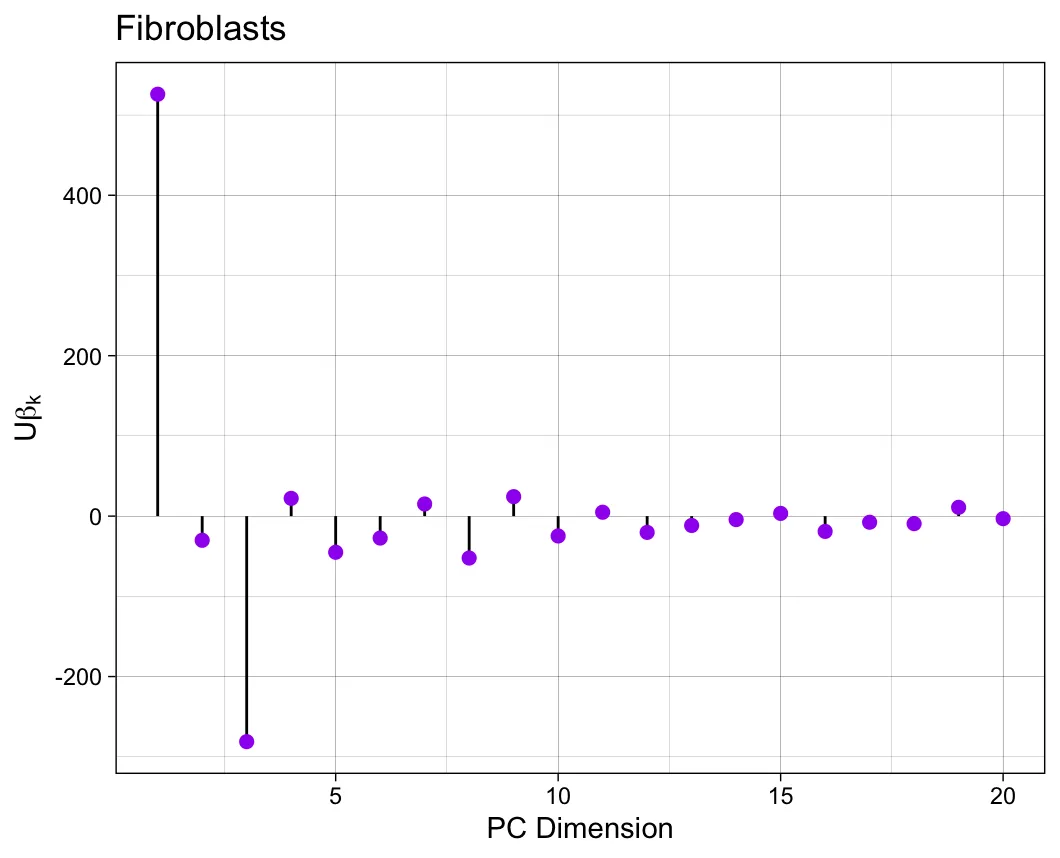
plotBetas(out,cluster = "Fibroblasts")
不同主成分的影响程度
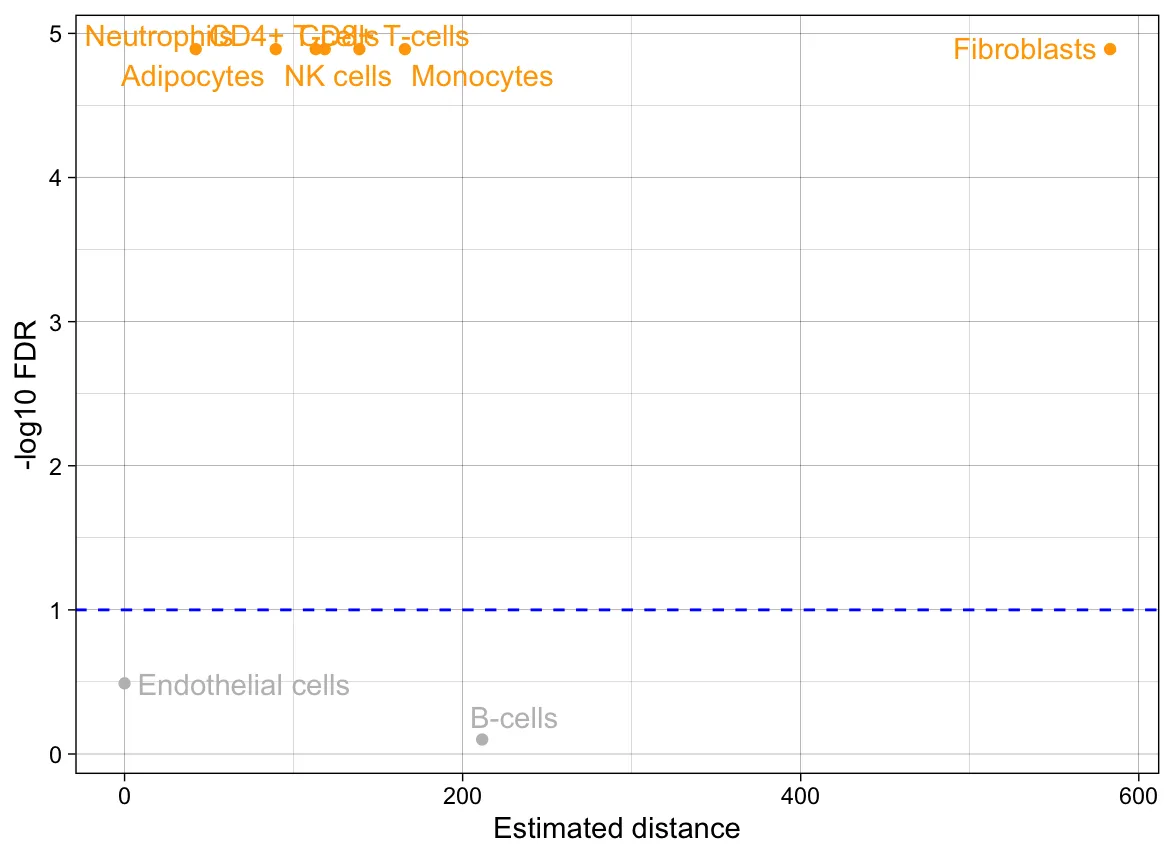
FDRDistPlot(out)
这个图既说明了不同细胞亚群之间的距离差异排序,也说明了不同处理后细胞亚群是否存在统计学差异。
所有的图片都是基于ggplot绘制的,可以提取原始数据自行修改美化。
参考资料:
1、Robust identification of perturbed cell types in single-cell RNA-seq data. Nat Commun.2024 Sep 1;15(1):7610.
2、scDist:
https://github.com/phillipnicol/scDist/tree/master
3、生信碱移:
https://mp.weixin.qq.com/s/N-XEc9xFCnwf4PHbaraGaw
注:若对内容有疑惑或者有发现明确错误的朋友,请联系后台(欢迎交流)。更多内容可关注公众号:生信方舟
- END -






















 13万+
13万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








