OmicsTools软件介绍
我开发了一款本地电脑无限使用的零代码生信数据分析作软图神器电脑软件OmicsTools,欢迎大家使用OmicsTools进行生物医学科研数据分析和作图,该软件件能让大家在不需要任何编程和代码编写的基础上,分析次数没有限制,可以无限使用,让您在自己的电脑上快速进行大量的生信分析和加速大家的科学研究。
我开发的本地电脑无限使用无限分析作图的生信零代码一键分析电脑软件神器OmicsTools 软件在github上的zihaoxingstudy1/OmicsTools仓库中,大家可以下载安装OmicsTools进行各种生信分析和可视化作图。
我开发的在本地电脑无限使用无限分析作图的生信零代码一键分析电脑软件神器OmicsTools 软件下载github官方网址: https://github.com/zihaoxingstudy1/OmicsTools ,也可以到我的生信交流q群931846486的群文件中下载到OmicsTools分析软件,大家可以下载安装OmicsTools进行各种生信分析和可视化作图。


甲基化组学全流程分析和可视化教程
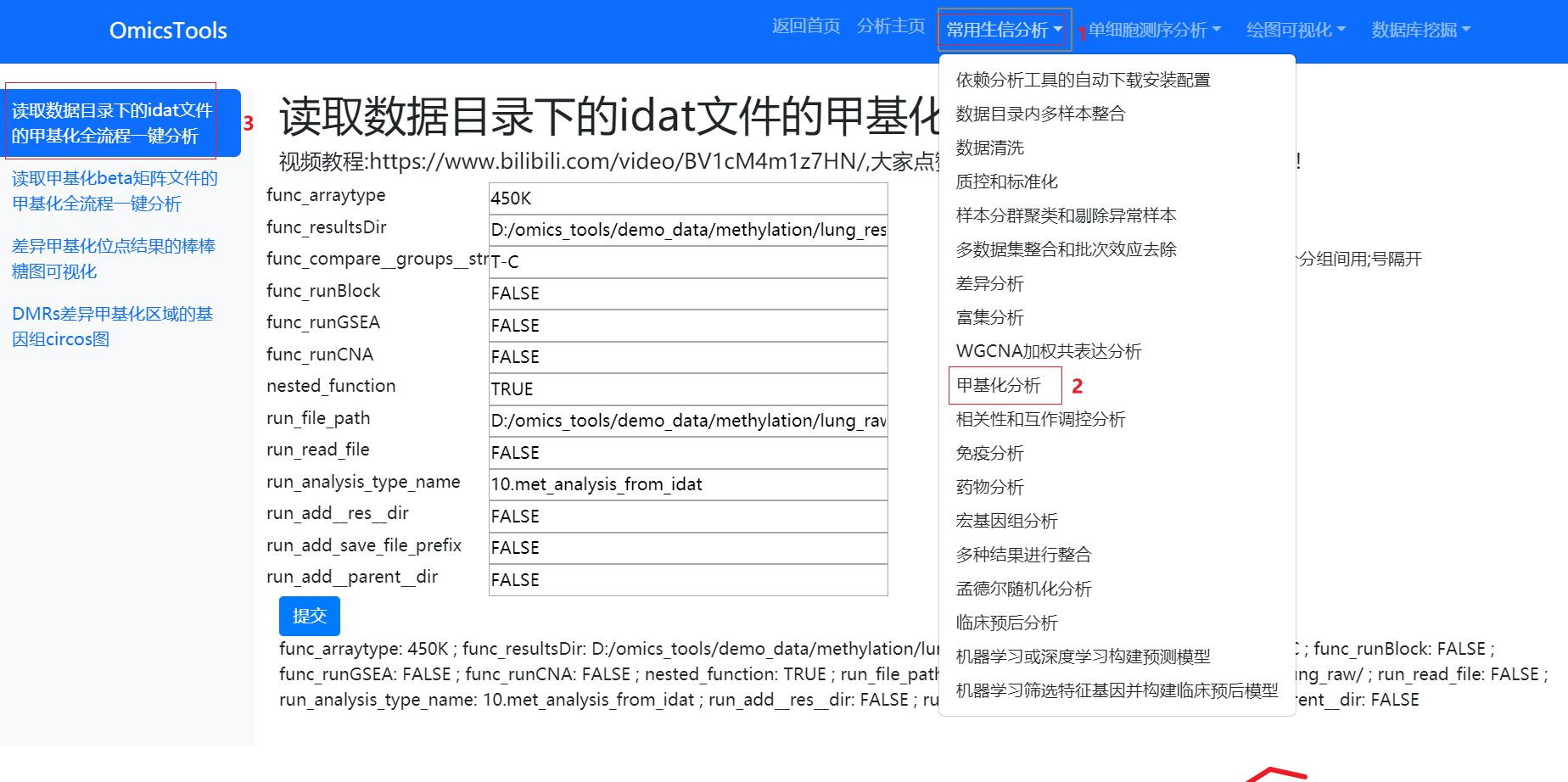
读取数据目录下的idat文件的甲基化全流程一键分析
功能简介
- 甲基化分析模块可以实现甲基化芯片450K, 870kEPIC数据的自动读取,可以读取idat文件,也可以读取beta甲基化矩阵文件
- 甲基化数据的缺失值插值
- 甲基化数据的质控分析
- 甲基化数据的归一化处理分析
- 甲基化数据的SVD分析
- 甲基化数据的Combat去除批次效应分析
- 甲基化数据的DMP差异甲基化位点分析
- 甲基化数据的DMR差异甲基化区域分析
- 甲基化数据的差异block分析
- 甲基化数据的GSEA分析
- 甲基化数据的CNA分析
参数解释
func_arraytype:可选450k或EPIC,EPIC是870K的甲基化芯片
func_resultsDir: 分析结果要保存的目录
func_compare__groups__str: 做DMP和DMR分析时候要指定的比较分组,多个分组间用;号隔开
func_runBlock:是否进行比较耗时的block分析,默认为FALSE
func_runGSEA: 是否进行比较耗时的GSEA分析,默认为FALSE
func_runCNA: 是否进行比较耗时的CNA分析,默认为FALSE
nested_function: 是否嵌套函数
run_file_path: 甲基化.idat格式的原始数据所在的目录
run_read_file:是否要读取文件
run_analysis_type_name: 分析项目名称
run_add__res__dir: 是否要创建res_dir结果目录
run_add_save_file_prefix: 是否要添加结果保存文件的前缀
run_add__parent__dir:是否在上一级目录下创建目录或保存结果
提交
参数给定的默认值
func_arraytype: 450K ;
func_resultsDir: D:/omics_tools/demo_data/methylation/lung_results/ ;
func_compare__groups__str: T-C ; func_runBlock: FALSE ; func_runGSEA: FALSE ; func_runCNA: FALSE ; nested_function: TRUE ;
run_file_path: D:/omics_tools/demo_data/methylation/lung_raw/ ;
run_read_file: FALSE ; run_analysis_type_name: 10.met_analysis_from_idat ;
run_add__res__dir: FALSE ; run_add_save_file_prefix: FALSE ;
run_add__parent__dir: FALSE
窗口截图
运行中的显示信息
执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data/methylation; 分析结果日志保存的路径为: D:/omics_tools/demo_data/methylation\10.met_analysis_from_idat_last_final_run_res_log.csv
运行完成的显示信息
结果展示

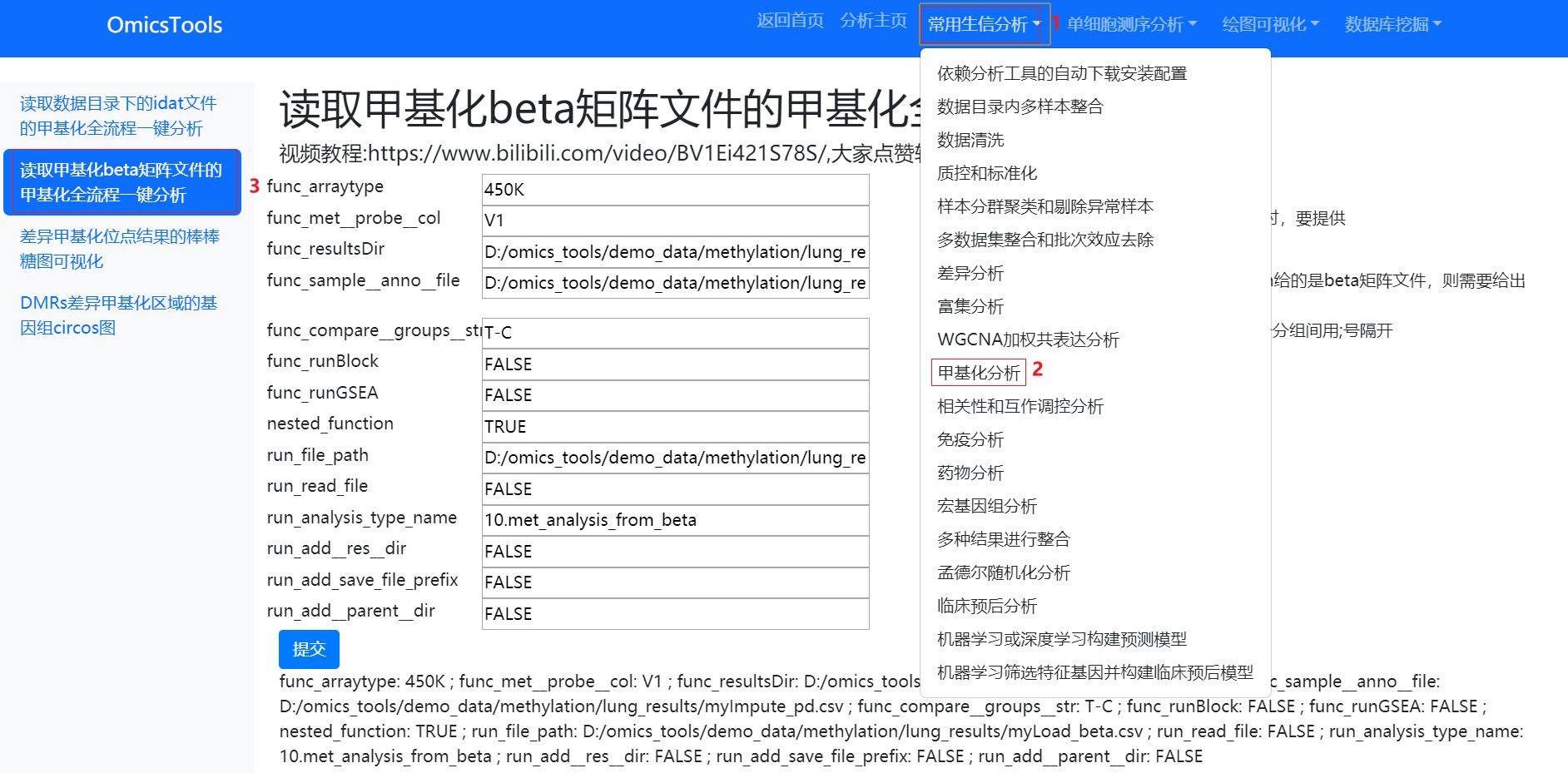
读取甲基化beta矩阵文件的甲基化全流程一键分析
功能简介
- 甲基化分析模块可以实现甲基化芯片450K, 870kEPIC数据的自动读取,可以读取idat文件,也可以读取beta甲基化矩阵文件
- 甲基化数据的缺失值插值
- 甲基化数据的质控分析
- 甲基化数据的归一化处理分析
- 甲基化数据的SVD分析
- 甲基化数据的Combat去除批次效应分析
- 甲基化数据的DMP差异甲基化位点分析
- 甲基化数据的DMR差异甲基化区域分析
- 甲基化数据的差异block分析
- 甲基化数据的GSEA分析
- 甲基化数据的CNA分析
模块使用讲解
参数解释
func_arraytype: 可选450k或EPIC,EPIC是870K的甲基化芯片
func_met__probe__col:甲基化探针所在的列名,当从beta文件开始分析时,要提供
func_resultsDir:分析结果要保存的目录
func_sample__anno__file:样本注释信息的文件,默认是空,如果file_path给的是beta矩阵文件,则需要给出sample.anno.file
func_compare__groups__str:做DMP和DMR分析时候要指定的比较分组,多个分组间用;号隔开
func_runBlock: 是否进行比较耗时的block分析,默认为FALSE
func_runGSEA:是否进行比较耗时的GSEA分析,默认为FALSE
func_runCNA:是否进行比较耗时的CNA分析,默认为FALSE
nested_function:是否嵌套函数
run_file_path:甲基化beta矩阵的文件路径
run_read_file:是否要读取文件
run_analysis_type_name:分析项目名称
run_add__res__dir:是否要创建res_dir结果目录
run_add_save_file_prefix;是否要添加结果保存文件的前缀
run_add__parent__dir:是否在上一级目录下创建目录或保存结果
提交
给定参数的默认值
func_arraytype: 450K ;
func_met__probe__col: V1 ;
func_resultsDir: D:/omics_tools/demo_data/methylation/lung_results/ ; func_sample__anno__file: D:/omics_tools/demo_data/methylation/lung_results/myImpute_pd.csv ; func_compare__groups__str: T-C ; func_runBlock: FALSE ;
func_runGSEA: FALSE ;
func_runCNA: FALSE ;
nested_function: TRUE ;
run_file_path: D:/omics_tools/demo_data/methylation/lung_results/myLoad_beta.csv ;
run_read_file: FALSE ;
run_analysis_type_name: 10.met_analysis_from_beta ;
run_add__res__dir: FALSE ;
run_add_save_file_prefix: FALSE ;
run_add__parent__dir: FALSE
窗口截图
运行中的显示信息
分析正在执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data/methylation/lung_results; 运行结果日志保存的路径为: D:/omics_tools/demo_data/methylation/lung_results\10.met_analysis_from_beta_last_final_run_res_log.csv
结果展示
样本的PCA分群聚类分析和剔除异常样本
样本的PCA分群聚类分析
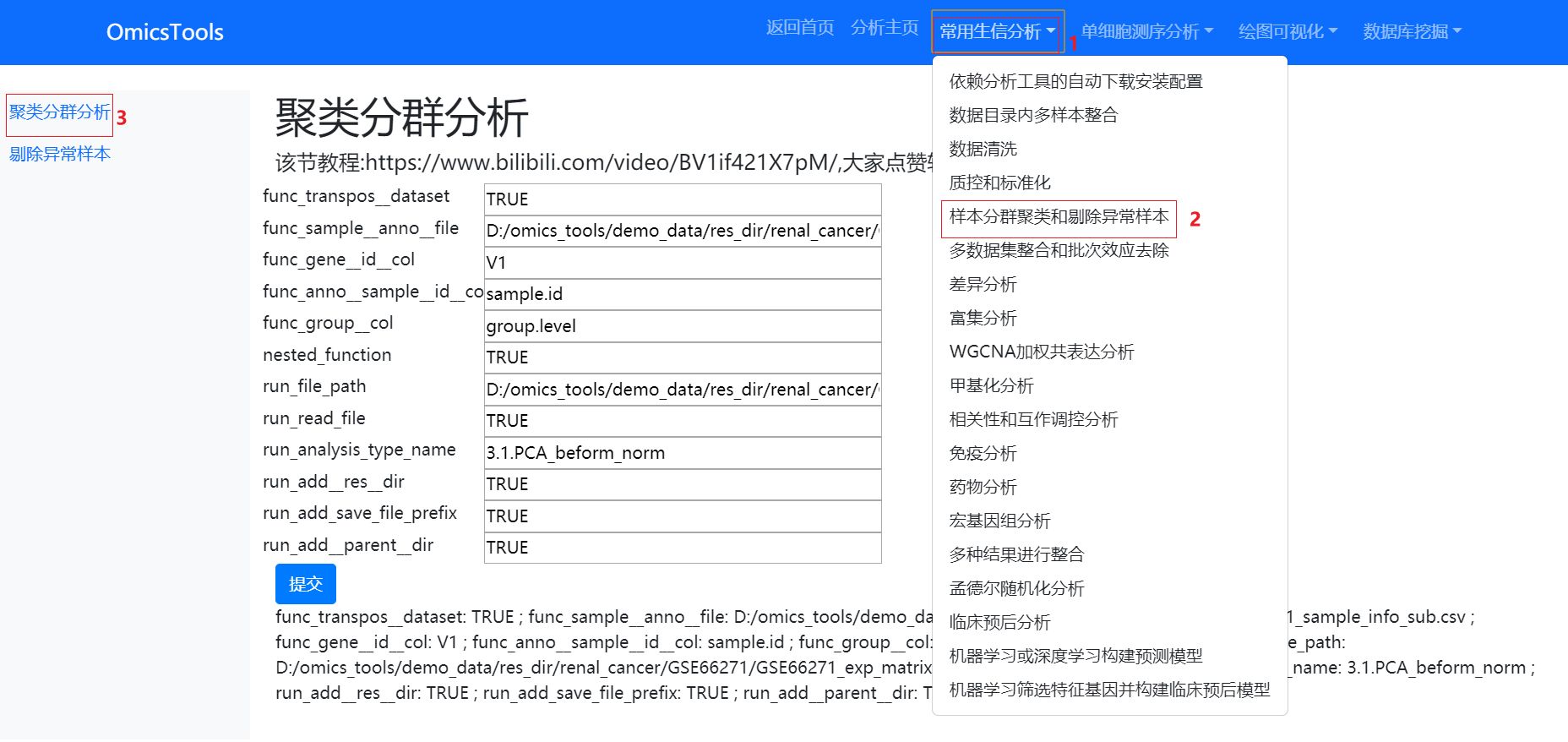
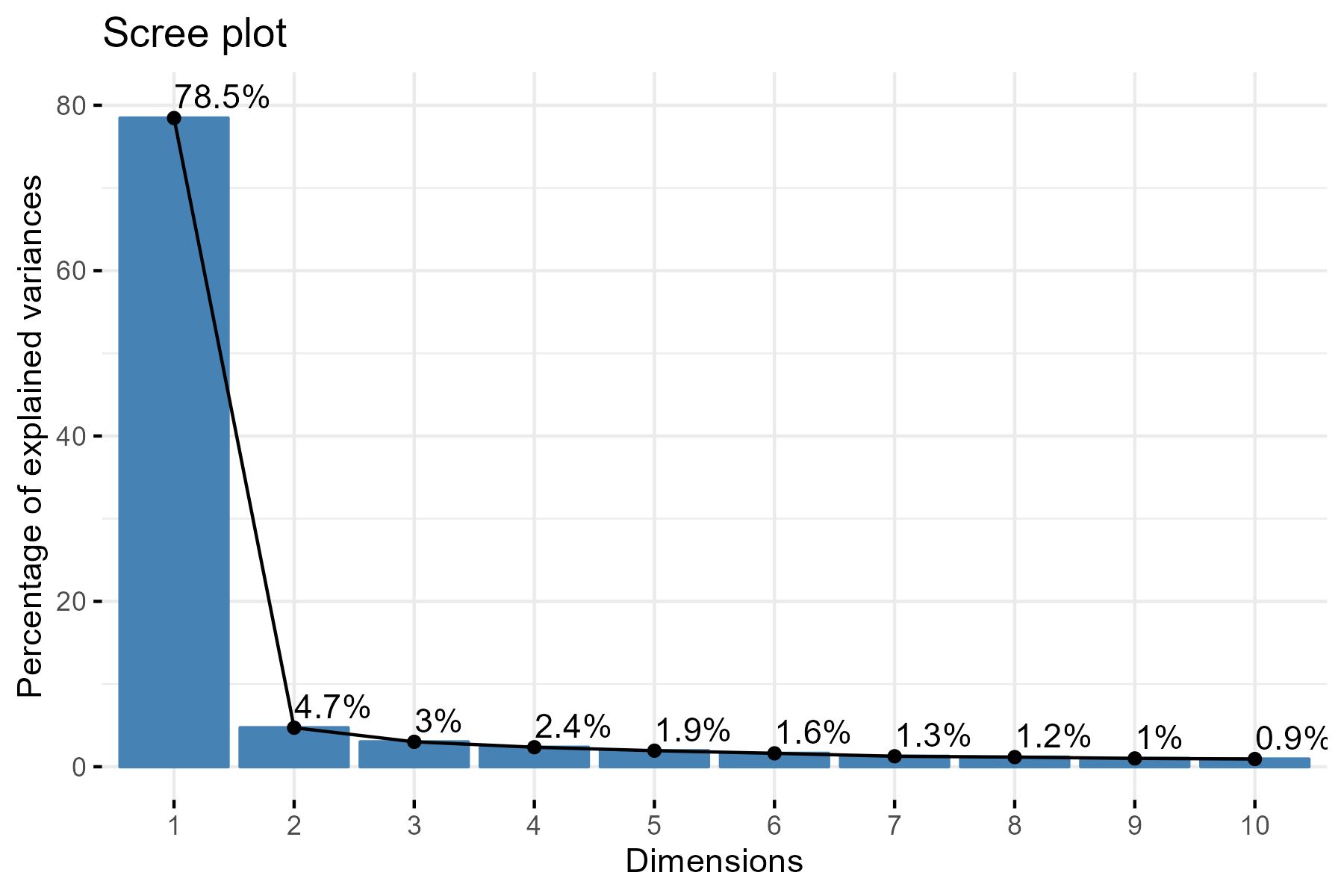
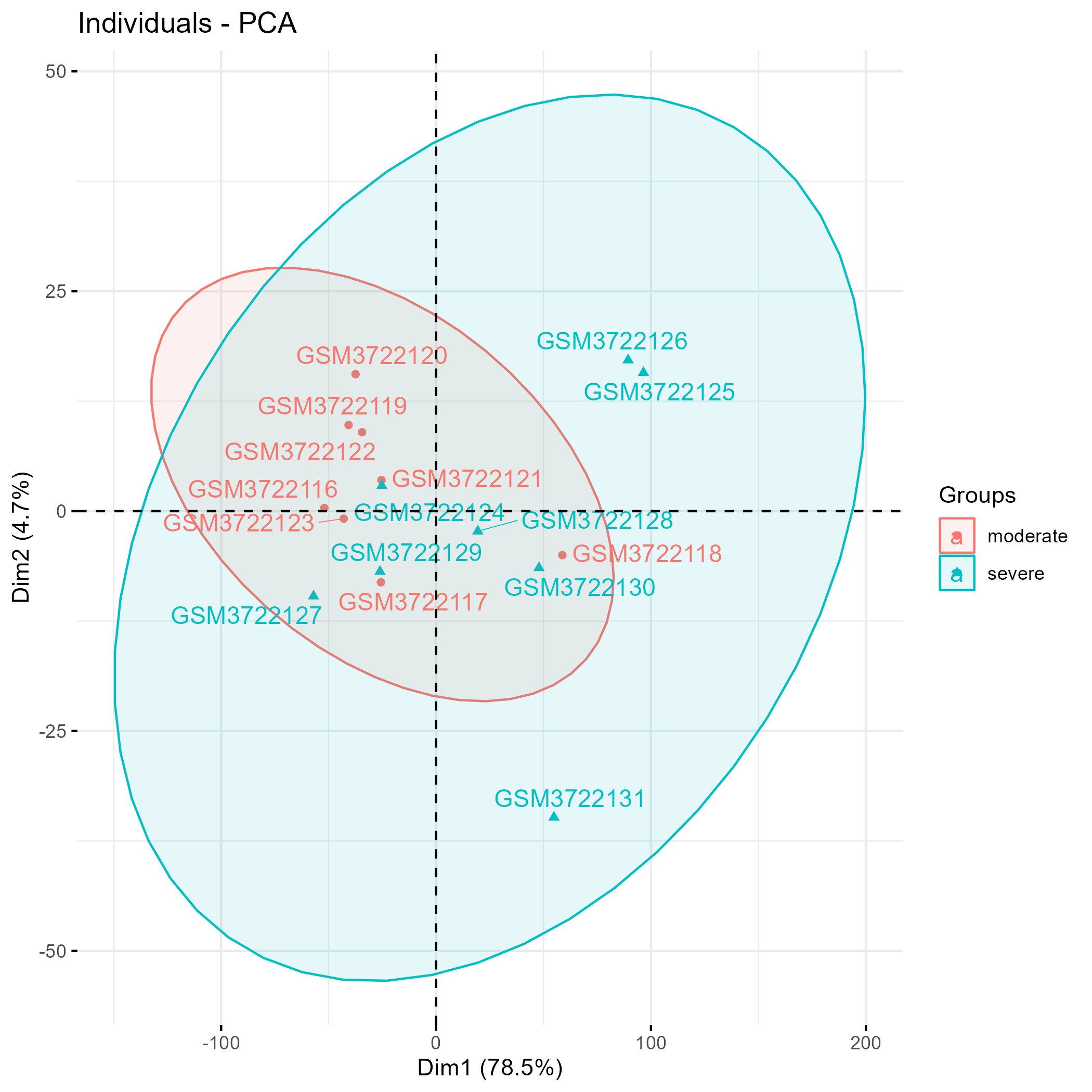

删除异常样本

剔除异常样本后再次进行PCA分群聚类分析


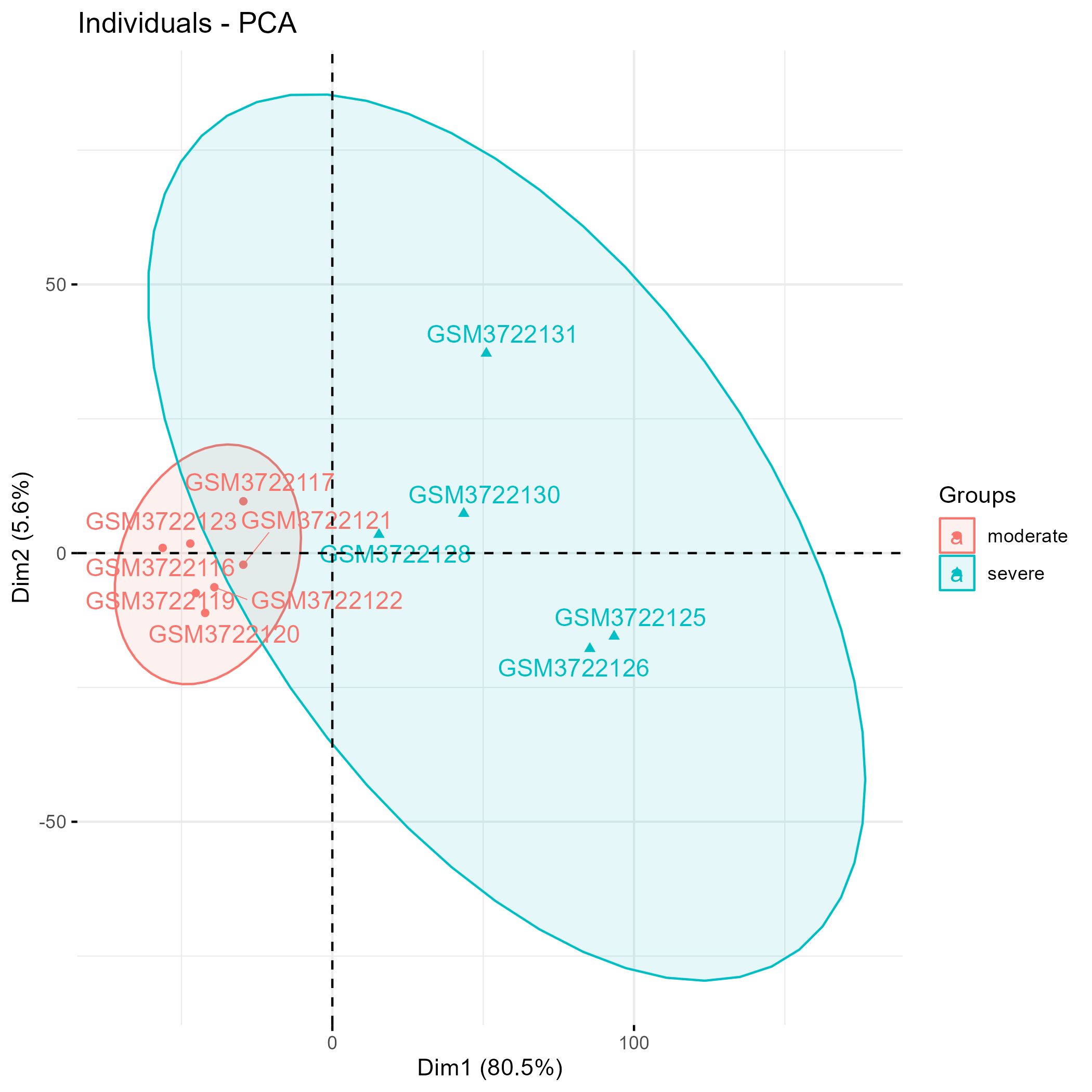
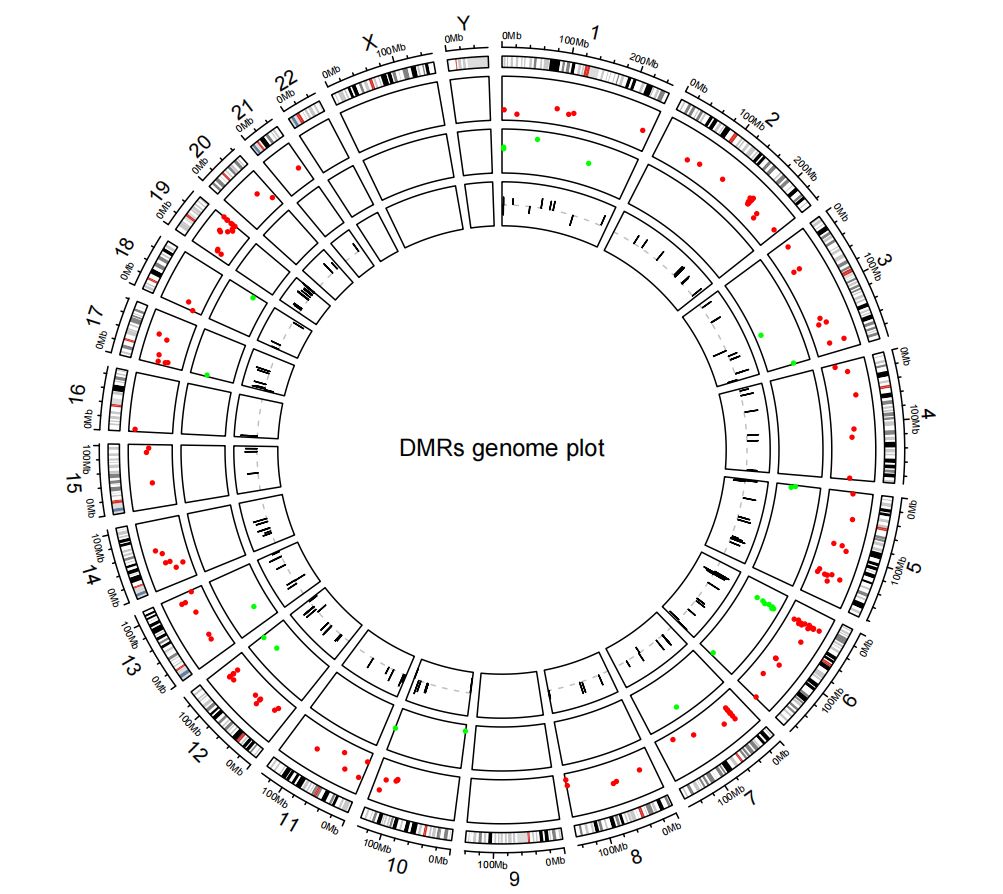
DMRs差异甲基化区域的基因组circos图
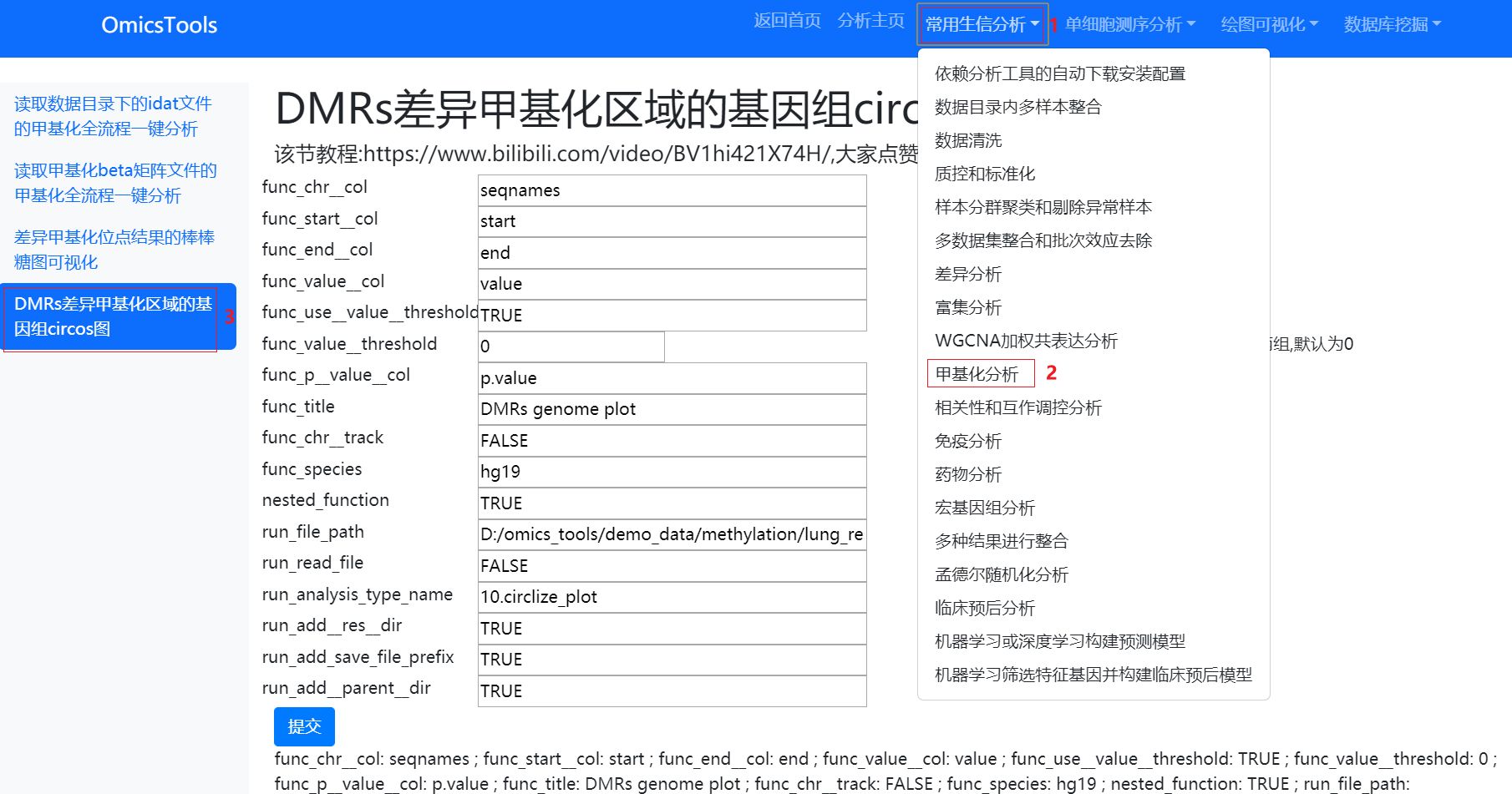
参数解释
func_chr__col: bed文件中染色体编号所在的列名
func_start__col: bed文件中起始位置所在的列名
func_end__col: bed文件中终止位置所在的列名
func_value__col: bed文件中结果数值所在的列名
func_use__value__threshold: 是否对value的阈值进行筛选,默认为TRUE
func_value__threshold: bed文件中结果数值的阈值,默认为0
func_p__value__col: bed文件中p值所在的列名
func_title:图表的标题
func_chr__track:是否绘制不同颜色的染色体轨道
func_species:物种的基因组版本号
nested_function:是否嵌套函数
run_file_path:甲基化DMRs结果的文件路径
run_read_file:是否要读取文件
run_analysis_type_name:分析项目名称
run_add__res__dir:是否要创建res_dir结果目录
run_add_save_file_prefix:是否要添加结果保存文件的前缀
run_add__parent__dir:是否在上一级目录下创建目录或保存结果
提交
已给定参数的默认值
func_chr__col: seqnames ;
func_start__col: start ;
func_end__col: end ;
func_value__col: value ;
func_use__value__threshold: TRUE ;
func_value__threshold: 0 ;
func_p__value__col: p.value ;
func_title: DMRs genome plot ;
func_chr__track: FALSE ;
func_species: hg19 ;
nested_function: TRUE ;
run_file_path: D:/omics_tools/demo_data/methylation/lung_results/myDMR_BumphunterDMR.csv ; run_read_file: FALSE ;
run_analysis_type_name: 10.circlize_plot ;
run_add__res__dir: TRUE ;
run_add_save_file_prefix: TRUE ;
run_add__parent__dir: TRUE
运行中的显示信息
分析正在执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data/methylation\10.circlize_plot; 运行结果日志保存的路径为: D:/omics_tools/demo_data/methylation\10.circlize_plot\10.circlize_plot_last_final_run_res_log.csv
窗口截图
运行完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data/methylation\10.circlize_plot; 分析结果日志保存的路径为: D:/omics_tools/demo_data/methylation\10.circlize_plot\10.circlize_plot_last_final_run_res_log.csv
结果展示
结果文件列表

Circos图
差异甲基化位点结果的棒棒糖图可视化
参数解释
func_data__type:数据类型选择methylation
func_sequence__type: 序列类型,是DNA还是protein
func_gtf__anno__file: hg19的基因组注释gtf文件,450K和EPIC都是用的hg19基因组
func_gene__list__str:选择绘制的基因,多个基因用分号分割来批量绘制
func_gene__col: DMP结果文件中基因名称的列名
func_pos__col: DMP甲基化位点位置所在的列名
func_pos__name__col: 甲基化探针名称所在的列名
func_value__col: DMP结果的组间甲基化差值所在的列名
func_pval__col: DMP结果的p值所在的列名
nested_function: 是否嵌套函数
run_file_path: 甲基化DMP结果的文件路径
run_read_file: 是否要读取文件
run_analysis_type_name: 分析项目名称
run_add__res__dir: 是否要创建res_dir结果目录
run_add_save_file_prefix: 是否要添加结果保存文件的前缀
run_add__parent__dir:是否在上一级目录下创建目录或保存结果
提交
已给定的参数默认值
func_data__type: methylation ;
func_sequence__type: DNA ;
func_gtf__anno__file: E:/data/download/Homo_sapiens.GRCh37.75.genome_anno.csv ; func_gene__list__str: HOXB3;CLDN18 ;
func_gene__col: gene ;
func_pos__col: MAPINFO ;
func_pos__name__col: V1 ;
func_value__col: Diff.Value ;
func_pval__col: P.Value ;
nested_function: TRUE ;
run_file_path: D:/omics_tools/demo_data/methylation/lung_results/myDMP_C_to_T_with_gene_anno.csv ; run_read_file: FALSE ;
run_analysis_type_name: 9.met_lollipot_plot ;
run_add__res__dir: TRUE ;
run_add_save_file_prefix: TRUE ;
run_add__parent__dir: TRUE
窗口截图
运行中的显示信息
执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data/methylation\9.met_lollipot_plot; 分析结果日志保存的路径为: D:/omics_tools/demo_data/methylation\9.met_lollipot_plot\9.met_lollipot_plot_last_final_run_res_log.csv
运行完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data/methylation\9.met_lollipot_plot; 分析结果日志保存的路径为: D:/omics_tools/demo_data/methylation\9.met_lollipot_plot\9.met_lollipot_plot_last_final_run_res_log.csv
结果展示
结果文件列表
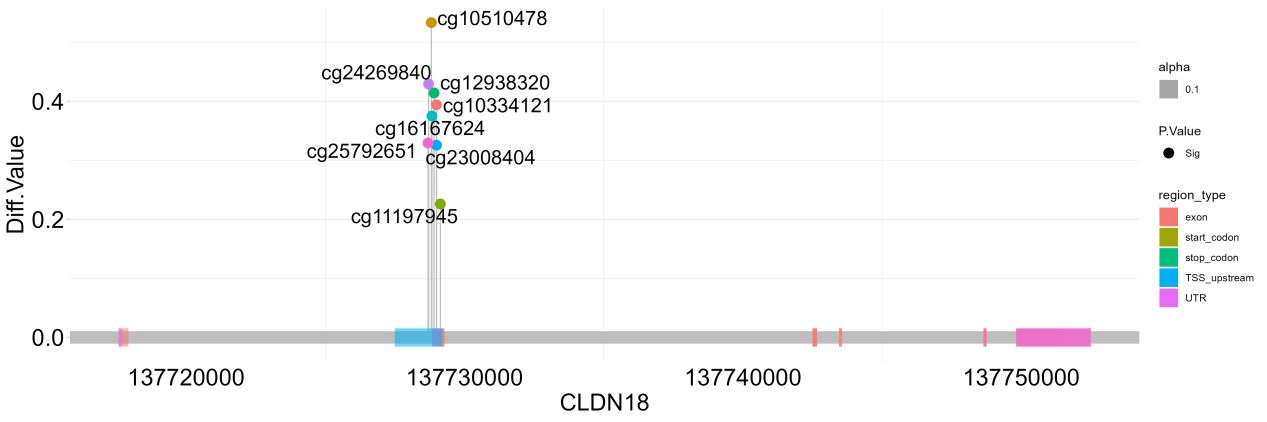
结果图

差异甲基化DMPs的统计分析条形图可视化



差异甲基化基因的火山图绘制
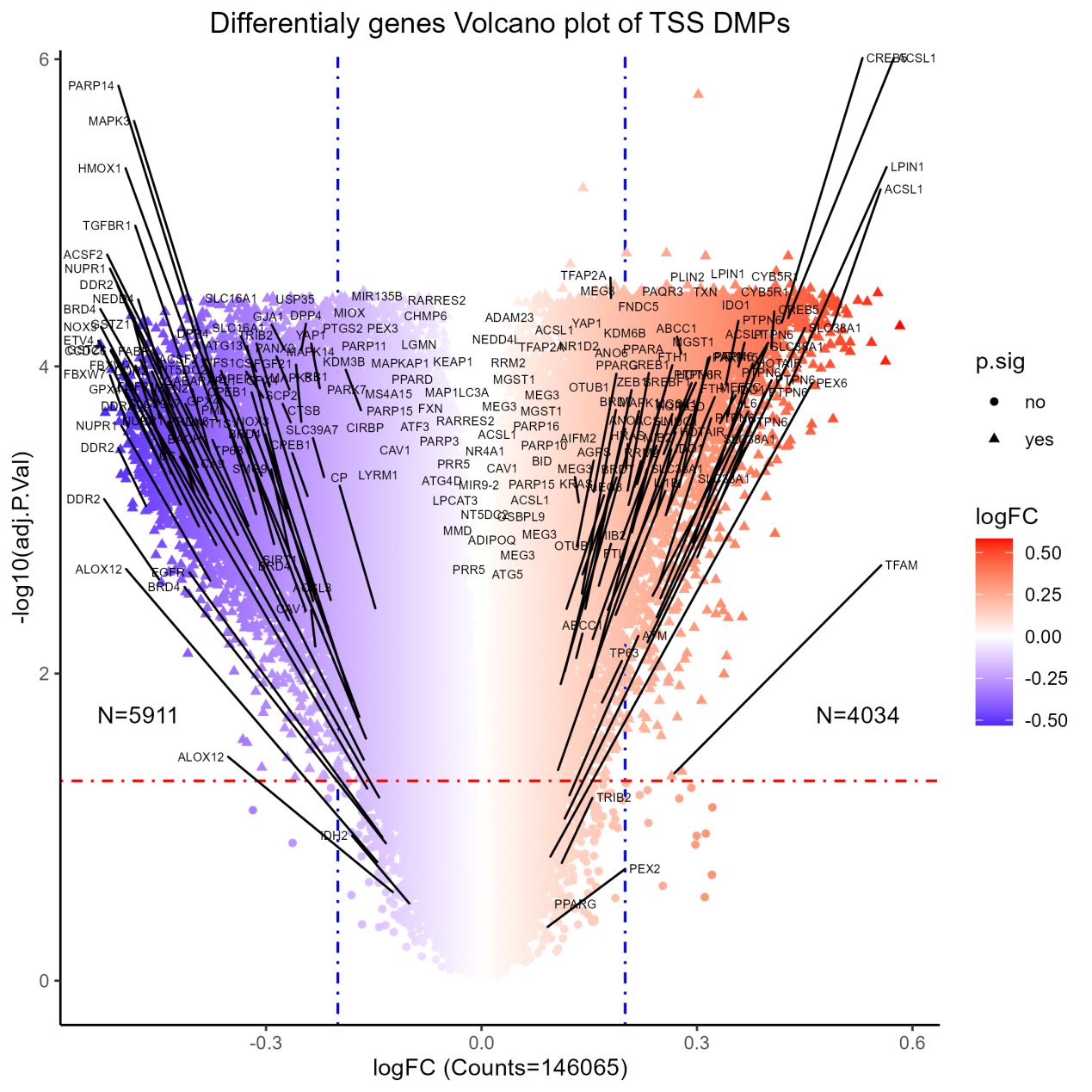
差异甲基化基因的GO,KEGG和GSEA富集分析
差异甲基化基因的GO和KEGG富集分析
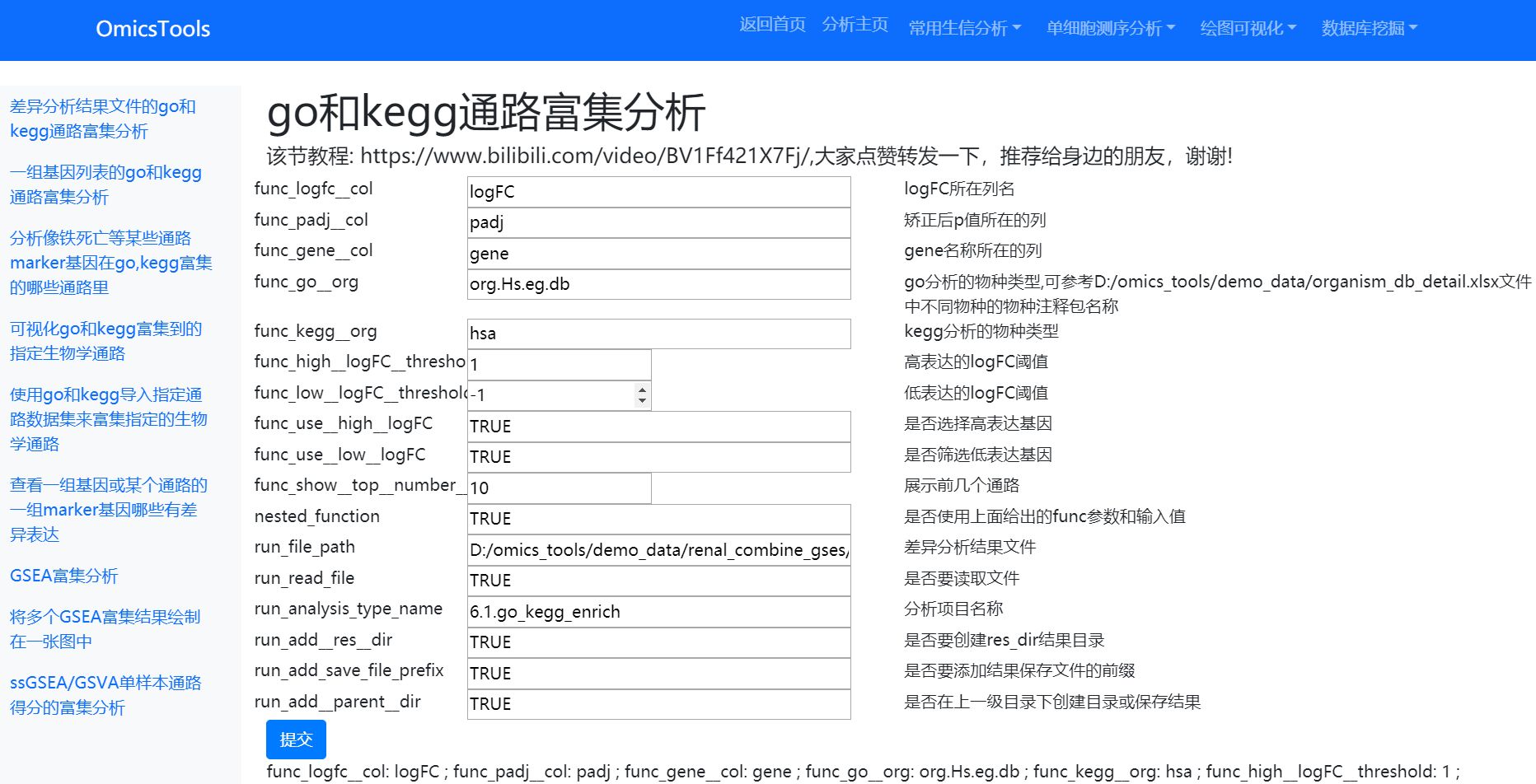
GO富集分析结果图
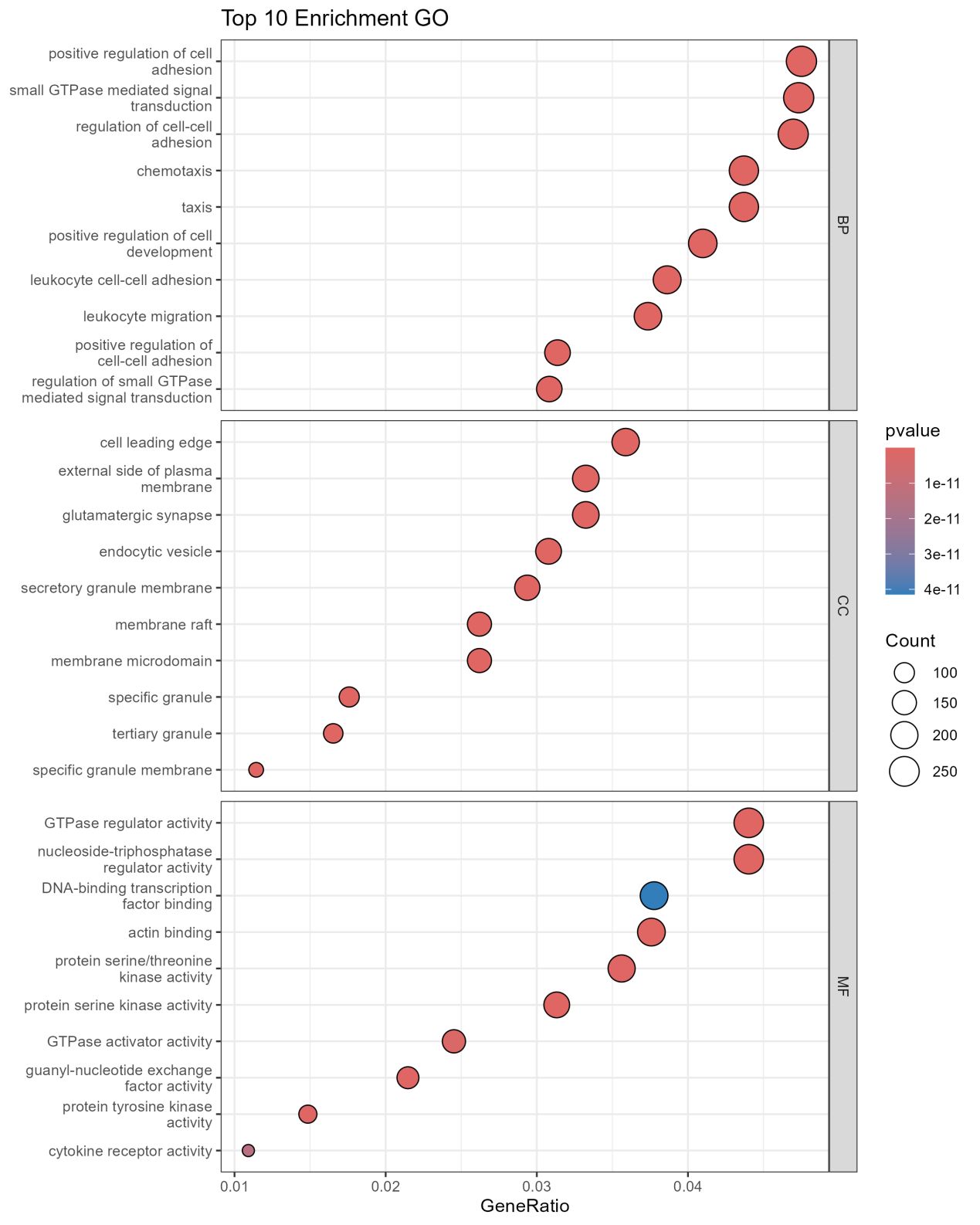
KEGG富集分析结果图
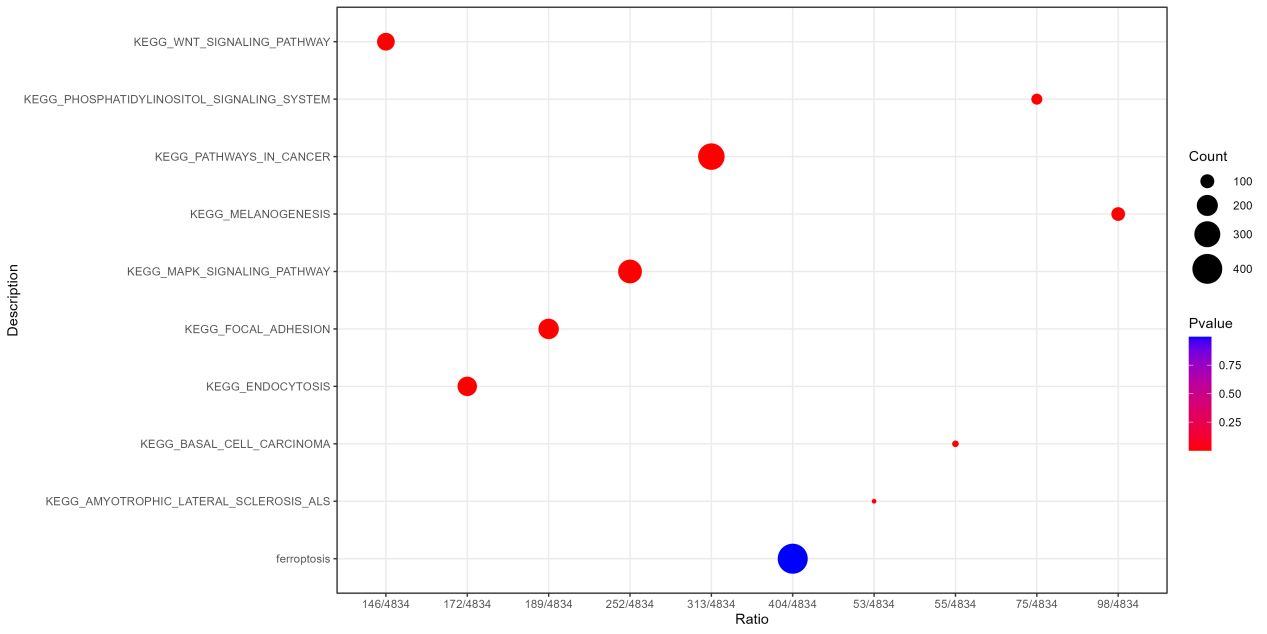
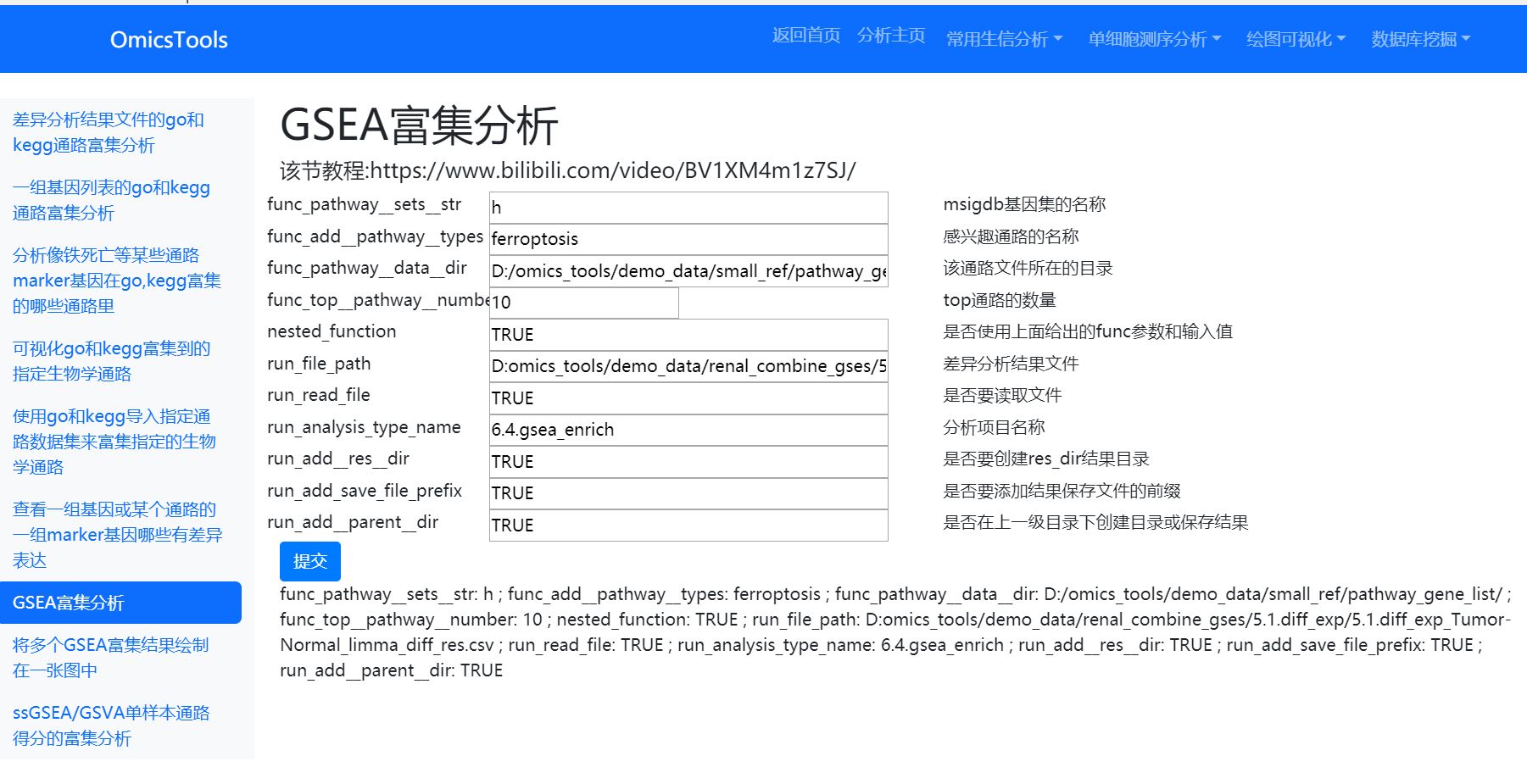
GSEA富集分析
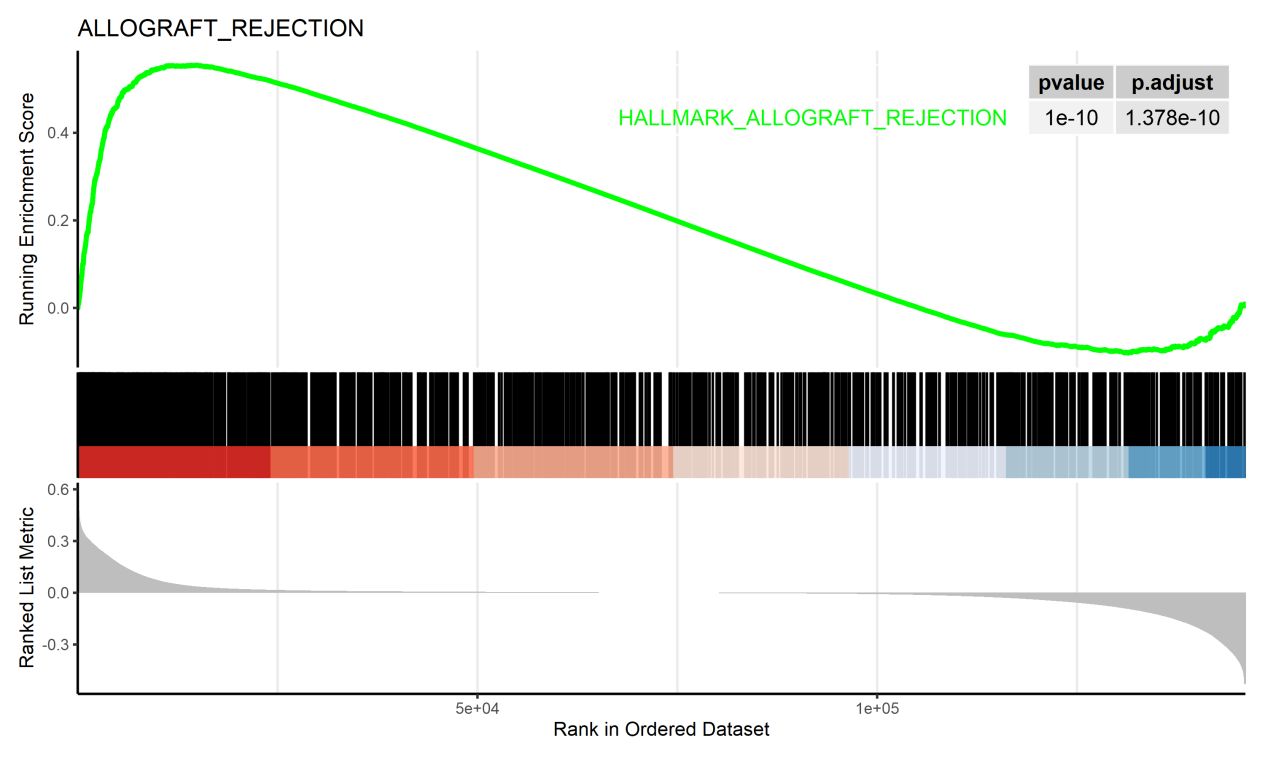


























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










