Cellranger-单细胞测序数据表达定量
Cellranger-单细胞测序数据表达定量
1、Cellranger安装
2、参考基因组下载
3、表达定量
1、Cellranger软件安装
首先进去Cellranger官网进行简单注册,随后根据将其下载到自己的服务器中 Cellranger官网
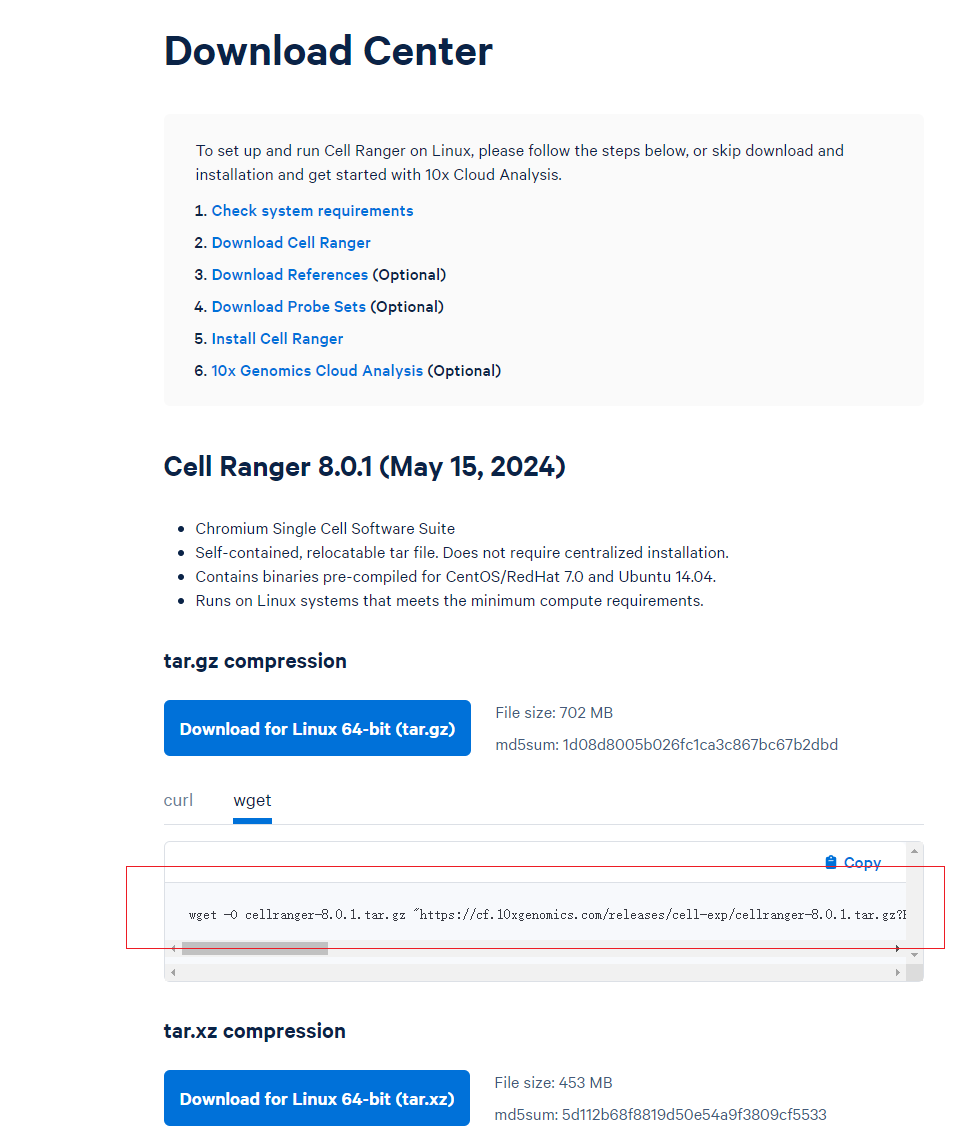
conda activate base
wget -O cellranger-8.0.1.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-8.0.1.tar.gz?Expires=1729879664&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA&Signature=YVKVK113B0AImruKKjdFbTZcUKTGJOY6T-r5j41L9sSxdDoWDQCFoXr-0MdRXoCx5d0ud61bLzZ-vg7vpQXkMYk~Z0CEQqCpFhhkoEeBvzF4RVdyBr2aBosaMAq95firdEhAlzxD-~y0mpXsjO7DjFavsZTUOMPjvu5V9-eaT8ItbnDR06ZkuPi1RoKm3Wv2nrDmz4CJUIUs2vn7YPlZ9-beN6UidysA7WHT86ofcxJX0aPGUvi4t6yLaXaK8ZeTpSvii-wEtaiaXDXBOuo6plhy6hMzAZMS6fYv72oFXHSwPUT8WW6bZaBf8OLkKj6W3hQO5QE5xb1QnbcEXT3d~Q__"
# 官网也提供curl下载
# 检查md5sum值是否完整
md5sum cellranger-8.0.1.tar.gz
# 解压并安装
tar -zxvf cellranger-8.0.1.tar.gz
# 将cellranger添加到环境变量
vim ~/.bashrc
export PATH=/yourpath/cellranger-8.0.1:$PATH # yourpath为你的软件路径
source ~/.bashrc

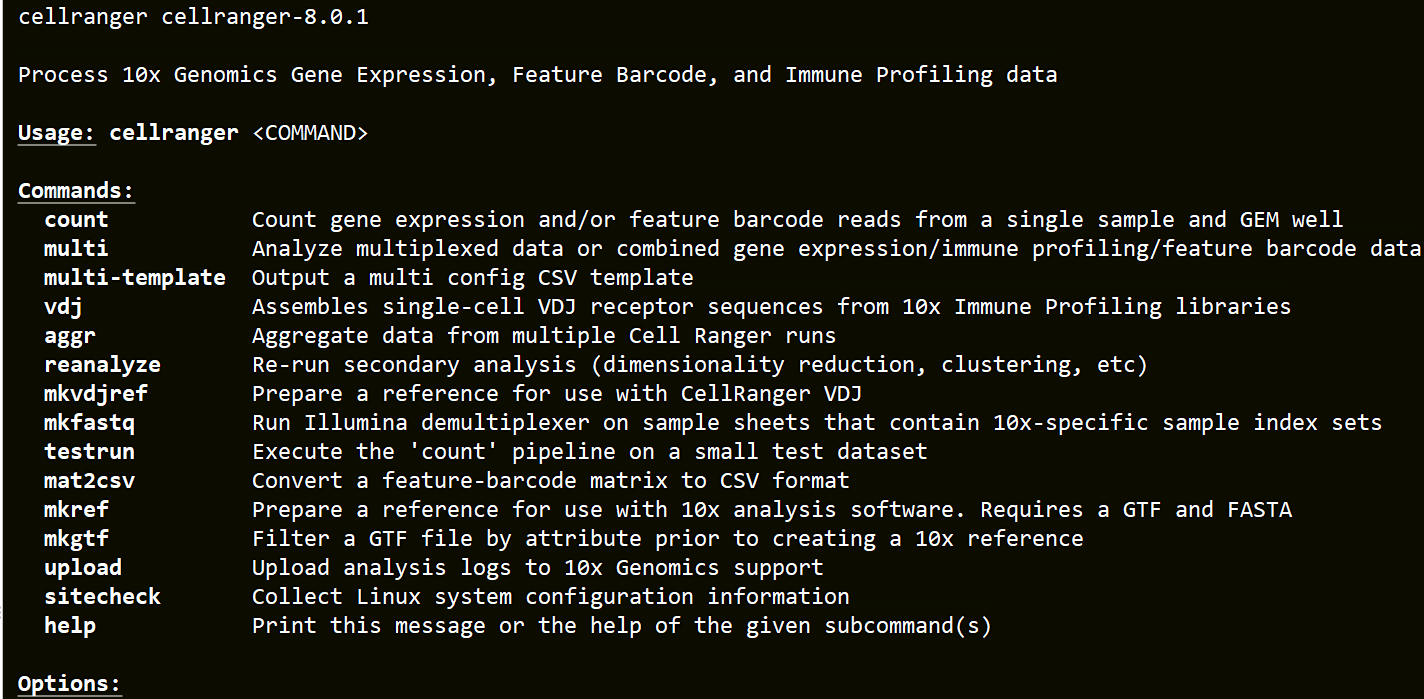
在终端当中输入cellranger命令检测是否安装成功,出现以下内容即为安装成功
2、下载参考序列
官方提供了几个参考序列,包括人类,小鼠,大鼠和V(D)J参考序列
# Human reference (GRCh38) - 2024-A
wget "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-2024-A.tar.gz"
# Mouse reference (GRCm39) - 2024-A
wget "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCm39-2024-A.tar.gz"
# Rat reference (mRatBN7.2) - 2024-A
wget "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-mRatBN7-2-2024-A.tar.gz"
# Human V(D)J reference (GRCh38)
wget "https://cf.10xgenomics.com/supp/cell-vdj/refdata-cellranger-vdj-GRCh38-alts-ensembl-7.1.0.tar.gz"
# 解压缩
tar -zxvf refdata-gex-GRCh38-2024-A.tar.gz
根据自己的研究需求进行下载
3、原始测序数据下载
可以选择GEO或者NGDC的GSA for Human下载原始数据,此处以GSA为例HRA003293,数据需要申请,可以选择open开放数据
wget -r -nH --cut-dirs=2 --ftp-user=name --ftp-password=password ftp://download.big.ac.cn/gsa-human/HRA003293/ 




 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章


















 7791
7791

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








