单细胞转录组 —— 原始数据处理实战(Cell Ranger)
前言
我们前面介绍了单细胞转录组原始数据的处理步骤,以及每一步中涉及的各种问题及解决方案。下面我们将介绍几个常用的原始数据的处理流程。
参考基因组
首先是参考基因组的选择,大多数 scRNA-seq 实验都是利用人体或小鼠组织、器官组织或细胞培养物进行的。
这些基因组组装和注释的更新比较频繁。有两种常用的组装文件来源:
UCSC的组装文件名为hg19、hg38、mm10等GRC:如GRCh37、GRCh38、GRCm38等
UCSC 和 GRC 的主要版本在主要染色体上是一致的(如 hg38 中的 chr1 等于 GRCh38 中的 chr1),但在附加等位基因和所谓的 ALT 位点上有所不同,这些位点在次要版本(如 GRCh38.p13)之间会发生变化。
更多信息请查阅 Genome Reference Consortium 和 Heng Li’s blog。
基因组注释文件定义了基因组(基因)的转录区,以及注释具有外显子内含子边界的确切转录本,并为新定义的特征分配类型–如蛋白质编码、非编码等。
人类和小鼠基因组注释的常用来源是 RefSeq、ENSEMBL 和 GENCODE。
其中 RefSeq 是这三个来源中最保守的一个,每个基因注释的转录本往往最少。RefSeq 转录本 ID 以 NM_ 或 NR 开头,如 NM_12345。
ENSEMBL 和 GENCODE 非常相似,可以互换使用。其中基因名称以 ENSG(人类)和 ENSMUSG(小鼠)开头;转录本分别以 ENST 和 ENSMUST 开头。
除了基因 ID,大多数基因还有一个常用的名称(gene symbol);例如,人肌动蛋白 B 的 ID 为 ENSG00000075624,symbol 为 ACTB。
人类基因名称由 HGNC 定期更新和定义。老鼠的基因名称是由一个类似的组织 MGI 决定的。
目前的 ENSEMBL/GENCODE 数据库中人类基因组注释包含约 6 万个基因,其中 2 万个为蛋白质编码基因,以及 23.7 万个转录本。
大多数基因可按类型粗略地分为蛋白质编码基因、长非编码 RNA、短非编码 RNA 和假基因。
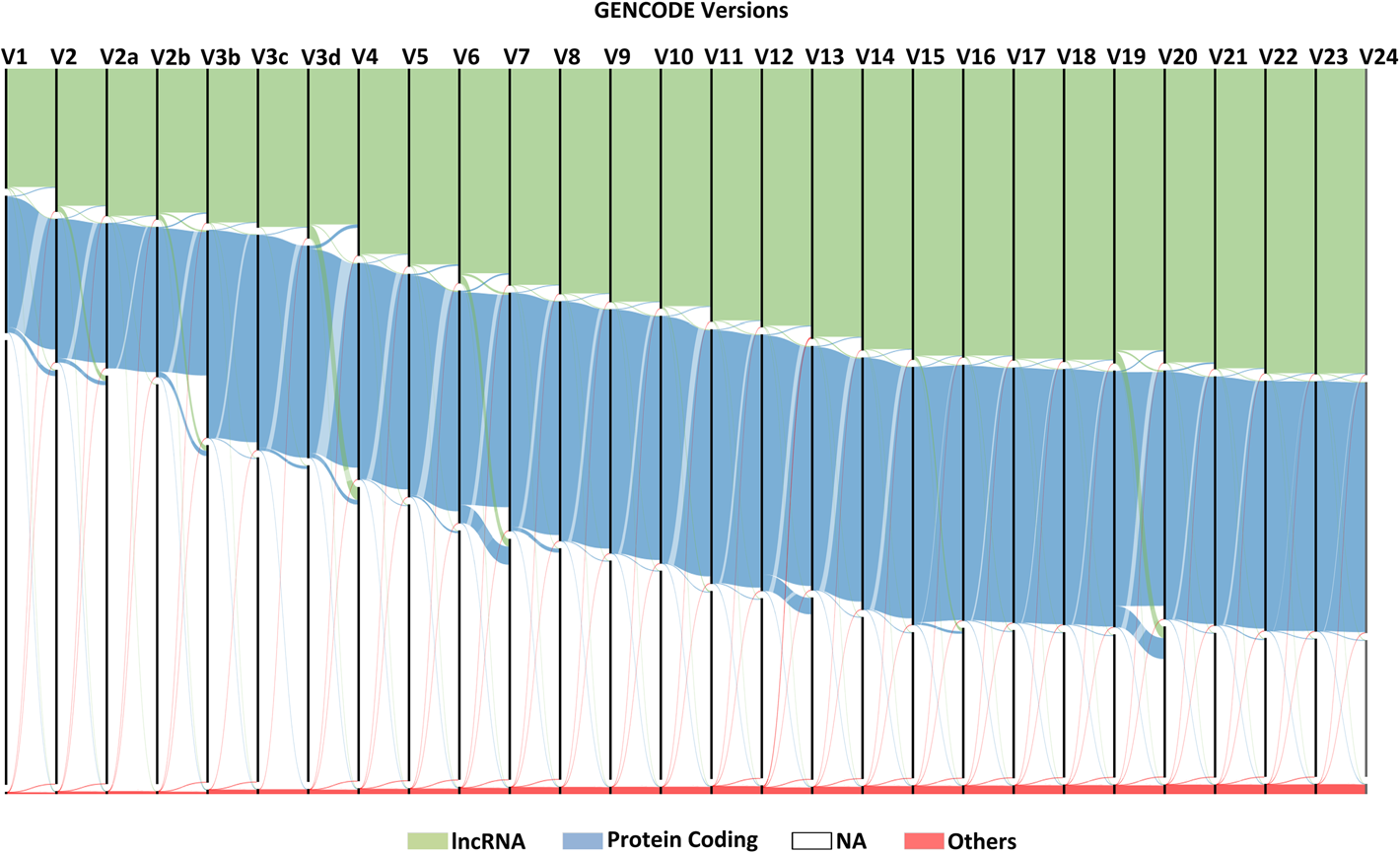
如图所示,不同注释版本之间基因也不一样
数据处理流程
scRNA-seq 原始数据处理流程包括四个主要步骤:
- 将
cDNA片段映射到参考文献 - 将
reads分配给基因 - 将
reads分配到细胞(根据barcode) - 计算
RNA分子的数量(无重复UMI数量)
处理的结果会产生一个基因与细胞的计数矩阵,用来估算每个细胞中每个基因的 RNA 分子数量。
Cell Ranger
Cell Ranger 是处理 10x Genomics Chromium scRNAseq 数据的默认工具。它使用 STAR 软件将 reads 与基因组进行剪接比对。
它根据 reads 在基因组中的比对位置,将其分为外显子、内含子和基因间三类。即如果 reads 至少有 50% 的位置与外显子相交,则分为外显子类;如果为非外显子且与内含子相交,则分为内含子类;否则为基因间(如下图所示)。
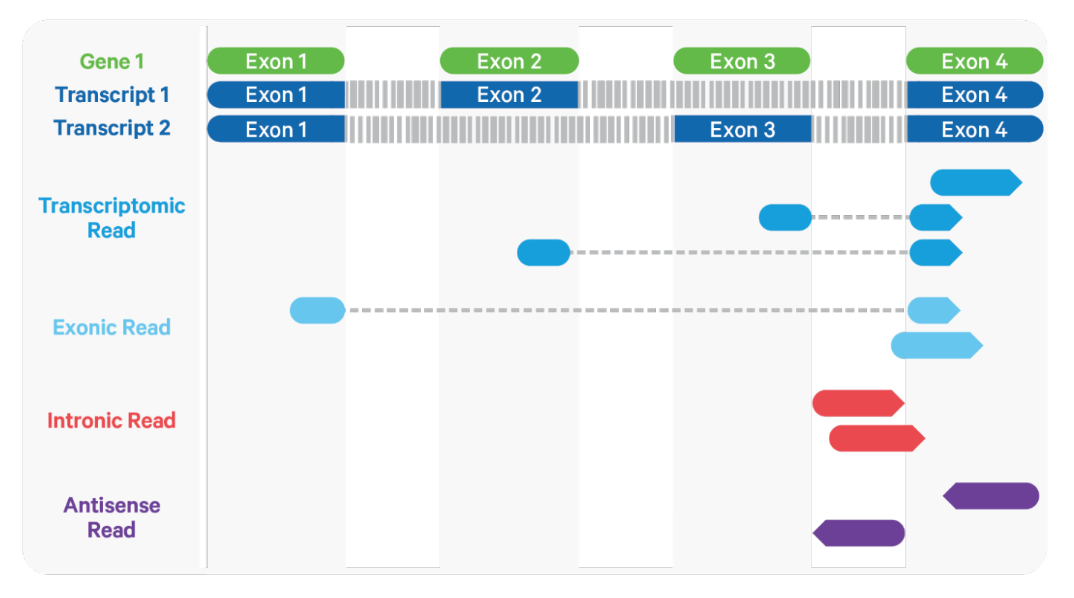
在对 reads 进行类型分配后,会调整比对质量。即对于与一个外显子位点对齐但也与一个或多个非外显子位点对齐的 reads,外显子位点会被优先考虑,reads 会被认为有把握比对到外显子位点,并被赋予最高比对质量。








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章


















 7345
7345

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










