最近想使用文献中的数据集,发现每篇文献提供的数据格式都不太一样,创建seurat对象会遇到一些问题。
以标准的输入文件为例,命名为barcodes.tsv.gz、features.tsv.gz、matrix.mtx.gz,分别是细胞标签,基因,表达矩阵,但是文章提供的数据可能并不是标准格式,需要打开文件确认。
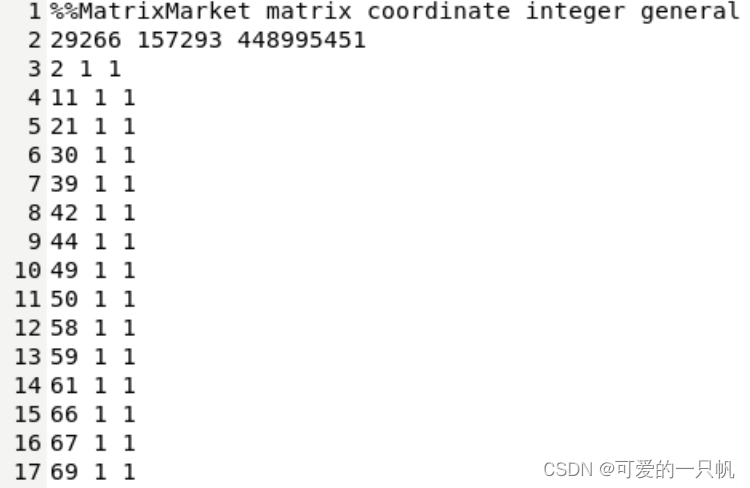
以SCP1852数据为例,发现有四个文件:

AllNuclei_snRNA_counts.mtx.gz打开是表达矩阵(即matrix.mtx.gz),三列分别为基因、细胞、表达量。
 AllNuclei_snRNA_counts_colnames.txt.gz打开是细胞信息(即barcodes.tsv.gz)
AllNuclei_snRNA_counts_colnames.txt.gz打开是细胞信息(即barcodes.tsv.gz)

AllNuclei_snRNA_counts_rownames.txt.gz打开是基因信息(即features.tsv.gz),标准的features文件是两列,包括ensemble ID和symbol,这里只有一列,最简单的方法就是复制一下,变成两列,不然后面用Read10X读取文件会显示报错error in [.data.frame(category.matrix, , gene.group, drop = f) : undefined columns selected。





















 2994
2994











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








