WGCNA使用非常广泛,网上流传的示例代码也非常多,但还是推荐大家直接参考官方代码,最全面且权威:https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/Tutorials/index.html
今天不讲代码,只讲讲这些图怎么看,能表达什么意义。
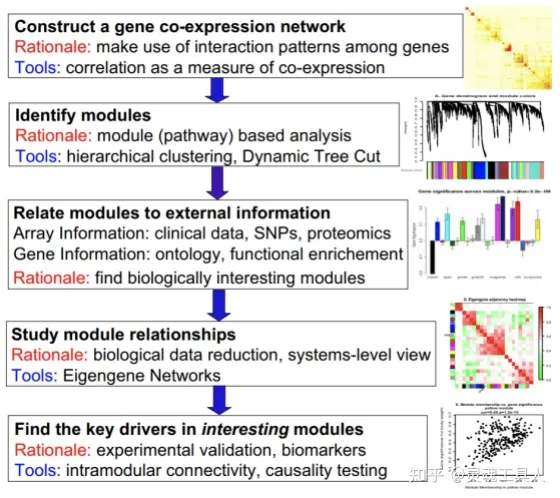
WGCNA文章[1]给出了工作流程图,主要包括:构建共表达网络→模块识别→模块与性状关联→模块相关性→关键模块中识别核心基因
在构建共表达网络之前,还有一步比较关键的步骤——寻找最优软阈值(soft thresholding或power),使构建的网络更符合无标度拓扑结构。这时,我们会看到如下图:

确定最优软阈值主要参考左图,即在不同软阈值(x轴)情况下的无标度拟合指数(scale-free fit index,y轴)。其中红线表示主观选择的无标度拟合指数取值,如图为0.9,一般我们选择0.8~0.95之间较为稳妥。从左图看,当无标度拟合指数在0.9时,符合构建无标度网络的最小软阈值为6,因此可以选择6作为最优软阈值用于后续分析。右图为不同软阈值情况下的网络连通度。
基于最优软阈值构建共表达网络,将基因划分到不同模块后,可以绘制基因聚类树:
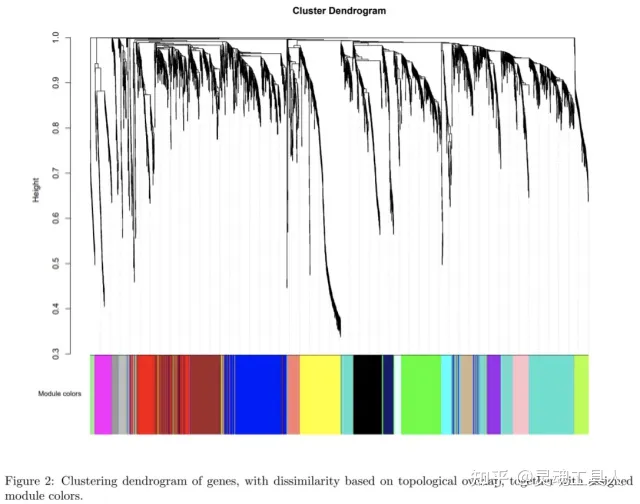
这个图可以分为两部分看:上半部分是基因的层次聚类树状图,下半部分是基因模块,也就是网络模块。上下对应,可以看到距离较近的基因(聚类到同一条分支)被划分到了同一模块。
接着,计算模块与感兴趣的临床特征之间的相关性与显著性,绘制相关性热图:
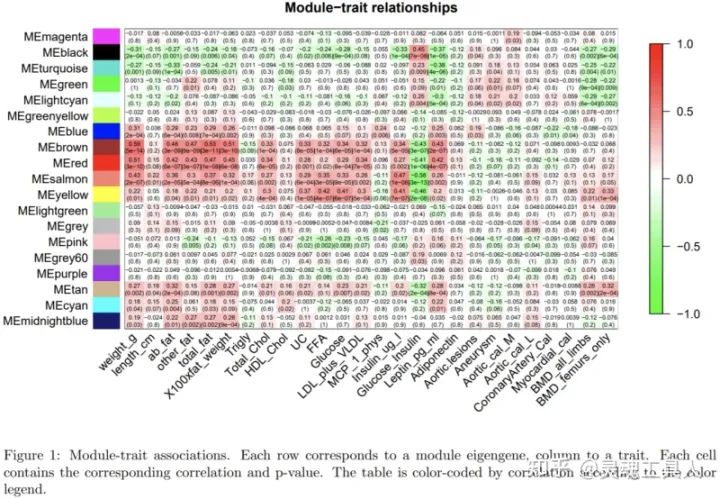
上图中,最左侧的颜色块代表模块,最右侧的颜色条代表相关性范围。中间部分的热图中,颜色越深相关性越高,红色表示正相关,绿色表示负相关;每个单元格中的数字表示相关性和显著性。如上图,brown模块与weight性状表现为正相关且相关性最高。此时,我们可以选择相关性最高的brown模块作为关键模块。一般,我们会按相关性的绝对值筛选最相关模块,即负相关模块也应该考虑在内。需要注意的是,grey模块中包含了所有未参与聚类的基因,因此是无效模块,不应用于后续分析。
基于拓扑重叠矩阵,可以绘制基因之间的相关性热图。其中颜色越深,说明基因之间的相互作用越强。由于对角线表示模块内部基因之间的相互作用,所以自然是对角线上的颜色最深:

也可以看看模块之间的聚类树和相关性热图,探索模块之间的互作关系:

可以将感兴趣的性状,如weight也加入到图中。如上图,可以发现red、brown、blue模块之间有很强的相关性,甚至比他们与weight之间的相关性更强。
之后针对关键模块brown和感兴趣的性状weight进一步挖掘,看看基因与模块的相关性(Module Membership, MM)和基因与性状的相关性(Gene Significance, GS)之间是否有某种关联。 `

通过以上散点图,可以发现MM和GS呈正相关,说明这些与性状高度相关的基因,在关键模块中也扮演着举足轻重的角色。
我们也可以基于关键模块中基因之间的相似性,构建关键模块网络,以Cytoscape或VisANT软件可视化展示网络中基因之间的互作关系:

最后,也是最重要的——基于关键模块筛选核心基因。这一步的可用方法比较多,没有固定限制。比如,可以根据MM>0.8且GS>0.3筛选核心基因[2];可以根据关键模块的网络degree筛选top 30作为核心基因[3];也可以计算kME值,依据|kME|≥0.7筛选[4];或者将网络导入Cytoscape后,利用插件cytoHubba筛选[5]。
1. Langfelder P, Horvath S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 2008;9:559. Published 2008 Dec 29. doi:10.1186/1471-2105-9-559
2. Song ZY, Chao F, Zhuo Z, Ma Z, Li W, Chen G. Identification of hub genes in prostate cancer using robust rank aggregation and weighted gene co-expression network analysis. Aging (Albany NY). 2019;11(13):4736-4756. doi:10.18632/aging.102087
3. Zhang X, Feng H, Li Z, et al. Application of weighted gene co-expression network analysis to identify key modules and hub genes in oral squamous cell carcinoma tumorigenesis. Onco Targets Ther. 2018;11:6001-6021. Published 2018 Sep 19. doi:10.2147/OTT.S171791
4. Panahi B, Hejazi MA. Weighted gene co-expression network analysis of the salt-responsive transcriptomes reveals novel hub genes in green halophytic microalgae Dunaliella salina. Sci Rep. 2021;11(1):1607. Published 2021 Jan 15. doi:10.1038/s41598-020-80945-3
5. Li CY, Cai JH, Tsai JJP, Wang CCN. Identification of Hub Genes Associated With Development of Head and Neck Squamous Cell Carcinoma by Integrated Bioinformatics Analysis. Front Oncol. 2020;10:681. Published 2020 May 22. doi:10.3389/fonc.2020.00681








 本文介绍WGCNA工作流程,包括确定最优软阈值、构建共表达网络、基因聚类及模块识别等步骤,并解析模块与性状关联分析、关键模块核心基因筛选等内容。
本文介绍WGCNA工作流程,包括确定最优软阈值、构建共表达网络、基因聚类及模块识别等步骤,并解析模块与性状关联分析、关键模块核心基因筛选等内容。


















 4464
4464

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










