桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.这期分享一篇 2022年8月发表于 Signal Transduct Target Ther (IF 39.3)的文章,作者基于单细胞转录组测序揭示肺腺癌和鳞癌之间的异质性免疫图谱。该文章使用桓峰基因公众号里面生信分享教程即可实现,有需要类似思路的老师可以联系我们!
摘 要
对非小细胞肺癌(NSCLC)患者免疫治疗策略的选择和临床反应的预测至关重要。单细胞转录组测序(scRNA-seq)技术为解剖肺腺癌(LUAD)和肺鳞状细胞癌(LUSC)这两种主要非小细胞肺癌亚型之间的不同免疫特征提供了机会。在这里,我们对来自19例NSCLC患者的40个肿瘤样本和匹配的邻近正常组织的72475个免疫细胞进行了scRNA-seq,并绘制了系统的免疫细胞转录组图谱。基于癌症基因组图谱(TCGA)的不同细胞组成、差异表达基因(DEGs)、细胞-细胞相互作用、伪时间轨迹、转录组因子和预后因素的联合分析,揭示了细胞毒性和效应T细胞和NK细胞以及不同功能巨噬细胞(Mφ)亚型在LUAD和LUSC之间免疫微环境异质性中的核心作用。在LUAD和LUSC中,Mφ的优势亚型分别为FABP4-Mφ和SPP1-Mφ。重要的是,我们发现了一个新的淋巴细胞相关的Mφ簇,我们将其命名为SELENOP-Mφ,并进一步确定了其在两种类型中的抗肿瘤作用,特别是在LUAD中。我们对免疫异质性的全面描述和Mφ簇的定义有助于在临床实践中为肺癌患者设计个性化的治疗方案。

生信分析流程
相关数据集选择:
19位癌和癌旁配对患者(10 LUAD and 9 LUSC)Single-cell RNA sequencing
1014 NSCLC patients (513 LUAD and 501 LUSC) from the TCGA database
基因集选择:
筛选出的差异基因
生信分析方法:
根据文章的分析流程提取所有的分析内容,整理出来就13个分析条目,每个条目都包括分析的内容,这些分析构成了整个文章,本文属于机器学习的临床预测分析类文章,下面我们就看看哪些分析可以利用桓峰基因公众号的教程来实现,点击分析条码就会跳转到对应公众号的教程,跟着教程做,您也能发轻松发高分,如下:
说明转录因子与靶基因之间的调控网络 (Cytoscape);
研究结果
1. 肿瘤和相应正常组织中LUAD和LUSC样本的高分辨率免疫细胞型图
a. 一个原理图,突出了研究设计和分析的工作流程。
b. t-SNE图显示所有免疫细胞的鉴定细胞类型。
c. 72,475个单个免疫细胞的t-SNE图,用疾病亚型着色。
d. 热图显示细胞类型特异性标记基因在不同免疫细胞中的表达。
e. 来自每个病人的细胞类型的比例。

2. LUAD和LUSC异质性中免疫细胞不同的基因表达模式
a. t-SNE图显示LUAD和LUSC的细胞类型分布。
b. 显示LUAD和LUSC各细胞类型特征基因表达的热图。
c. 根据LUAD和LUSC之间的DEGs,不同颜色的条形图显示了GSEA在每种细胞类型中富集的GO功能通路的差异。
d. 小提琴图比较LUAD和LUSC之间所有免疫细胞中MARCO、ALOXAP5、IGKC和SPP1的表达水平。
e. TCGA LUSC数据的总生存曲线。
f. LUAD和LUSC组织的多色免疫组化染色用于验证免疫细胞中癌症亚型特异性基因的表达。
g. 不同样品来源的所有免疫细胞中转录因子对基因表达调控的热图。
h. LUAD和LUSC中各免疫细胞不同基因间的TF基因调控网络。

3. LUAD和LUSC中不同免疫细胞类型之间的配体-受体相互作用
a. 和弦图展示了LUAD和LUSC不同免疫细胞类型之间的相互联系。
b. LUAD和LUSC中不同免疫细胞类型相互作用对的分子相互作用对。
c. 点图显示肿瘤亚型特异性配体和受体在每种细胞类型中的表达。颜色和大小表示效应大小。
d. LUAD和LUSC中特异性配体和受体的表达。气泡大小根据表达式表示LUAD/LUSC的比值。
e,f. 点图显示了IL-17、t细胞受体、PD-L1表达和PD-1检查点途径和血管生成所涉及的选定相互作用对的平均表达水平和百分比。

4. LUAD和LUSC中T和NK细胞的重新聚集和发育轨迹
a. 所有样本中15853个CD4+ T细胞的聚类。
b. 点图显示每个CD4亚群中前5个标记基因的表达。
c. 每种免疫细胞类型在LUAD、LUSC、NL(SC)和NL(AD)中的百分比。
d,e. monocle 推测CD4+ T细胞的发育轨迹,以细胞亚型和样本组着色。
f. 不同标记基因的代表性基因表达水平。
g. 所有样本8735个CD8+ T细胞聚类。
h. 各免疫细胞类型在LUAD、LUSC、NL(SC)和NL(AD)中的百分比。
i. monocle推测CD8+ T细胞的发育轨迹,伪时间染色,样本组细胞亚型划分。
j. 不同标记基因的代表性基因表达水平。
k. 所有样本中20,584个NK细胞的聚类。
l. LUAD、LUSC、NL(SC)和NL(AD)各NK亚型的平均比例。

5. Mφ在LUAD和LUSC中的重聚和基因表达模式
a. 所有样本的聚类为11016 Mφs。
b. LUAD(上)和LUSC(下)中Mφ亚型的t-SNE。
c. 各Mφ亚簇中特定标记基因的小提琴图。
d. 方框图显示FABP4、FCN1、SPP1和SELENOP在LUAD和LUSC中的差异表达。
e. 基于M1、M2、血管生成和吞噬作用标记基因,ssGSEA对各Mφ亚型进行功能活性评分的差异。
f. 各Mφ亚型特异性差异表达基因热图及其GO注释。
g. LUAD和LUSC之间不同Mφ亚型肿瘤相关分子的热图。
h. 多色免疫组化染色,用于验证LUSC与LUAD在Mφ中抗体转录物的高表达。

6. Mφ在LUAD和LUSC中的轨迹分析显示出明显的特征
a,b. 所有肺样本中Mφ的分化轨迹,每个点用伪时间(a)和Mφ亚型(b)(下)标记,分为两个分支。
c,d. 伪热图显示了分枝1中Mφ分化过程中的各种基因,聚类为5个簇,散点分布图显示了伪时间内各状态下一些特定基因的表达变化。
e,f. 伪热图显示了分支2中参与Mφ分化过程的各种基因,聚为5个簇,散点分布图显示了伪时间内各状态下一些特定基因的表达变化。
g,h. 分支1 (g)和分支2 (h)中TFs及其靶基因的配体-受体连接网络

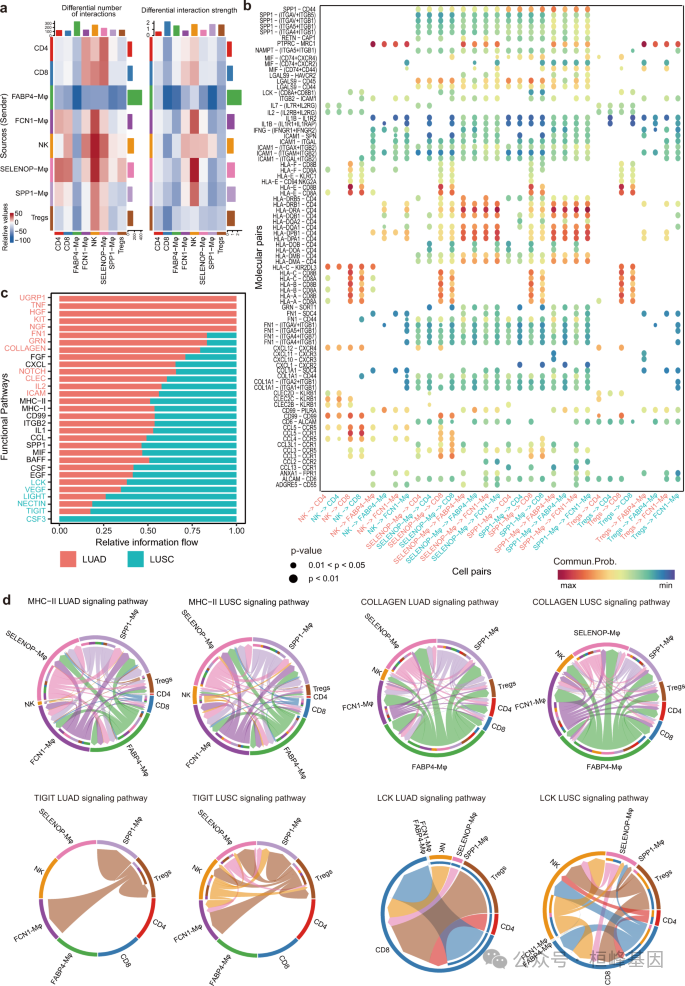
7. LUAD和LUSC中Mφ亚簇与淋巴细胞之间的差异通讯模式
a. 热图显示了LUAD和LUSC中Mφ亚型与淋巴细胞相互作用的不同数量和强度。
b. 选择了参与LUAD和LUSC细胞类型之间信号传递的重要配体-受体对。
c. 根据LUAD和LUSC之间推断网络中整体信息流的差异对选定的重要信号通路进行排名。
d. 和弦图显示LUAD和LUSC中MHC-II、Collagen、TIGIT和LCK信号的细胞间通讯网络。
Reference
Wang, C., Yu, Q., Song, T. et al. The heterogeneous immune landscape between lung adenocarcinoma and squamous carcinoma revealed by single-cell RNA sequencing. Sig Transduct Target Ther 7, 289 (2022). https://doi.org/10.1038/s41392-022-01130-8
单细胞生信分析教程
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【15】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (CellPhoneDB)
SCS【16】从肿瘤单细胞RNA-Seq数据中推断拷贝数变化 (inferCNV)
SCS【17】从单细胞转录组推断肿瘤的CNV和亚克隆 (copyKAT)
SCS【18】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (iTALK)
SCS【21】单细胞空间转录组可视化 (Seurat V5)
SCS【22】单细胞转录组之 RNA 速度估计 (Velocyto.R)
SCS【24】单细胞数据量化代谢的计算方法 (scMetabolism)
SCS【25】单细胞细胞间通信第一部分细胞通讯可视化(CellChat)
SCS【26】单细胞细胞间通信第二部分通信网络的系统分析(CellChat)
SCS【28】单细胞转录组加权基因共表达网络分析(hdWGCNA)
SCS【29】单细胞基因富集分析 (singleseqgset)
SCS【30】单细胞空间转录组学数据库(STOmics DB)
SCS【31】减少障碍,加速单细胞研究数据库(Single Cell PORTAL)
SCS【32】基于scRNA-seq数据中推断单细胞的eQTLs (eQTLsingle)
SCS【33】单细胞转录之全自动超快速的细胞类型鉴定 (ScType)
SCS【34】单细胞/T细胞/抗体免疫库数据分析(immunarch)
SCS【35】单细胞转录组之去除双细胞 (DoubletFinder)
SCS【36】单细胞转录组之k-近邻图差异丰度测试(miloR)
SCS【39】单细胞转录组之降维散点图的美化 (SCpubr)
SCS【40】单细胞转录组之便捷式细胞类型注释(scMayoMap)
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
























 18
18

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








