文章解读
文献来源:Prediction of pKa Using Machine Learning Methods with Rooted Topological Torsion Fingerprints: Application to Aliphatic Amines | Journal of Chemical Information and Modeling (acs.org)
代码复现参考文章:(文章所采用的是诺华内部PKa数据,无法复现)
1.分子相似性计算代码及其理论:
使用rdkiit进行分子指纹(Fingerprint)的类似度计算 - 知乎 (zhihu.com)
2.QSAR中训练集以及验证集划分的依据(time-split):
3.线性插值的原理以及matlab代码实现方法:
没想到会用到:线性插值(Linear Interpolation)原理及使用-CSDN博客
The Difference Between Extrapolation and Interpolation (thoughtco.com)
4.在深度学习网络中使用Grid Search对神经网络进行调参:
调参(2):使用网格搜索算法调试神经网络的参数(搬运) - 知乎 (zhihu.com)python/keras中用Grid Search对神经网络超参数进行调参_如何利用工具调整一个神经网络的超参数-CSDN博客
一、引言:
酸碱解离常数PKa是确定化合物电离状态的关键参数,在药代动力学(ADMET)中具有重要作用。在文章的研究方法中,作者通过尝试五种常见的机器学习方法:RF、PLS、XGBoost、lasso regression、SVM。通过药企诺华高通量测量的PKa值以及外源数据集,建立了脂肪族胺的PKa模型,这部分获取的数据集通过RMSE验证,模型显示出一致的良好预测统计量。结果表明,根拓扑扭转指纹结合ML方法为建立准确的PKa预测模型是一种很有前途的方法。
二、先前PKa预测方法以及预测方式:
1.对于分子式中非共轭结构原子的描述方式:
由于PKa高度依赖于电离原子周围的局部结构环境,先前存在QSPR方法(定量结构-性质关系的方法),通过原子描述符以宽度优先的模式描述PKa的结构特征,并在以可电离原子为中心的拓扑球体中传播。作者利用分子树状指纹(利用与根原子的相对距离来描述原子的空间结构)和PLS建立了PKa模型。
2.对于分子式中共轭结构原子的描述方式:
某些具有Π电子系统的化学基团不能通过“非共轭结构原子描述符“所明确描述,对于这部分Π电子共轭基团建模,需要不同的树描述符来构建预测模型。
3.先前QSPR的缺陷以及Jelfs等人对QSPR方法的改进:
先前大多数报道的QSPR的PKa模型都是用源自描述符来描述电离位点附近的化学环境,但并不能直接对路径和原子连通性进行编码(例如常见的Π-Π共轭结构)。为了解决这个问题,现存分子指纹与分子力场描述符补充提高准确性,一方面通过对不同的电子效应构建不同的树状指纹。(休克尔型连续共轭、连续共轭结构上的吸供电子效应)。
在文章发表前有很多人通过分子·指纹与分子力场描述符相互补充的方法来提高准确性,Jelfs等人进一步通过分子树状指纹与AM1的半经验描述符相结合,构建的PLS模型优于单一分子树状指纹描述符的模型。Milletti等人将分子相互作用能的计数作为分子描述符来构建用于PKa预测的PLS模型。(Milletti的分子相互作用能来源于AutoDock的Grid功能)。
三、作者提出的基于路径的原子描述符(RTorsion):
1.RTorsion与标准拓扑扭转指纹的差异:
① RTorsion是由拓扑扭转指纹衍生而来的根指纹,标准的拓扑扭转描述符由四个连续成键的非氢原子构成,而RTorsion描述符包括由根原子和主链以及支链的1到n个键组成的路径,实现了从每个距离级别上描述原子属性。
②RTorsion通过显式地对路径和原子连接进行编码,通过捕获氟原子以及三氟甲基基团的电子效应来弥补标准拓扑扭转指纹预测性能的不足。
2.RPair以及RMorgan分子描述的基本原理以及差异:
2.1RPair的定义以及表示方法:
RPair是一种基于原子对的原子描述符,原子对描述符的定义形式为(根原子1,与根原子相隔的化学键,原子类型(根原子的相对位置数字)),例如苄位的碳原子为(N1, 1, C2)。RPair与传统的源自对指纹的不同之处在于RPair只捕获来自根原子、距离根原子有n个键的原子对。在计算机硬盘中以idx的形式排列存储。
2.2RTorsion的定义以及表示方法:
RTorsion是一种基于路径的指纹,路径的定义形式为(根原子元素,非氢邻近的原子数-2,Π原子数量)基于构象元素(扭转角)的拓扑类似物与标准拓扑扭转指纹的不同之处在于它包括由1到n个键组成的路径,路径中的每个原子使用不同的原子类型(原子类型、Π电子对数量)来描述。例如4号位的碳原子(N,0,0)(C,0,0)(C,1,1)(C,1,1)。

2.3 RMorgan的定义以及表示方法:
RMorgan是一种圆形的原子描述符,通过计算根原子周围的基团来判别附近基团的原子元素、非氢邻位原子数、根原子附着氢原子数量,最后使用idx来编码局部环境。
2.4三种分子指纹的向量转变方式:
三种分子指纹都是通过RDKit,为了最小化特征冲突,除了研究指纹折叠对模型性能的影响之外,选择(1 073 741 824)长的指纹长度作为默认值。指纹中在训练集中全部为零的元素被删除,他们对模型构建没有贡献。
如下,Figure S4显示了用于模型构建的非全零特征元素的数量。指纹半径设为7个键(n=7),从percent of AE in range可以看出在七个键之外的脂肪胺PKa的影响很小。
2.5指纹特征折叠率:用于表示一个长度i的指纹串在一个元素位置上平均保留多少个非零特征。
文章作者测试了一组较短的指纹长度128、2048、8192、131072,并且与指纹长度进行了比较。较短指纹长度i的特征碰撞程度由其相对于较长指纹长度
的特征折叠氯来评估,如下公式。

四、数据集的划分:
1.训练集以及验证集的划分:
本次实验以氮原子为根原子,采用时间分割(time-split)的方法以14499个PKa数据集作为训练集,以726个PKa数据集作为验证集。用于训练的数据集由于PKa分布区间大,超越生理PH条件的特征使得脂肪胺PKa预测更加准确。
2.数据集的来源:
数据来源于诺华高通量测试的PKa数据以及Bioloom从文献中所收集的数据库,总计有13042个PKa数据来自于诺华内部12799个化合物的测量,有2183个PKa值来自于BioLoom收集的2145种化合物。
3.数据集的清洗以及整理:
3.1数据集的清洗:
这一部分工作由诺华内部工作人员手动完成,清除了数据测量的不确定值(不能重复的值)。
3.2数据集的整理:
每个重复实验所得到的PKa值在”可能的情况下“被分配到主要的电离点,将赋值限制近似的宏观常数。
4.训练”训练集“的方法:五倍交叉验证以及调整和选择ML模型。
五、模型构建和评估度量:
1.分子相似性的评价方式:
1.1整体相似度的评估:
利用规则位型Morgan2指纹和谷本系数计算了两种化合物的整体相似性。c为A和B两个指纹共有的比特数,a和b分别为指纹a和指纹b中设置的位数。

1.2局部相似度的评估:
利用RTorsion指纹和Dice系数计算了两个电离点的局部环境相似性。下述公式中FPi,n中的n为分子指纹的比特数大小,同理
。最后再采用Butina算法进行聚类,聚类值为0.8说明局部相似性良好。

六、结论解读:
1.训练数据集的多样性:
训练集的多样性评价主要参考整个分子结构的全局结构层次和关注电离点附近的局部结构层次。
1.1全局相似度评价:
利用Morgan2指纹以及相似度为0.8的两两谷本系数和聚类的相似矩阵来评价指纹的整体结构多样性训练集得到的诺华数据集和BioLoom数据集之间的全局相似度为0.12,std为0.05,相似度为0.8的聚类产生8478个单例和2065个聚类。
1.2局部相似度评价:
利用RTorsion方法,在相似度为0.8的情况下,通过配对Dice系数和簇的相似举证来评价电离位点周围的局部多样性。使得数据集的平均成对(诺华数据集和BioLoom数据集)RTorsion相似度为0.05,标准差为0.1,相似度为0.8的聚类产生3045个单例和1673个聚类。
1.3数据多样性的总体评价:
通过上述全局相似度评价以及局部相似度评价,聚类结果说明作者的数据集具备了全局相似性以及局部相似性。

2.通过五种机器学习方法以及三种分子指纹预测的结果:
2.1 5次cross valid运行后的模型表现:
5次 cross valid训练集的最大局部相似度为0.89,标准差为0.16,平均78%的训练集化合物的最大局部训练相似度>0.8。通过对每一种机器学习方法和三种分子指纹的模型训练,表现最优的是SVR-RTorsion(RMSE、MAE最低,最高)。

3.模型预测能力验证:
3.1测试集的划分:
文中作者基于每个化合物与测试集的最大局部相似度,将每个5-fold-cross-valid的测试集划分为多个子集,计算每个相似子集的模型性能信息。经过5-fold-cross-valid,①对于电离位点与训练集相似的化合物(最大相似度>0.8)模型给出的均根方误差在0.6甚至更高。②对于电离位点开始偏离训练值得化合物(最大相似度介于0.6和0.8之间),lasso-RTorsion和XGBoost-RTorsion等性能最好的模型仍然达到了均根方误差0.75甚至更高。

3.2 通过线性插值来评价RTorsion的作用:
对于最大相似度为1的测试集,除了PLS-RPair和Lasso-RPair的均根方偏差达到了0.55,剩下的所有模型在插值方面都表现的相当好(均根方偏差介于0.28和0.29之间),这进一步证明了简单的原子对描述符对于PLS和Lasso来说是远远不够的。RTorsion中的路径和原子连通性,对于预测PKa很重要。




3.3 通过线性外推来评价机器学习算法XGBoost的作用:
通过对上述Figure4的观察,XGBoost和RF与RTorsion和RPair一起应用时,在线性插值的中的表现同样良好,但是在测试集5-fold-cross-valid中,XGBoost给出的预测值相较于RF更加准确。在Figure4 (A),(B)中可以看到,XGBoost的均根方误差相较于RF一般低0.1。
3.4 通过训练中的类似物的数量来评价预测性能:
一个电离位点附近有多少个PKa类似物才能达到准确的预测,如Figure 5所示XGBoost-RTorsion模型cross-valid的均根方 对 训练集邻近的数量关系(局部相似度>0.8)的依赖关系。根据图Max Sim 1所示,局部相似度>0.8的样本数量大于50时,均根方误差最小。
通过三张图标对比,局部相似性达到1的测试集样本在样本数量增大的趋势下,均根方误差的变化幅度最小。

3.5 通过调整PKa范围来评价预测性能:
原文中作者评估了模型在不同PKa范围内的表现,Figure 6显示了XGBoost-RTorsion模型在不同局部相似性条件以及不同PKa条件下的均根方偏差。结果显示,对于训练集局部相似性接近的样本点(Max Sim ≥0.8)的均根方误差相差不大,而训练集局部相似性不接近的样本点(Max Sim <0.8),在PKa的两端均根方偏差显著增大。

3.6 指纹长度以及特征碰撞对预测精度的影响:
对于特别大的数据集,在QSPR建模中使用短且折叠的指纹(2048)来减少计算成本,指纹折叠会导致特征冲突和潜在信息丢失。
原文中作者采用长位(1073741824)的指纹用于测试指纹长度对精度的影响,与4个较短指纹(131072、8192、2048、128)的结果进行比较。这些较短的指纹特征折叠率在1.1和178之间。
通过Figure 7的图表显示,XGBoost和 SVR在较短长度下精度明显优于PLS和Lasso。

图标横轴一侧的括号旁为指纹元素的特征数量 ,XGBoost-RTorsion在特征长度以及特征数量为2048(11)时的均根方误差与1073741824(1)时差别不大,丢失的潜在信息相较于其他机器学习算法少。
3.7外部时间分割测试集对训练模型评估:
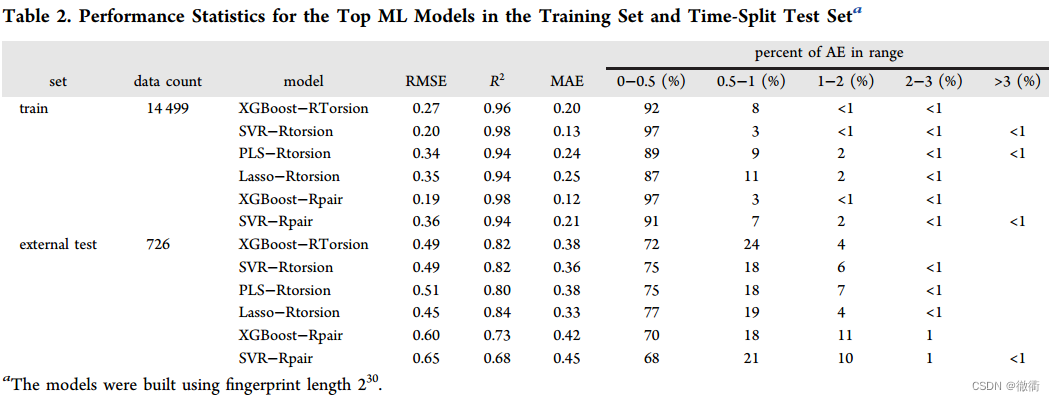
原文中作者采用外部时间分割测试集对训练后的模型进行评估。在该测试集中,56%的电离位点与训练的局部最大相似度<0.8,平均最大局部相似度为0.77,std为0.19。PKa范围、分子量、ClogP覆盖范围与训练集相当,如Table 2 所示。

七、全文总结:
在这项研究中,作者报告了一种基于机器学习方法和RTorsion指纹的PKa预测方法,本文以脂肪胺的结果说明了模型的预测统计性,用时间分割测试集进行了前瞻性预测。
RTorsion被证明是PKa预测的Roubost描述符,并且在多种机器学习方法中表现一致。
Lasso Regression对时间分裂检验集的预测最准确(RMSE 0.45, MAE 0.33, = 0.84),XGBoost和SVR紧随其后。
XGBoost和SVR在短的,特征数多的RTorsion下也可以很好地工作。
八、基础概念补充:
1.RMSE:均根方误差(Root Mean Square Error)
均根方误差指的是预测值与真实值偏差的平方与观测次数n比值的平方根,用于衡量预测值与真实值之间的误差,并且对数据中的异常值较为敏感。
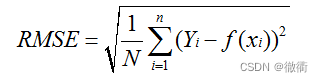
2.MAE: 平均绝对误差(Standard Deviation)
平均绝对误差指的是预测值与真实值偏差的绝对值与观测次数n壁纸的平方根,也用于衡量预测值与真实值之间的误差。

3.RMSE与MAE之间的关系:
RMSE相当于L2范数,MAE相当于L1范数。次数越高,计算结果就越与较大的值有关,而忽略较小的值,所以这就是为什么RMSE针对异常值更敏感的原因(即有一个预测值与真实值相差很大,那么RMSE就会很大)
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








