Xena是一个目前比较好用的下载TCGA数据的网站,不多废话,直接讲操作。
UCSC Xena https://xena.ucsc.edu/
https://xena.ucsc.edu/

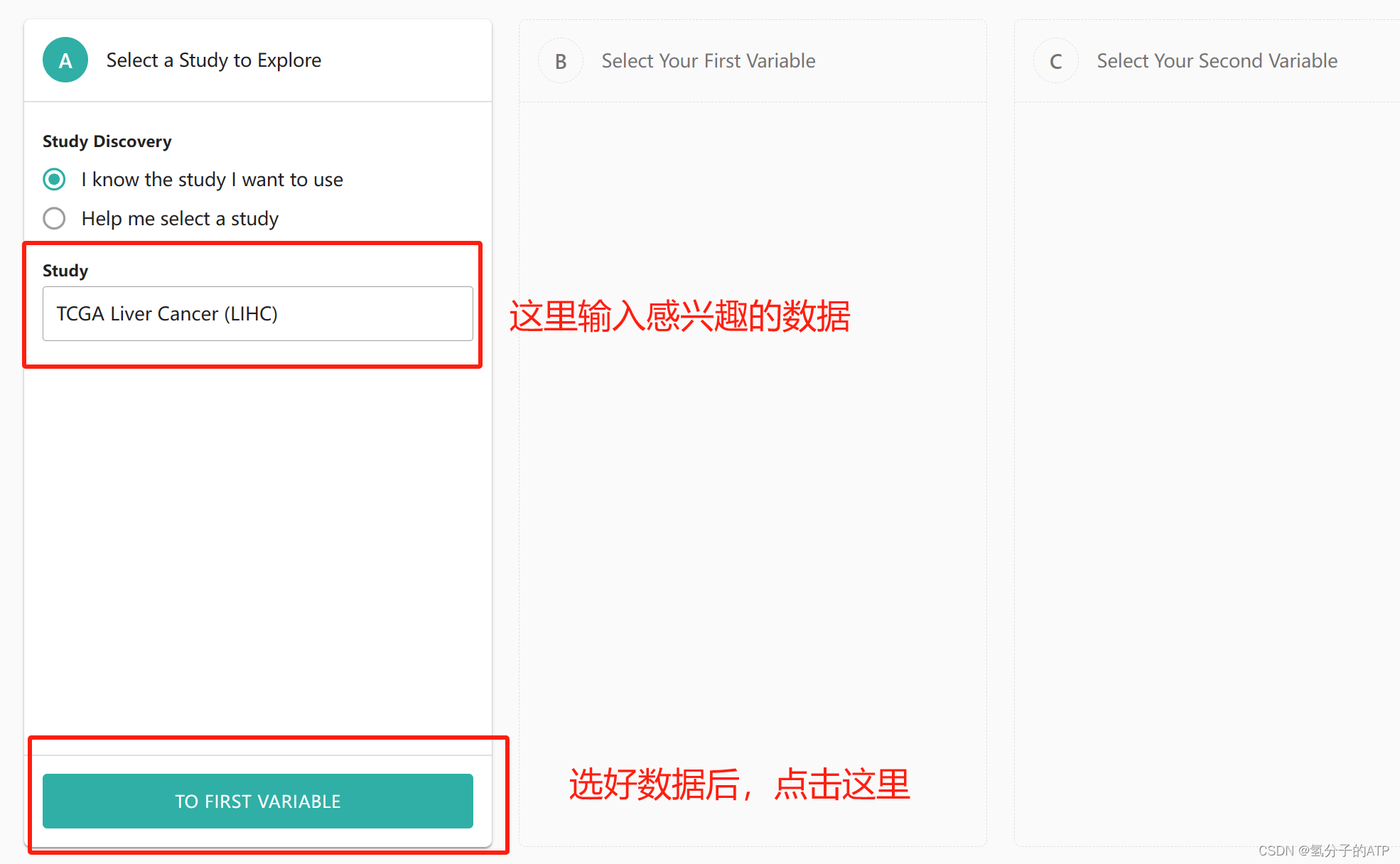
首先,点击“Launch Xena”启动网站,可以直接跳转进入可视化页面。第一步操作是选择数据集,在“Study”一栏中输入感兴趣的数据集,然后选择,再点击页面下方的“TO FIRST VARIABLE”就可以到下一步选择变量了。这里我选择的是TCGA的肝癌数据。
变量可以勾选“Genomic”(基因)或“Phenotypic”(表型),本文先勾选的Phenotypic。
勾选Phenotypic后可以选择数据的各种表型数据进行展示,这里选择了最基本的样品类型“sample type”,勾选所需的表型数据后就可以去选择下一个变量了。

第二个变量我们勾选“Genomic”(基因),获取基因的数据,随后输入我们需要的基因(基因集)的基因名(注意不要写错名字,否则会显示找不到数据),这里我输入的是EGFR。下一步选择所需的数据类型,Xena上的数据包括拷贝数、DNA甲基化、外显子表达、基因表达等多种数据,这里我选择的是最常用的基因表达数据(“gene expression RNAseq”是基因表达,“IlluminaHiSeq”是测序技术)。选好之后就可以确定了。

确定之后就可以看到Xena以热图的方式展示出了刚刚选择的样本类型和基因了。
看到热图后注意到,第二和第三栏中都有一些灰色的部分,这些是缺失了数据信息的样本,我们分析的时候不需要这一部分数据,所以要去掉。
在右上角的长得像取色笔的图标和三条横线的图标可以对样品进行选择等操作。
点开三条横线的图标,里面有“remove samples with nulls”的操作指令,这个指令可以去除掉数据缺失的样品,经过加载之后,热图中的灰色部分就会被清除掉。
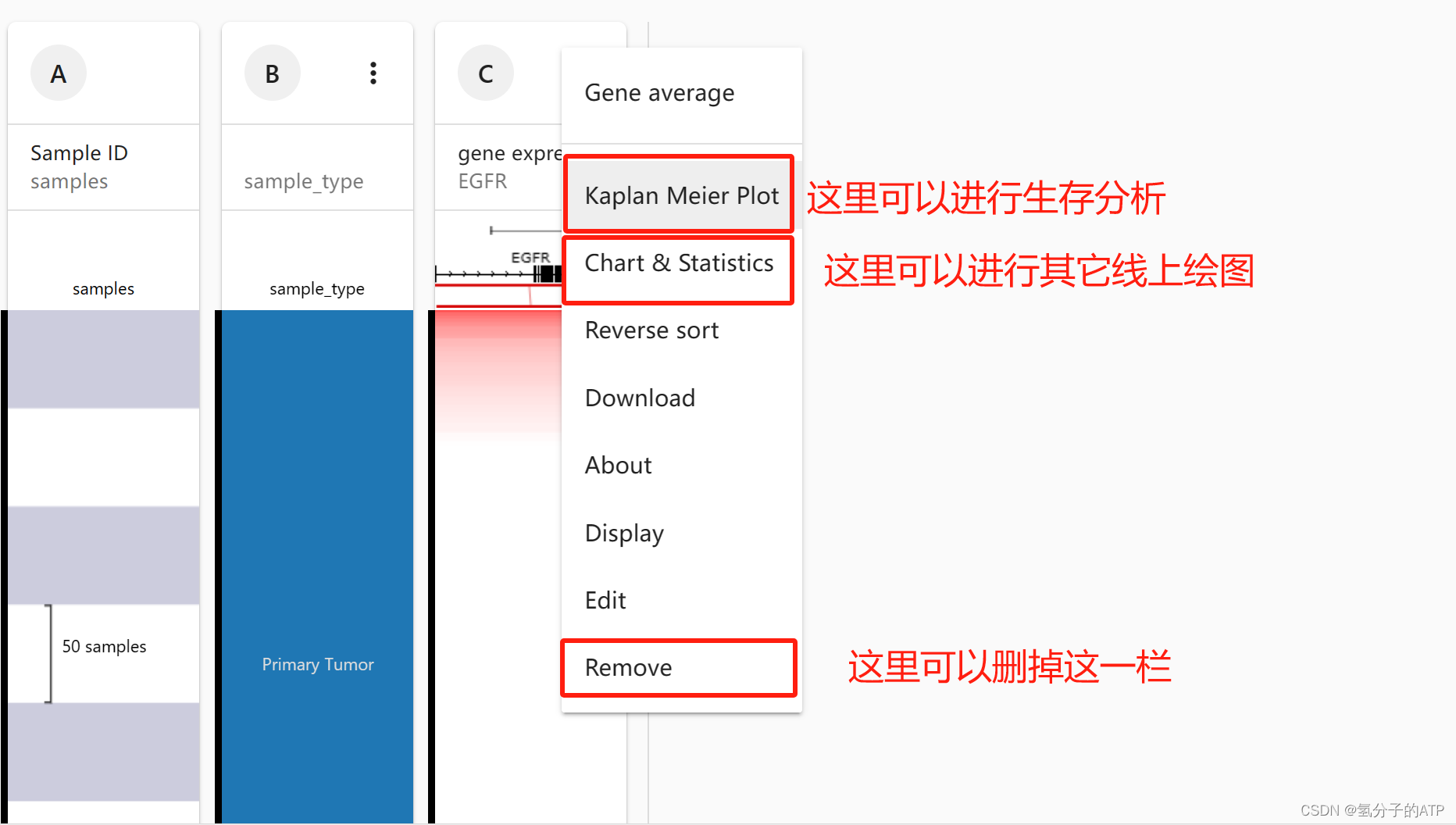
下一步是进行绘图,点击EGFR一栏右上角的三个点,可以看到有“Charts & Statistics”这个选项,点开这个选项,就可以线上作图了。Xena提供的绘图有组间比较(“COMPARE SUBGROUP”),分布(“SEE A COLUMN DISTRIBUTION”)和散点图(“MAKE A SCATTER PLOT”)。这里我示范的是EGFR基因表达在不同组织样品中的组间比较。

另外一个常用的线上绘图就是生存分析的绘图,点开EGFR一栏右上角的三个点,里面有“Kaplan Meier Plot”的一个选项,这个选项就是作生存分析的KM图。但是,做生存分析之前要把正常组织的样本(Solid Tissue Normal)和肿瘤组织(Primary Tumor)的样本区分开来。
使用右上角的取样笔,点击第二栏的“Primary Tumor”,就可以选取肿瘤组织的样本了,然后点开右上角的三条横线,选择“keep samples”就可以保留选中的样本,这样我们就可以进行生存分析了。

然后我们选择“Kaplan Meier Plot”就可以看到EGFR在肝癌中的生存分析图了。分析结果的p = 0.1181,有点拉跨。总之使用Xena进行线上分析绘图的操作就基本上讲完了。
























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








