
气泡图简介
气泡图(Bubble plot)用于呈现三维数据的关系。这种图通常使用气泡的位置、大小和颜色来表示变量之间的关系,使得观察者能够直观的理解复杂数据集。其基本组成由X, Y坐标的位置,气泡的大小和气泡的颜色构成。气泡图的基础是二维平面上的坐标系,其中每个气泡由其在X轴和Y轴上的位置决定。这些坐标可以表示两个变量或维度,例如时间、空间或不同的类别。每个气泡的大小代表了第三个维度的数值,通常气泡越大表示的数值越大。这个大小维度可以表示各种新信息,比如数据点的频率、重要性等。颜色是气泡图中的第三个可用于信息的维度。通过对气泡着色,可以进一步传达关于数据的分类、群组或其他属性的信息。颜色也可以用于强调特定的趋势或模式。
标签:#微生物组数据分析 #MicrobiomeStatPlot #气泡图 #R语言可视化 #Bubble plot
作者:First draft(初稿):Defeng Bai(白德凤);Proofreading(校对):Ma Chuang(马闯) and Jiani Xun(荀佳妮);Text tutorial(文字教程):Defeng Bai(白德凤)
源代码及测试数据链接:
https://github.com/YongxinLiu/MicrobiomeStatPlot/项目中目录 3.Visualization_and_interpretation/BubblePlot
或公众号后台回复“MicrobiomeStatPlot”领取
气泡图应用案例
这是Jakob Stokholm课题组2023年发表于Nature Medicine上的文章,第一作者为Cristina Leal Rodríguez,题目为:The infant gut virome is associated with preschool asthma risk independently of bacteria. https://doi.org/10.1038/s41591-023-02685-x.
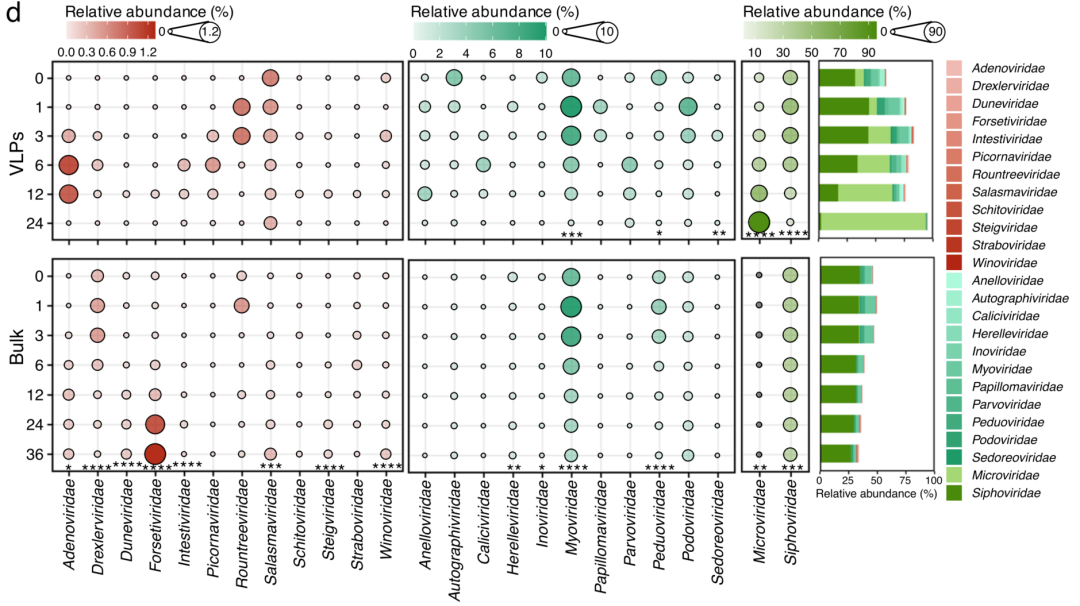
图 2d | 生命前两年或三年病毒家族相对丰度的动态。
仅绘制了VLP富集型基因组中流行率>1%的病毒家族。为了更好地显示每个病毒家族的变化,根据每个时间点VLP富集物的平均相对丰度,将病毒家族分为三组(即最大平均相对丰度≤1%(左,n=12,红色),最大平均相对丰富度>1%和<40%(中,n=11,浅绿色),最大相对丰度≥40%(右,n=2,深绿色))。条形图显示了所有25个病毒家族的相对丰度比例,如右侧图例所示。P值是通过以“研究”为随机因素的线性混合模型获得的。P<0.0001,P<0.001,**P<0.01,*P<0.05。
结果
在流行率>1%的42个病毒家族中,有25个家族占1608个富含VLP的宏基因组中病毒丰度的99%以上。其中,有五个家族发生了重大变化,只有微小病毒科的数量随着婴儿年龄的增长而增加(图2d)。当在大量宏基因组中检查这25个病毒家族时,检测到21个家族,占5990个大量宏基因组病毒丰度的99%以上。其中,14个家族在婴儿年龄增长时的丰度存在统计学显著差异(P<0.05),其中一半的家族数量增加,如腺病毒科、盾形病毒科和毛喉病毒科;另一半减少,如细胞病毒科、Myoviridae、Microviridae和Pedooviride。此外,前12个月,福氏病毒科的平均相对丰度较低,<0.3%,但在第24个月和第36个月分别稳步增加到0.97%和1.24%。值得注意的是,我们还发现,一些科的相对丰度在第1个月或第3个月达到峰值(即肌病毒科、Pedooviride、Rountreeviridae、Siphoviridae),然后逐渐减少,这表明某些病毒分类群在生命早期并没有发生一致的变化(图2d)。
气泡图R语言实战
源代码及测试数据链接:
https://github.com/YongxinLiu/MicrobiomeStatPlot/
或公众号后台回复“MicrobiomeStatPlot”领取
软件包安装
# 基于CRAN安装R包,检测没有则安装
p_list = c("ggplot2", "dplyr", "readxl", "gapminder", "viridis", "scales",
"RColorBrewer", "magrittr", "stringr")
for(p in p_list){if (!requireNamespace(p)){install.packages(p)}
library(p, character.only = TRUE, quietly = TRUE, warn.conflicts = FALSE)}
# 加载R包 Load the package
suppressWarnings(suppressMessages(library(ggplot2)))
suppressWarnings(suppressMessages(library(dplyr)))
suppressWarnings(suppressMessages(library(readxl)))
suppressWarnings(suppressMessages(library(gapminder)))
suppressWarnings(suppressMessages(library(viridis)))
suppressWarnings(suppressMessages(library(scales)))
suppressWarnings(suppressMessages(library(RColorBrewer)))
suppressWarnings(suppressMessages(library(magrittr)))
suppressWarnings(suppressMessages(library(stringr)))实战1
横纵坐标为分类变量。示例数据来自于:nature期刊Single-nucleus profiling of human dilated and hypertrophic cardiomyopathy.https://doi.org/10.1038/s41586-022-04817-8.
# 读取数据
# Read data
sing.df <- read_xlsx("data/41586_2022_4817_MOESM18_ESM.xlsx")
# 调整因子顺序
# Change factor order
sing.df$Gene <- factor(sing.df$Gene, levels = rev(unique(sing.df$Gene)))
sing.df$CellType <- factor(sing.df$CellType, levels = unique(sing.df$CellType))
# 绘制优化图形
# Plot
p1 <- ggplot(sing.df, aes(x = CellType, y = Gene, fill = Avg_Expr, size = Pct_Expr0)) +
geom_point(shape = 21, color = "darkgray", stroke = 0.5) +
theme_minimal(base_size = 14) +
scale_y_discrete(breaks = levels(sing.df$Gene),
limits = c(levels(sing.df$Gene)[1:16], NA,
levels(sing.df$Gene)[17:22], NA,
levels(sing.df$Gene)[23:27])) +
scale_size(range = c(1, 6), breaks = seq(0.2, 1, 0.2),
labels = scales::percent_format(accuracy = 1)) +
scale_fill_gradientn(colors = c("white", "#4CAF50", "#2E7D32"),
na.value = "lightgray") +
theme(axis.text.x = element_text(angle = 60, vjust = 1, hjust = 1, face = "bold"),
axis.text.y = element_text(face &







 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 259
259

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








