1. 加载参考基因组
这里我加载的依然是hg19,只是示范流程,换成自己的参考基因组就行
Tools-->Run igvtools

会多生成一个hg19.fa.fai文件
点击Genomes-->create a .genome file

2. 导入注释文件
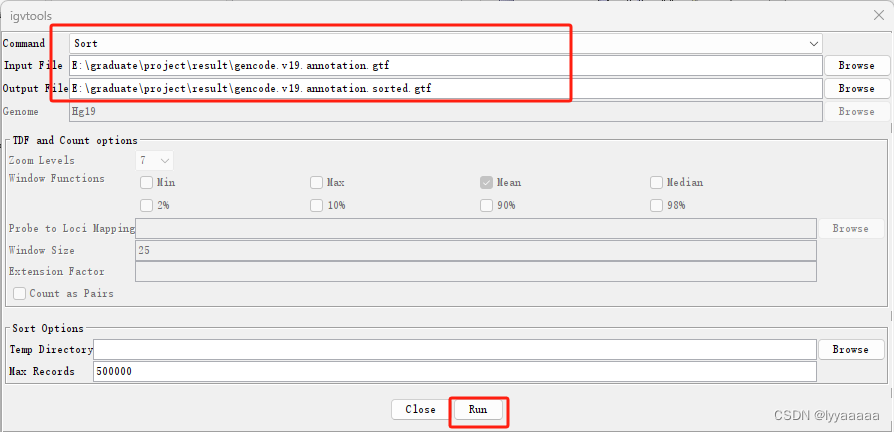
会多生成一个.sorted.gtf文件
点击File-->load from file加载.sorted.gtf

这步是创建索引,点Go创建索引,会多一个以idx结尾的文件
加载sorted.gtf

此时看上去什么都没有,但是这时候选择一条具体的染色体就会出现东西

3. 导出基因名和位置信息
值得注意的是导入自己的参考基因组并不能根据基因名去定位,所以需要导出基因所在的位置,根据位置去查询
查看gtf前10行
head -n 10 gencode.v19.annotation.sorted.gtf
less -S gencode.v19.annotation.sorted.gtf | grep -w "gene" | awk '{for (i=1; i<=NF; i++) if ($i == "gene_name") print $1"\t"$4"\t"$5"\t"$(i+1)}' | sed 's/[";]//g' > gene.info3.xls

注意查找基因的时候,后面冒号是英文



参考链接:
IGV 导入本地基因组及注释文件 - 简书 (jianshu.com)
























 2771
2771

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








