简介
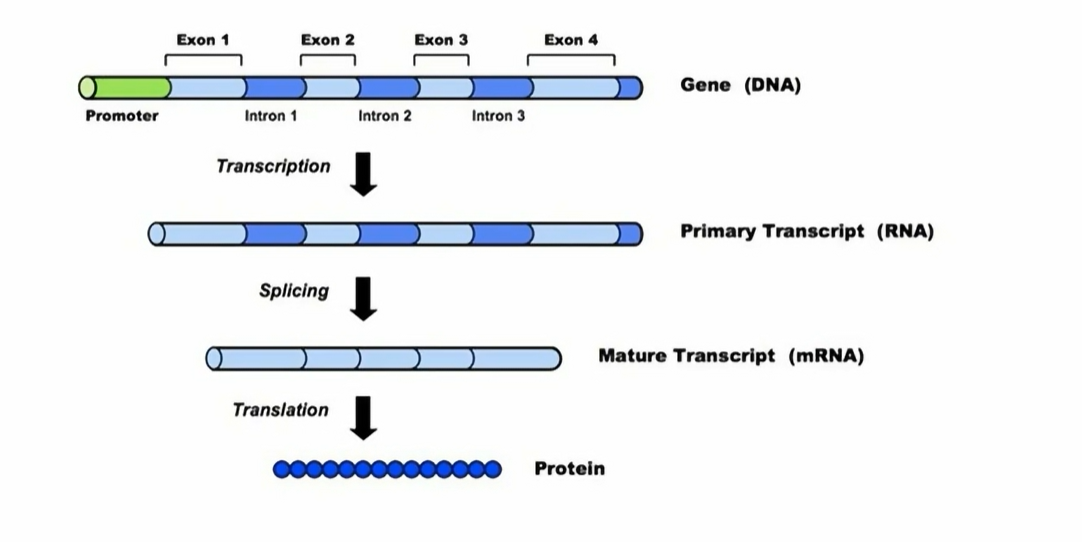
RNA测序(RNA-Seq、转录组)是一种高通量测序技术,用于研究细胞中的RNA群体,包括mRNA、非编码RNA、小RNA等。这项技术为我们提供了一个全面了解基因表达、识别转录本结构、研究RNA编辑和变异以及发现新的RNA分子的手段。RNA-Seq的基本步骤包括RNA提取、RNA分离和富集、cDNA合成、文库构建和测序。
事实上,比起RNA——表型,蛋白质能更好地与表型联系起来。毕竟RNA大多数情况承担了遗传信息传递的中间信使的职责。但是要做蛋白质组的话,目前的技术还有一定的局限性:蛋白质组数据更难解读,异质性更大,成本更高(要使用质谱),蛋白质存在翻译后修饰……
转录组实际上应用的场景更多,而且现在价格也相当便宜。
来源:21.stat115 chapter 4.1 rna-seq applications_哔哩哔哩_bilibili
RNA-seq的应用
最常见的:
-
在特定情况下的基因表达差异(比如把斑马鱼放到不同温度下,看一下有哪些基因和低温响应有关。或者正常生理状况和患病的状况)
-
基因敲除后的基因表达(是否有剂量补偿效应?)
-
新转录本或新基因的发现(人类有2w左右基因,但是有5w左右的转录本。大多数情况下,基因组数据只是做一个参考,我们并不能直接从中知道mRNA表达产物的情况。而且还有可变剪切的存在)
-
发现基因突变(在转录本中就发现单个碱基都突变,而不需要做全基因组测序或者基因克隆)
-
在不知道基因组的情况下预测基因(在RNAseq时,可以选择无参考基因组的比对方式)
-
发现融合基因(易位导致的致癌基因)
RNA QC(质控)
因为RNA保存比DNA难很多,降解的RNA往往非常难用于测序建库。比如储存时间过长,长的RNA往往更加不稳定…….
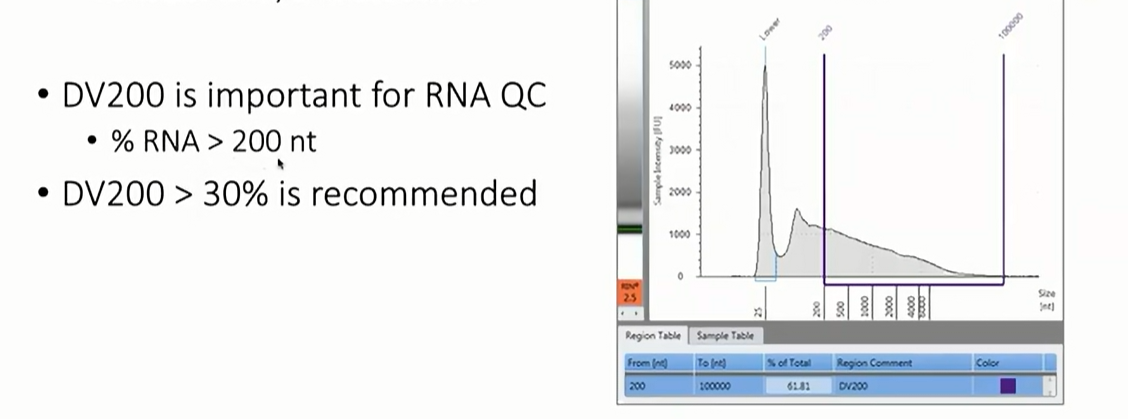
电泳检测RNA质量
对RNA跑胶,使用DV200指标来检测RNA的质量,即>200bt 的片段占的比值,因为mRNA的长度往往超过200bp。DV200>30%是一个不错的值
from:22.stat115 chapter 4.2 rna-seq experimental design_哔哩哔哩_bilibili
计算机的方式
显然,有不错的软件来进行QC的部分操作,如FASTQC,快捷,并且有可视化的方式来查看RNAseq的质量。
通常刚开始的几个bp总是质量较差
实验设计
建库前处理
-
使用试剂盒或者一些实验操作来去除rRNA、tRNA(这些RNA的含量占比极高,但通常不是我们想要研究的对象)
-
根据mRNA的末端ployA,来得到mRNA,这是最常用的(这里收集到的RNA是经过可变剪切、移除了内含子,没有tRNA和rRNA的)
-
strand特异的方法,可以识别lncRNA(因为lncRNA可能没有标准的翻译框)
根据不同的实验目的选择不同的实验方案。
现在常见的测序是illumina 双端 150bp的配置。
话费200$ 就可以得到5亿个这样的reads,通常会有几十Gb的下机数据(raw data)
重复设计
RNA-seq实验中需要做重复,其中分为技术重复和生物学重复。
-
简单来说,
技术重复(technical repeat)就是使用同一个样本,用相同的流程进行测序。多次重复,求SE,然后看准确度。对于RNAseq来说,这样操作的差异并不会很大,在实际操作中不推荐(即分开建库、分开测序)。 -
生物学重复就更加普遍了,也是非常basic的操作,防止批次效应。同一个处理,只要需要3次以上的生物学重复。
应该设计多少的生物学重复?
-
1个:作为预实验,粗略看一下效果
-
≥2:适合对于细胞系的实验
-
≥3:对于动物实验
-
更多:对于人类样本,需要更加严谨大规模的重复
RNA-seq Alignment
BWA?
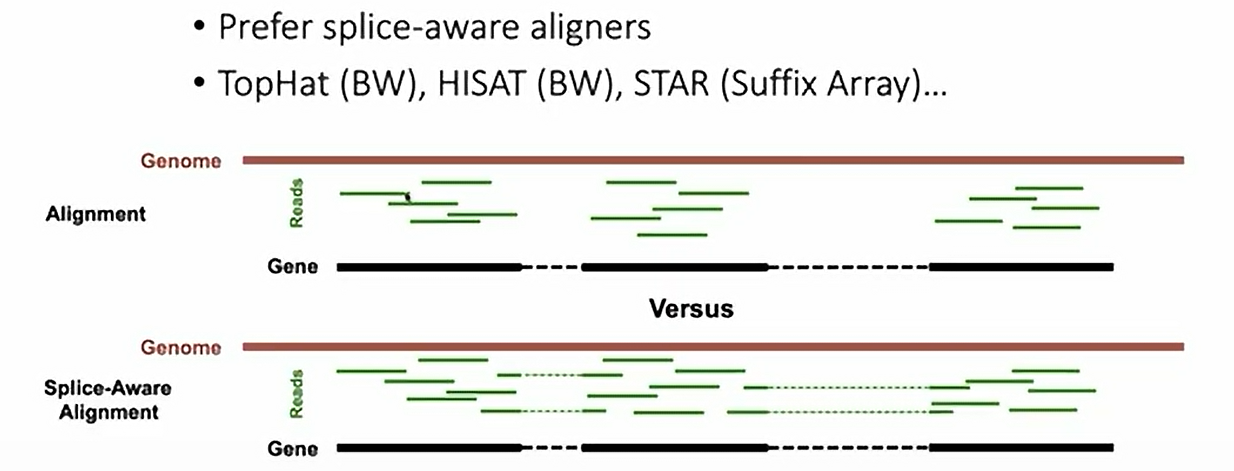
还记得我第一次做转录组的时候,就用了BWA进行比对。印象很深刻,但是老师和我说,RNA-seq有很多比对软件,唯独不能用BWA。
因为BWA主要用于DNA比对,不能跨过内含子,而我们知道对RNA反转录再测序的话,就会丢失内含子的序列。
所以需要用到TopHat、HISAT、STAR这样的剪切敏感比对(splice aware alignment)软件
























 7万+
7万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








