01 m6A是什么
目前在细胞RNA中已经识别到了超过100种化学修饰,其中RNA甲基化修饰在生命活动中有着非常重要的作用(Xu et al 2020)。RNA甲基化是指在甲基转移酶的催化下,在RNA分子上的某一个原子上添加一个甲基基团(CH3)。RNA甲基化修饰类型有很多,比如:m6A RNA甲基化﹑m5C RNA甲基化﹑m1A RNA甲基化、m7G RNA甲基化等(Oerum et al 2021)(图1)。

图1 不同类型RNA的转录后修饰(Oerum et al 2021)
m6A RNA甲基化,即RNA分子腺嘌呤第 6 位氮原子上的甲基化修饰(N6-methyladenosine,m6A),是真核生物mRNA最常见的一种转录后修饰,占RNA甲基化修饰的80% (Qin et al 2020)(图2)。在调控mRNA稳定性,剪切和翻译等方面具有重要的作用(Xu et al 2020, Qin et al 2020)。

图2 mRNA的m6A修饰过程(Qin et al 2020)
02 参与m6A修饰的酶
m6A这种甲基化修饰是可逆的,由m6A甲基转移酶(writers)催化产生,被m6A去甲基酶(erasers)去除,被m6A结合蛋白(readers)识别并发挥功能(Ma et al 2019, Zhu et al 2023)(图3)。
-
Writers:包括METTL3/14、WTAP和KIAA1429等,主要作用是催化mRNA上腺苷酸发生m6A修饰。
-
Erasers:包括FTO和ALKHB5等,作用是对已发生m6A修饰的碱基进行去甲基化修饰。
-
Reader:主要功能是识别发生m6A修饰的碱基,从而激活下游的调控通路如RNA降解、miRNA加工等。

图3 m6A的修饰调控(Zhu et al 2023)
03 m6A修饰的功能
m6A修饰主要发生在RRACH (R = G or A and H = A,C, or U)这类motif上,这类motif主要在终止密码子和3’UTR处富集,即m6A主要发生在终止密码子和3’UTR附近(An and Duan 2022)。m6A修饰广泛存在于各类生物中,包括病毒、酵母、植物和人类等。该修饰对细胞行使各种功能起着重要作用,如结合其他reader蛋白,促进miRNA成熟、介导mRNA前体的可变剪接、介导mRNA降解、驱动翻译,还有诱导出核、X染色体失活等(Qin et al 2020)(图4)。

图4 m6A修饰的功能(Qin et al 2020)
04 m6A检测方法-MeRIP-seq
目前检测m6A的技术手段有高通量测序、比色法及液相色谱质谱联用(LC-MS)。比色法和LC-MS能够检测mRNA整体的m6A水平,LC-MS/MS是在液相质谱的基础上采用串联质谱,获得分子离子峰和碎片离子峰,对碱基同时进行定性和定量分析。但这两种方法都无法对m6A进行定位,常用于课题研究初期。高通量测序目前主要以m6A-seq/MeRIP-seq为主,它是目前研究m6A修饰使用最广泛的技术之一。
MeRIP-seq将甲基化DNA 免疫共沉淀(methylated DNA immunoprecipitation, MeDIP)技术、RNA 结合蛋白免疫共沉淀(RNA immunoprecipitation,RIP)技术和RNA 测序(RNA sequencing,RNA-seq)技术组合起来,高精度地检测全基因组(或全转录组)范围内的RNA甲基化。其原理为通过特异识别m6A修饰的抗体,对细胞内具有m6A修饰的RNA片段进行免疫共沉淀。对沉淀下来的RNA片段进行高通量测序,结合生物信息学分析,即可在全基因组范围内对m6A修饰情况进行系统研究。
4.1 常规MeRIP-seq
模式物种或一些常规的非模式物种通常RNA含量较高,容易提取,针对这些样本采用的实验流程如下(图5):1)Total RNA样品检测
-
琼脂糖凝胶电泳分析RNA降解程度以及是否有污染;
-
Qubit 2.0对RNA浓度进行精确定量;
-
Agilent 2100精确检测RNA的完整性。
2)mRNA获得及片段化:
-
通过polyA捕获mRNA,或rRNA剔除去掉rRNA;
-
将获得的mRNA打断成片段。
3)m6A富集
-
使用m6A抗体对片段化后的mRNA进行免疫共沉淀,富集并纯化m6A修饰的RNA片段。
4)文库构建
5)高通量测序和生物信息学分析

图5 常规MeRIP-seq实验流程
4.2 微量MeRIP-seq
在2013年首次发表的MeRIP-seq protocol中,需要300µg的RNA才能开展实验(Dominissini et al 2013),这样的RNA起始量极大的限制了该技术的应用。对于一些珍稀及RNA含量较低的样本,MeRIP-seq遥不可及。
2018年,多伦多大学何厚胜课题组推出了微量RNA的MeRIP-seq技术,该技术从RNA的起始量、RNA片段化的条件、清洗/洗脱条件等角度优化了MeRIP-seq实验流程,使得MeRIP-seq所需肿瘤的总RNA最低降至2µg,打破了传统protocol的限制(Zeng et al 2018),主要流程如下(图6):
1)RNA片段化
-
RNA片段化的温度为70℃,片段化时间具体根据RNA总量确定。
-
将RNA片段化的大小从100nt提高到200nt,这样可以将RNA片段的获得量提高2倍。
2)清洗条件
-
将IP buffer更换为低/高盐 buffer进行清洗,提高数据的信噪比。
3)建库方式
-
直接针对total RNA进行m6A抗体富集,利用残留的rRNA增加RNA起始量,之后剔除rRNA进行建库。

图6 微量MeRIP-seq实验流程
05 MeRIP-seq后续验证
无论是仅描述修饰位点分布特征的文章,还是深入研究修饰调控基因表达机制的文章,在进行高通量测序后,通常都需要对鉴定到的主要修饰位点进行后续的实验验证。
5.1 MeRIP-qPCR
1)MeRIP-qPCR的目的
与普通的qPCR有所不同,MeRIP-qPCR是验证某个目标基因上的某个修饰位点(以一小段区域的形式,MeRIP法精确不到单碱基)的修饰水平。得到的结果往往是一个%(IP/Input)的数值,它体现的是该位点的修饰水平(目标基因发生该修饰的转录本占该基因总转录本的比例),而普通qPCR验证的是某个基因的表达水平(共表达了多少转录本)。
2)MeRIP-qPCR的实验方法
MeRIP-qPCR就是MeRIP实验后进行一个qPCR。在RNA量充足的情况下,可以直接使用MeRIP后的产物分别进行逆转录和qPCR,其流程与普通qPCR相同。
3)MeRIP-qPCR为什么没有内参基因?
MeRIP-qPCR不需要使用内参来进行样本间的对比,此实验主要是通过计算发生修饰的转录本(IP)占该基因总转录本(Input)的比例,来体现该位点的修饰水平。
4)IP/Input/IgG都需要展示?
MeRIP-qPCR的结果用柱状图展示更为直观,文章中常用的有以下3种:
-
只有IP和Input的数据:展示%(IP/Input)值(图7)。

图7 %(IP/Input)值展示MeRIP-qPCR结果(Liu et al 2019, Tong et al 2021)
-
IP、IgG和Input均做:展示%(IP/Input)值、%(IgG/Input)值(图8)。

图8 %(IP/Input)和%(IgG/Input)值展示MeRIP-qPCR结果 (Wang et al 2021, Chen et al 2021)
-
IP、IgG和Input均做:展示Fold enrichment值(%(IP/Input)和%(IgG/Input)的比值)(图9)。
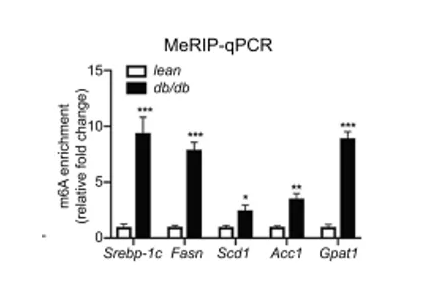
图9 %(IP/Input)和%(IgG/Input)的比值展示MeRIP-qPCR结果 (Zhou et al 2021)
5.2 SELECT法
通过MeRIP-seq得到的差异peak是一个个片段,其分辨率并不是单碱基。上述的MeRIP-qPCR也是对MeRIP-seq获得的差异修饰基因或候选基因进行验证。所以想要检测特定单碱基的m6A修饰水平需要另外的技术,如SELECT法、OCPDIA法、m6A-Rol-LAMP等。
SELECT法是北京大学贾桂芳课题组在2018年发表在Angewandte Chemie International Edition上的一种检测单碱基位点m6A修饰水平的技术(Xiao et al 2018)。该技术主要利用m6A会阻碍DNA聚合酶的延伸活性和连接酶的连接效率的特点进行检测。
-
在m6A修饰位点上下游设计两个DNA探针:Up Probe和Down Probe(探针两端均带有固定序列,可成为最后的qPCR引物),探针与RNA退火杂交(m6A修饰位点形成gap);
-
用Bst DNA polymerase和dTTP(或dNTP)进行延伸反应(若该位点存在m6A修饰,则延伸效率会降低);
-
用SplintR ligase(该酶可以连接与RNA片段互补的两个ssDNA)进行连接反应(若该位点存在m6A修饰,则会降低连接效率);
-
最后利用连接反应之后的DNA产物作为qPCR模板,通过qPCR定量检测该位点的m6A修饰水平。

图10 SELECT法检测示意图(Xiao et al 2018)
5.3 OCPDIA法
该技术在一定程度上与SELECT法类似,也是在单碱基分辨率下对发生m6A的RNA进行定量,但也有所不同,区别如下:
-
用挂锁探针取代m6A位点上下游探针;
-
由Bst 2.0 warm start DNA聚合酶和SplintR ligase分别执行延伸和连接反应,由于连接后的挂锁探针是环状的,因此利用Phi 29 DNA聚合酶进行滚环扩增反应(rolling circle amplication,RCA);
-
之后利用Nb.BsmI限制性内切酶切割扩增产物中的固定分子信标(Molecular beacons,MBs)序列,然后MBs会进一步结合在扩增产物上实现二次荧光信号放大;
-
所有反应过程均在等温扩增反应下进行。

图11 OCPDIA法检测示意图(Ma et al 2023)
结 语
随着对RNA甲基化修饰,尤其是m6A修饰在细胞功能中作用的深入理解,MeRIP-seq技术已成为研究这一领域的重要工具。从常规MeRIP-seq到微量MeRIP-seq的优化,这一技术的应用范围不断扩大,为珍稀样本的研究提供了可能。此外,MeRIP-qPCR、SELECT法和OCPDIA法等后续验证技术的应用,进一步提高了我们对m6A修饰位点的认识精度。这些技术的综合应用不仅加深了我们对RNA甲基化在疾病发生发展中作用的理解,也为未来的治疗策略提供了新的视角。随着技术的不断进步和创新, MeRIP-seq及其相关技术将在未来的分子生物学和医学研究中发挥更加重要的作用。

-
参考文献:
-
An and Duan. The role of m6A RNA methylation in cancer metabolism. Mol Cancer, 2022
-
Chen et al. Nuclear m6A reader YTHDC1 regulates the scaffold function of LINE1 RNA in mouse ESCs and early embryos. Protein Cell, 2021
-
Dominissini et al. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nat Protoc, 2013
-
Liu et al. m6A mRNA methylation regulates CTNNB1 to promote the proliferation of hepatoblastoma. Mol Cancer, 2019
-
Ma et al. One-base-gap circular probe-mediated dual amplification for isothermal detection of N6‑methyladenosine modifications. Anal Chem, 2023
-
Ma et al. The interplay between m6A RNA methylation and noncoding RNA in cancer. J Hematol Oncol, 2019
-
Oerum et al. A comprehensive review of m6A/m6Am RNA methyltransferase structures. Nucleic Acids Research, 2021
-
Qin et al. Role of m6A RNA methylation in cardiovascular disease. Int J Mol Med, 2020
-
Tong et al. Pooled CRISPR screening identifies m6A as a positive regulator of macrophage activation. Sci Adv, 2021
-
Wang et al. METTL3 promotes tumour development by decreasing APC expression mediated by APC mRNA N6-methyladenosine-dependent YTHDF binding. Nat Commun, 2021
-
Xiao et al. An elongation and ligation-based qPCR amplification method for the radiolabeling-free detection of locus-specific N6-methyladenosine modifications. Angew Chem Int Ed, 2018
-
Xu et al. m6A mRNA methylation is essential for oligodendrocyte maturation and CNS myelination. Neuron, 2020
-
Zeng et al. Refined RIP-seq protocol for epitranscriptome analysis with low input materials. Plos Biol, 2018
-
Zhou et al. N6 -Methyladenosine Reader Protein YT521-B Homology Domain-Containing 2 Suppresses Liver Steatosis by Regulation of mRNA Stability of Lipogenic Genes. Hepatology, 2021
-
Zhu et al. Crosstalk between m6A modification and alternative splicing during cancer progression. Clin Transl Med, 2023
























 5680
5680

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








