文章首发于简书链接(https://www.jianshu.com/p/c876a5636554)。发此备份。
基因家族的鉴定-基于windows系统上的本地blast
基因组的序列提取,详情请看我之前的教程TBtools的sequence toolkit常用功能介绍: - 简书
下面讲如何在windows系统上用blast做基因家族的鉴定:
1,下载
在NCBI网站上,下载blast工具
https://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=Download
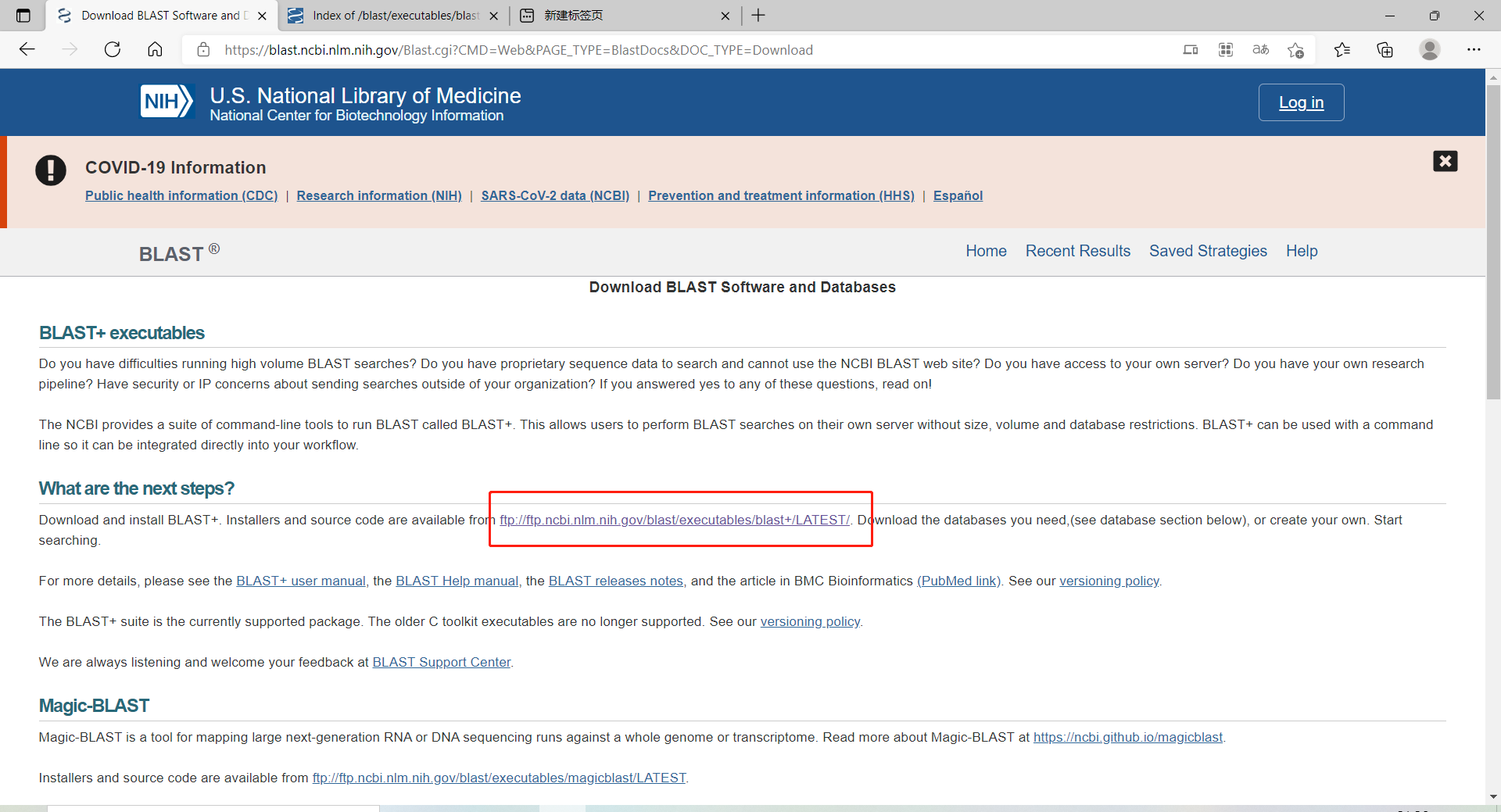
点击进入ftp下载地址,选择自己电脑合适版本的blast工具,这下载好慢呀
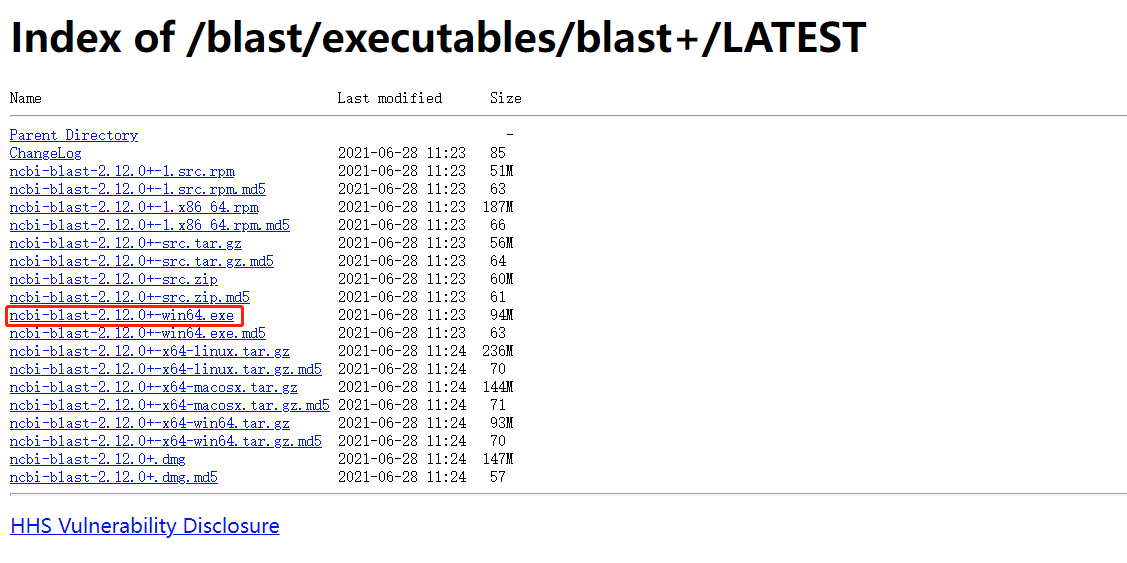
2,安装并准备好文件
安装好后,进入名为“bin”的文件夹,我用2.11版本的blast举例,由前人的文献我们知道了拟南芥的全部bZIP转录因子,我们想要知道毛果杨中的bZIP转录因子,其中一种方法就用拟南芥的bZIP成员进行blast比对。首先将这些拟南芥的bZIP转录因子的蛋白序列提取整理好成fasta格式的文件,将提取好的毛果杨4.0版本的全部蛋白质,二者都拷贝一份到这个bin文件夹,文件命名不要有空格和中文。
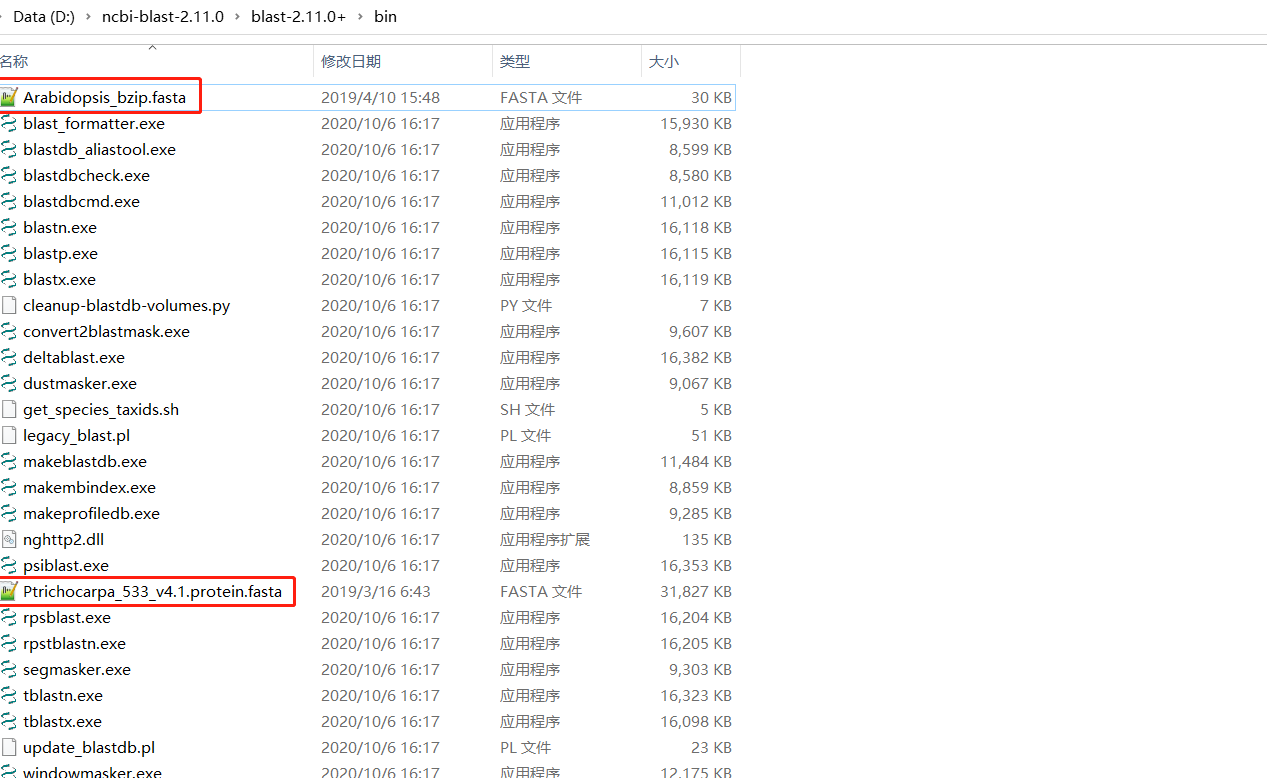
3,操作
在电脑的左下方,输入cmd,进入命令提示符:
我的bin文件夹的地址是D:\ncbi-blast-2.11.0\blast-2.11.0+\bin,所以我先进入D盘,输入命令:
D:
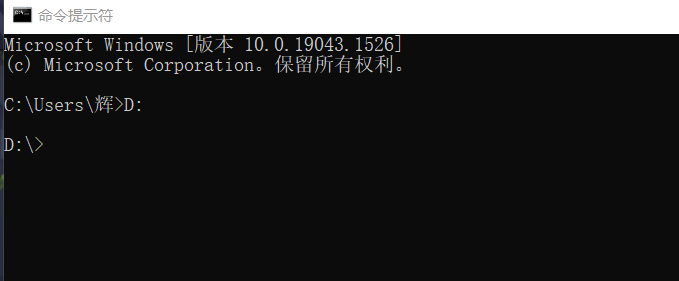
再输入命令,进入bin文件夹:
cd ncbi-blast-2.11.0\blast-2.11.0+\bin
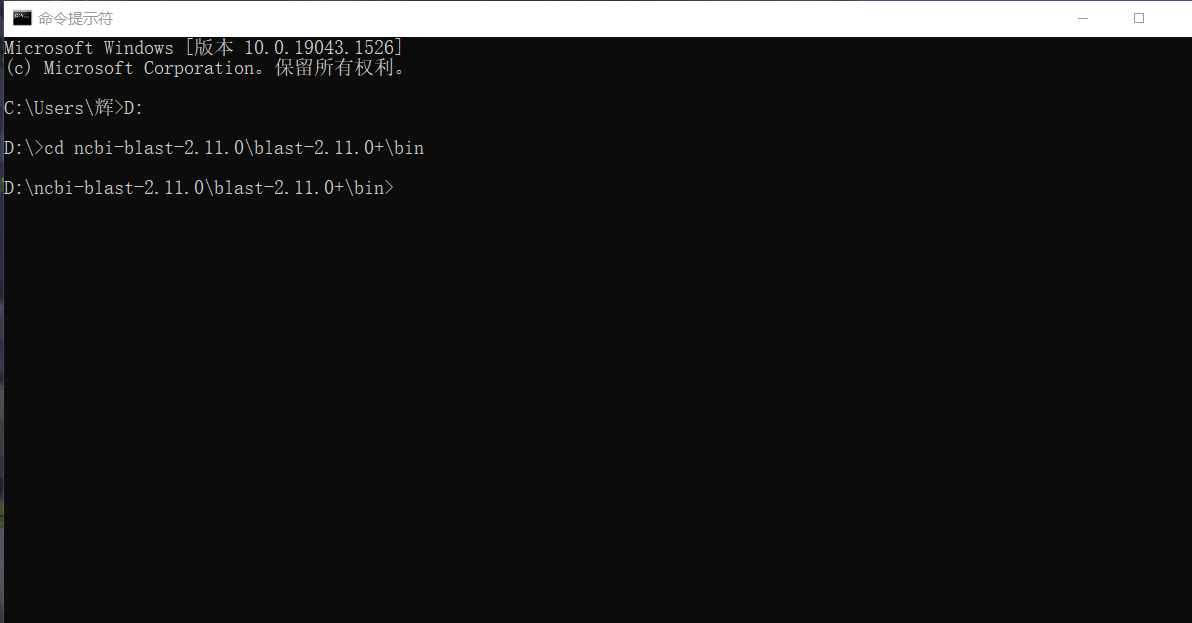
再对毛果杨的全部蛋白建库,生成dbname中间文件,命令:
makeblastdb -in Ptrichocarpa_533_v4.1.protein.fasta -dbtype prot -out dbname
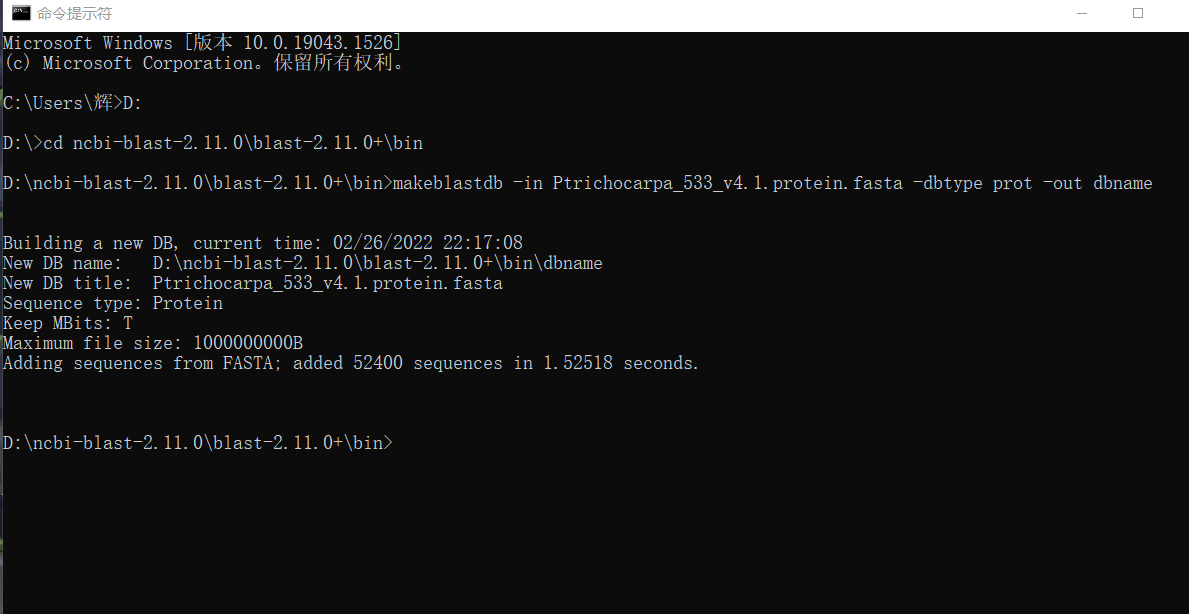
再用拟南芥的bZIP转录因子的作为比对序列,进行blast比对,并输出seq.blast的结果文件:
blastp -query Arabidopsis_bzip.fasta -out seq.blast -db dbname -outfmt 6 -evalue 1e-5 -num_threads 4
等待约20s,结束了
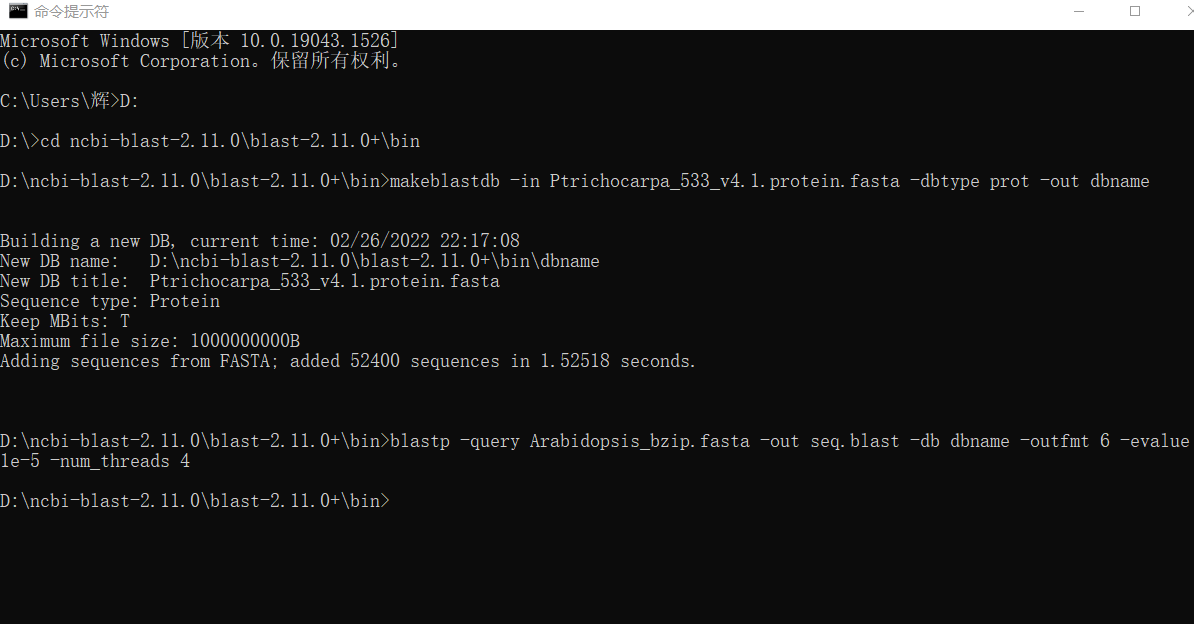
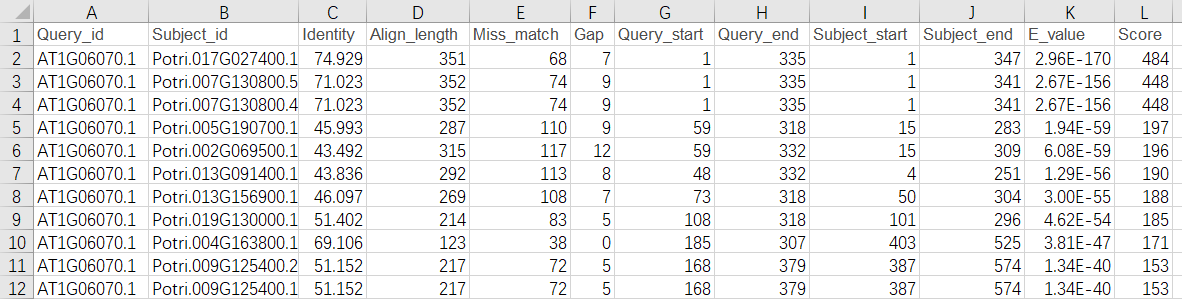
我们看回bin文件夹,会多出一些文件,包括seq.blast结果文件,用excel打开它并保存为excel的格式
结果:
更多关于blast的教程和结果解读分析,可以自行百度,网上的教程非常多。






















 4612
4612

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










