之前的文章写了Slingshot的简单用法,可以从整体看单细胞轨迹,但是不太容易看单个轨迹到底是哪些细胞群,实现方法:
1.运行Slingshot
sds <- slingshot(Embeddings(sc, "umap"),
clusterLabels = sc$Cluster,
start.clus = c("1"),#起始和终点均可选
stretch = 0)
#line图
col1 <- colorRampPalette(brewer.pal(8,'Accent'))(11)
plot(Embeddings(sc, "umap"),col = col1[sc$Cluster],pch=16,asp=1)
lines(SlingshotDataSet(sds), lwd=2, type = 'lineages', col = 'black')
#平滑曲线
lin1 <- getLineages(sds, clusterLabels = "Cluster",
start.clus = c("1"),reducedDim = "UMAP")
crv1 <- getCurves(lin1)
plot(Embeddings(sc, "umap"),col = col1[sc$Cluster],pch=16,asp=1)
lines(SlingshotDataSet(crv1), lwd = 3, col = 'black')
首先还是先运行slingshot,跟之前不同的是,这里建议使用Embeddings提取降维后的坐标信息,而不是sc直接转化为SingleCellExperiment,得到如下图。
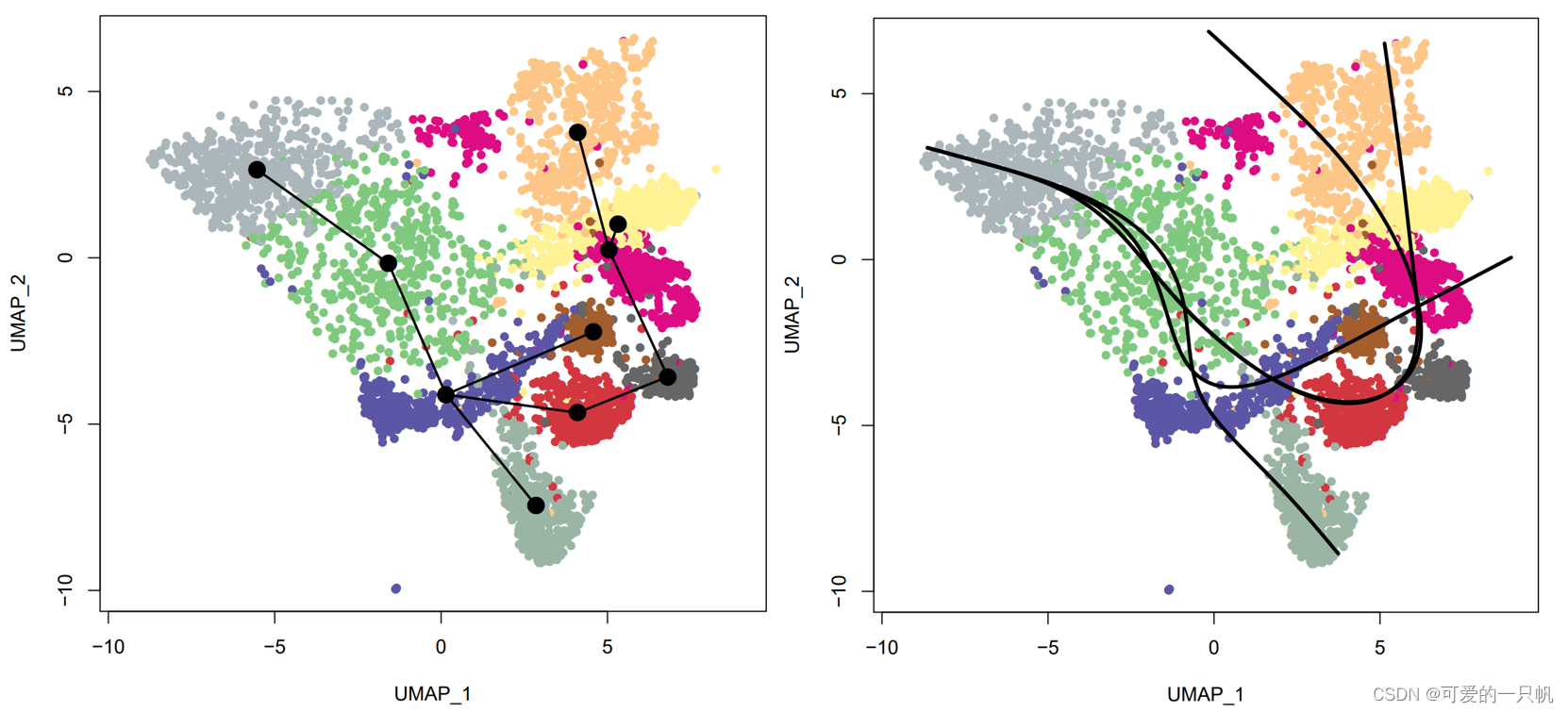
2.查看单个轨迹
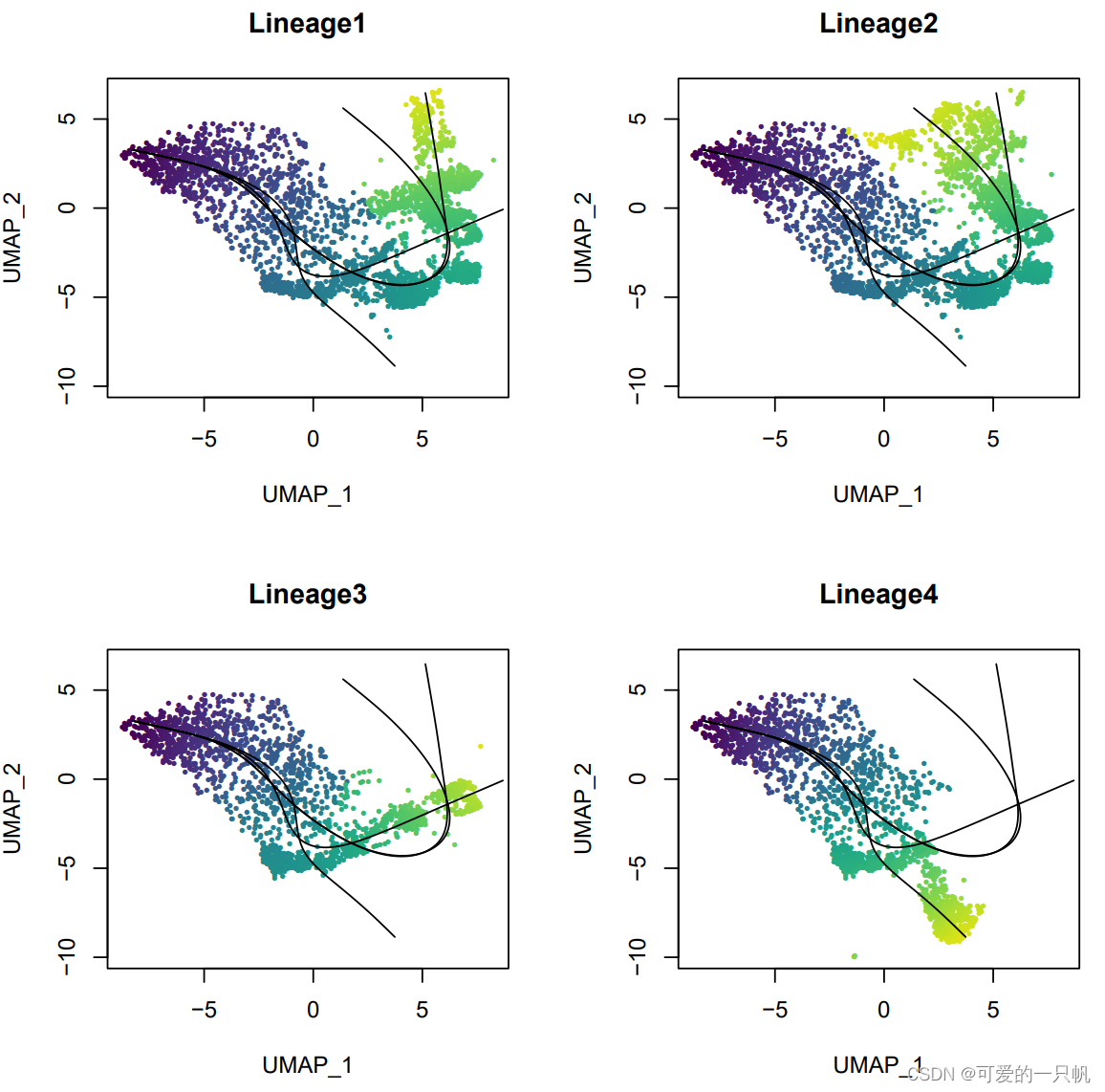
单个轨迹在sds文件的metadata里,可以看到有4条轨迹,根据拟时序顺序排列好的。
sds@metadata[["lineages"]]
# $Lineage1
# [1] "1" "0" "5" "7" "9" "6" "3"
# $Lineage2
# [1] "1" "0" "5" "7" "9" "6" "2"
# $Lineage3
# [1] "1" "0" "5" "8"
# $Lineage4
# [1] "1" "0" "5" "4"
3.提取单个轨迹作图
可以看到每个轨迹有哪些cluster,按照拟时序顺序由深到浅。
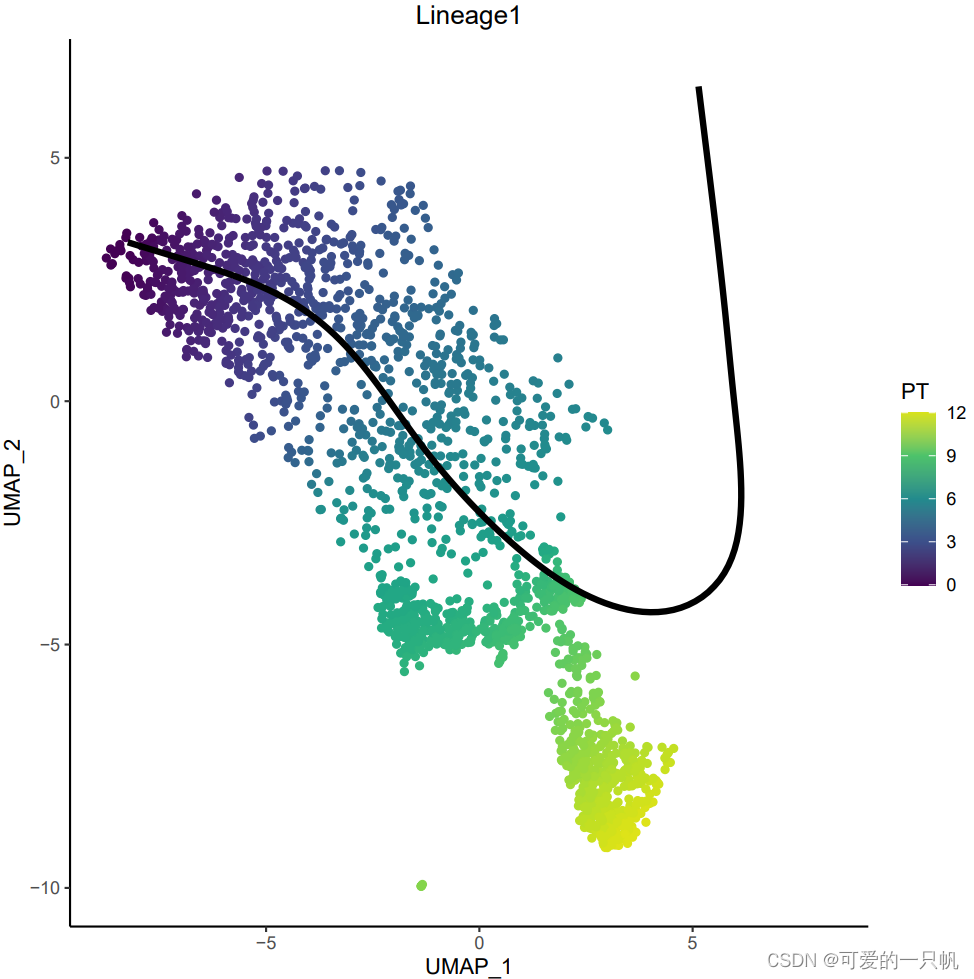
也可以提取slingPseudotime,用ggplot2作图,看得更明显一点。





















 855
855











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








