
网络图是生物信息学中常用的显示不同节点之间关联方向与关联程度的可视化方法。在富集分析中,网络图常被用于表示功能与估计到该功能的基因的联系。在蛋白质互作用网络中,网络图常用于表示编码基因之间的互作用类型与作用强度,基于这些信息,还可以通过某一节点与其他节点的连接数量来判断该节点在整个网络中的贡献度(degree)。绘制网络图常使用cystoscape软件,通过输入符合规范的数据,调整合适的参数,就可以得到一张包含多样化信息的网络。本文将从结果解读开始,先介绍网络图中常可以展示的信息类型,再介绍如何准备数据和调整参数。
1. 结果解读

这张图中包含的信息有:
1. 12个节点的对应关系
2. 不同节点有不同的颜色,代表所属组别,是定性属性
3. 不同的节点有不同的大小,代表某种定量属性
4. 不同连接线有不同的颜色,代表对应的互作用关系的作用类型,是定性属性
5. 不同连接线有不同的宽度,代表对应的互作用关系的作用强度,是定量属性
2. 数据准备

2.1 必需元素
如前所述,网络图是表示节点及其连接关系的图,因此,节点和对应关系是网络图的必需元素,也就是表格中的A列和B列。可以看到,有节点A1-A11均与节点B存在关联,反过来,节点B和节点A1-A11存在一对多的关系。
2.2 节点连接关系
既然节点之间存在连接关系,那么如何定义这种关系呢?在这里,我们使用了两个信息来描述,即C列的作用强度和D列的作用方向。作用强度通常可以由数据库给出,作用方向是使用-1表示负相关,1表示正相关。这样,我们就不仅知道两个节点有关联,还可以定量和定性的描述这种关联。
2.3 节点属性
在实际应用中,不同的节点具有不同的属性。比如用于描述miRNA-mRNA调节关系的网络中,每一个节点将会属于miRNA或者mRNA,那么我们就可以再添加一列信息用于描述这种属性。在本文中,我们假设A1-A11和B表示各不相同的12个基因,它们对于某一特定疾病具有不同的影响,如果增加患病风险,我们使用一个正值表示,数值越大,则表示增加的风险越多(risk factor),反之亦然。在表中,E列和G列分别定量地表示Gene1(A1-A11)与Gene2(B)对疾病的影响强度和方向。F和H则定性的表示这种方向。
3. 参数调整
3.1 输入准备好的表格

给不同的列定义作用类别
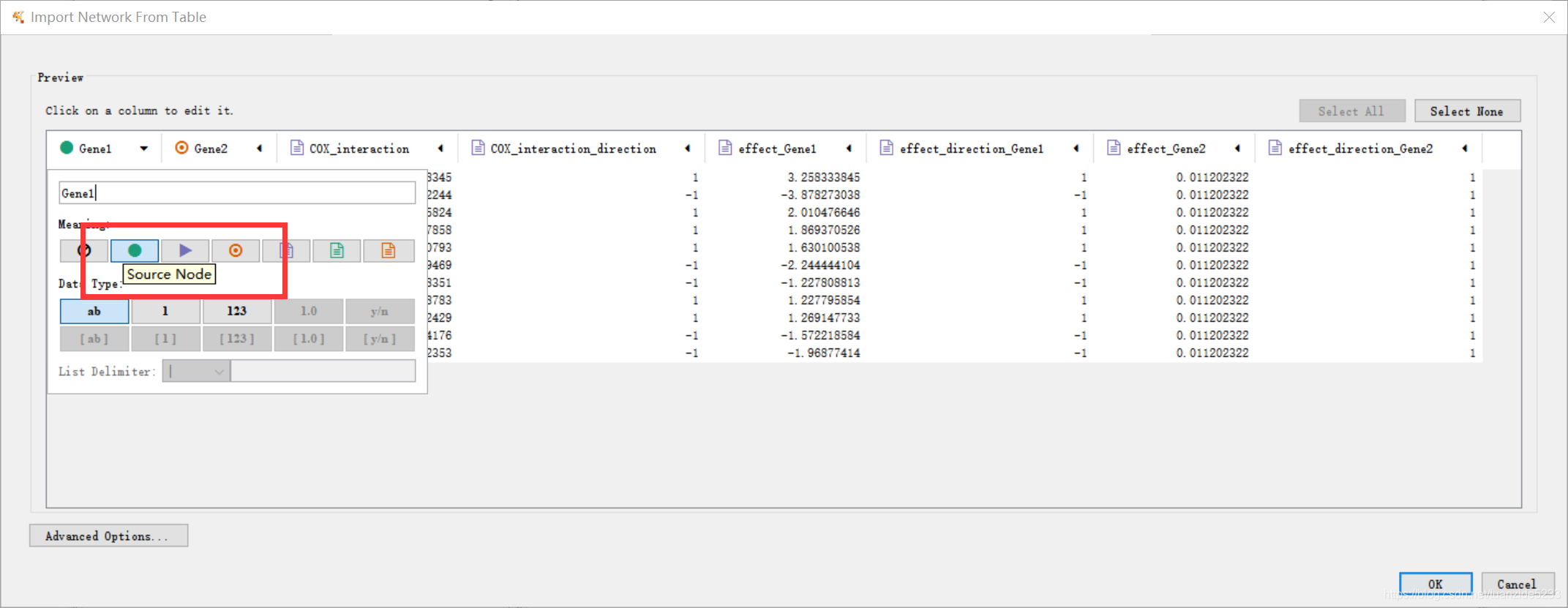
Gene1列定义为source node,表示作为一个出发点

Gene2列定义为target node,表示作为一个目标终点

cystoscape中的连接使用edge描述,因此edge attribute表示连接属性。说明这一列的数据都是用来描述前面两个对应的节点的连接属性的,即包含了连接属性的定量或定性信息。

source node attribute表示出发点属性。说明这一列的数据都是用来描述source node列的节点的节点属性,即包含了节点本身性质的一些信息,比如对疾病影响强度与方向。

target node attribute表示目标节点点属性。说明这一列的数据都是用来描述target node列的节点的节点属性,即包含了节点本身性质的一些信息,比如对疾病影响强度与方向。
之后就得到了最原始的一张网络图

3.2 在网络图中添加输入的参数信息
可以非常明显的看到这种图除了比较丑之外,还丢失了很多信息。事实上,它支包含了节点之间的对应信息,而节点属性和连接属性均没有体现在图上。下面








 本文详细介绍如何使用Cystoscape软件绘制生物信息学中的网络图,包括理解网络图展示的信息类型、数据准备步骤及参数调整技巧,帮助读者掌握绘制包含多样化信息的高质量网络图的方法。
本文详细介绍如何使用Cystoscape软件绘制生物信息学中的网络图,包括理解网络图展示的信息类型、数据准备步骤及参数调整技巧,帮助读者掌握绘制包含多样化信息的高质量网络图的方法。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 2329
2329

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








