代谢物组数据分析 基础流程Metabolomics analysis - 知乎
https://github.com/dgrapov/TeachingDemos/wiki/Partial-Least-Squares https://github.com/dgrapov/TeachingDemos/wiki/Partial-Least-Squares 单组学的多变量分析|1.PCA和PLS-DA
https://github.com/dgrapov/TeachingDemos/wiki/Partial-Least-Squares 单组学的多变量分析|1.PCA和PLS-DA
视频实操SCI作图课(3):OPLS-DA分析,组间差异的挖掘神器本次给大家介绍的是迈维云2.0的第三个高频小工具:OPLS-DA分析。 https://mp.weixin.qq.com/s?__biz=MzU2MzMzOTk1Mg==&mid=2247499171&idx=1&sn=305b1e43cc99438a63b76db9a9773c79&chksm=fc590a7ccb2e836a32592e9a3149e1ebfb040f3f6f43ea2d5fac826434e68b3c6d4fc26f1039&scene=178&cur_album_id=1762718371294330897#rd 代谢产物差异分析 | 生信菜鸟团
https://mp.weixin.qq.com/s?__biz=MzU2MzMzOTk1Mg==&mid=2247499171&idx=1&sn=305b1e43cc99438a63b76db9a9773c79&chksm=fc590a7ccb2e836a32592e9a3149e1ebfb040f3f6f43ea2d5fac826434e68b3c6d4fc26f1039&scene=178&cur_album_id=1762718371294330897#rd 代谢产物差异分析 | 生信菜鸟团  http://www.bio-info-trainee.com/8512.html
http://www.bio-info-trainee.com/8512.html
ropls: PCA, PLS(-DA) and OPLS(-DA) for multivariate analysis and feature selection of omics data https://www.bioconductor.org/packages/release/bioc/vignettes/ropls/inst/doc/ropls-vignette.html#the-ropls-package强烈推荐 : OPLS-DA在R语言中的实现 | 小蓝哥的知识荒原主成分分析(Principal Component Analysis,PCA)是一种无监督降维方法,能够有效对高维数据进行处理。但PCA对相关性较小的变量不敏感,而PLS-DA(Partial Least Squares-Discrimin
https://www.bioconductor.org/packages/release/bioc/vignettes/ropls/inst/doc/ropls-vignette.html#the-ropls-package强烈推荐 : OPLS-DA在R语言中的实现 | 小蓝哥的知识荒原主成分分析(Principal Component Analysis,PCA)是一种无监督降维方法,能够有效对高维数据进行处理。但PCA对相关性较小的变量不敏感,而PLS-DA(Partial Least Squares-Discrimin https://www.web4xiang.com/blog/article/3f4f7ea1/
https://www.web4xiang.com/blog/article/3f4f7ea1/
OPLS-DA法:代谢组学数据分析及可视化教程(Tutorial) - 知乎
OPLS-DA在R语言中的实现 | 小蓝哥的知识荒原主成分分析(Principal Component Analysis,PCA)是一种无监督降维方法,能够有效对高维数据进行处理。但PCA对相关性较小的变量不敏感,而PLS-DA(Partial Least Squares-Discrimin https://www.web4xiang.com/blog/article/3f4f7ea1/GitHub - dgrapov/TeachingDemos: Bioinformatics related demos and tutorials using the R programming language
https://www.web4xiang.com/blog/article/3f4f7ea1/GitHub - dgrapov/TeachingDemos: Bioinformatics related demos and tutorials using the R programming language
Orthogonal Partial Least Squares (OPLS) in R | R-bloggers
主成分分析(PCA)及其可视化的基础指南 https://mp.weixin.qq.com/s?__biz=MzIxMDExNDE0OQ==&mid=2247485703&idx=1&sn=6dd1718b68f635e05a9a559e12d18b7b&chksm=9768ca0da01f431bda6a6439aeddc85a4b7bec3dca439cef62db4aadfb7673c4b6a1be061d61&scene=21#wechat_redirect简便上手 | 利用Excel和小工具完成FC/p/VIP值计算 - 知乎
https://mp.weixin.qq.com/s?__biz=MzIxMDExNDE0OQ==&mid=2247485703&idx=1&sn=6dd1718b68f635e05a9a559e12d18b7b&chksm=9768ca0da01f431bda6a6439aeddc85a4b7bec3dca439cef62db4aadfb7673c4b6a1be061d61&scene=21#wechat_redirect简便上手 | 利用Excel和小工具完成FC/p/VIP值计算 - 知乎

主成分分析(Principal Component Analysis,PCA)是一种无监督降维方法,能够有效对高维数据进行处理。但PCA对相关性较小的变量不敏感,而PLS-DA(Partial Least Squares-Discriminant Analysis,偏最小二乘判别分析)能够有效解决这个问题。而OPLS-DA(正交偏最小二乘判别分析)结合了正交信号和PLS-DA来筛选差异变量
OPLS-DA:正交篇最小二乘法判别分析
原理:
OPLS-DA不同于PCA,它是一种有监督的判别分析统计方法。运用偏最小二乘回归建立代谢物表达量与样本类别之间的关系模型,来实现对样本类别的预测
OPLS-DA需要样本变量矩阵和样本分类矩阵两个文件确立样本关系,如下所示:

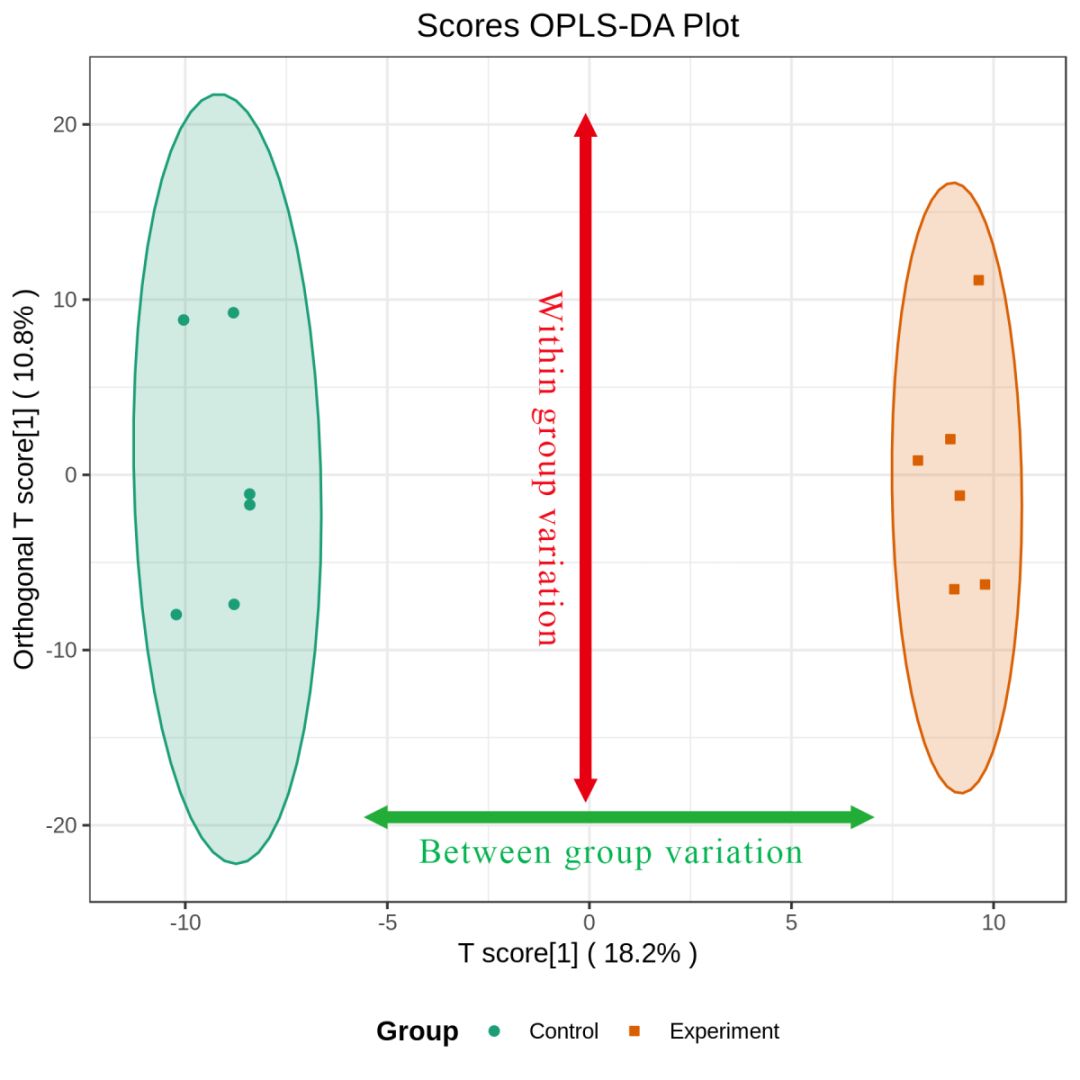
图1
结果图1:OPLS-DA得分图:
横坐标表示OSC过程中的主要成分的得分值( Tp ) , 所以从横坐标的方向可以看到组间的差异;
纵坐标表示OSC过程中的正交成分的得分值(TO) ,所以从纵坐标上看出组内的差异(组内样本间的差异)。

图2
结果图2:OPLS-DA的S-plot图
S-plot 图的横坐标表示主成份与代谢物的协相关系数,纵坐标表示主成份与代谢物的相关系数。
S-plot 图一般用来挑选与OSC过程中主要成分的相关性比较强的代谢物,从另一方面同时也可以挑选与Y相关性强的代谢物。越靠近两个角的代谢物重要度越强。红色的点表明这些代谢物的VIP值大于等于1 , 绿色的点表示这些代谢物的VIP值小于等于1。

图3
结果图3:OPLS-DA的模型验证 permutation Test图
模型验证 permutation Test 图的横坐标表示模型的准确率,纵坐标表示200次 permutation Test 中200个模型的准确率的频数,箭头表示本OPLS-DA模型准确率所在的位置。
其中R2X和R2Y分别表示所建模型对X和Y矩阵的解释率, Q2表示模型的预测能力,理论上R2、Q2数值越接近1说明模型越好,越低说明模型的拟合准确性越差。
通常情况下 , R2、Q2高于0.5较好,高于0.4即可接受。
从图中可以看出Q2为0.994 , R2Y为1 , R2X为0.685 , Q2和R2Y的P值均为0.005 ,说明 permutation Test 中只有1个随机分组模型结果优于本OPLS-DA模型,一般情况下P<0.05时模型最佳。
代谢组 转录组和蛋白组分别使用什么差异分析方法?为什么不可以使用相同的方法?
ChatGPT
代谢组学、转录组学和蛋白组学是三个不同层面的研究,分别关注代谢物、基因表达和蛋白质表达的变化。由于它们具有不同的数据特点和研究目的,所以需要使用不同的差异分析方法。
在代谢组学中,常用的差异分析方法包括t检验、方差分析(ANOVA)、多重比较校正方法(如Bonferroni校正、Benjamini-Hochberg校正)等。这些方法适用于比较不同样本组之间代谢物的相对丰度或浓度差异。代谢组学数据通常是高维度、非正态分布的,因此需要选择适合的统计方法进行分析。
转录组学中常用的差异分析方法包括DESeq、edgeR等。这些方法主要用于比较基因在不同样本组之间的表达水平差异。转录组学数据通常是基因表达水平的定量数据,因此需要使用适合的统计方法来确定差异表达基因。
蛋白组学的差异分析方法则有一些不同的特点。蛋白组学数据通常是通过质谱或免疫测定等技术获得的,可以获得蛋白质在样本中的相对丰度或表达水平。蛋白组学中的差异分析方法包括t检验、ANOVA、线性模型等,也可以使用多重比较校正方法进行统计显著性校正。
虽然转录组、代谢组和蛋白组都关注生物体内不同分子层面的变化,但它们之间有一些重要的区别。转录组研究基因表达水平的差异,代谢组研究代谢物的相对丰度差异,蛋白组研究蛋白质表达水平的差异。由于这些数据具有不同的特点和性质,不能简单地使用相同的差异分析方法。需要根据数据类型和研究目的选择适当的方法来进行差异分析,以确保结果的准确性和可解释性。







 https://zhuanlan.zhihu.com/p/79373522
https://zhuanlan.zhihu.com/p/79373522

















 3746
3746

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










