· 爱基百客云平台小工具使用
1.1 爱基百客云平台之GSEA分析
1.2 GSEA富集分析原理图
1.3 GSEA富集分析过程
1.4 参数设置
1.5 任务查看
1.6 结果
1.6.1 富集结果表格
1.6.2 富集可视化图解读
01
爱基百客云平台小工具使用
首先,打开爱基百客官网:http://www.igenebook.com;点击菜单栏最右侧“云平台”按钮。

弹出云平台界面(下图),输入账号、密码和验证码方可登录;进入云平台,可以轻松实现多种组学数据的分析和可视化,实现真正的“零代码、无门槛、操作简单”!

登陆后,如下图,我们进入到小工具专栏。当前云平台已上线了32款小工具供大家使用,包括基础绘图,高级绘图,差异检验,聚类分析,序列处理等子模块,本着用户至上的理念,平台小工具将会持续更新维护,积极接受用户的反馈和意见。

1.1
爱基百客云平台之GSEA分析
上期我们讲到了富集分析。那本期我们就接着来聊一聊富集分析里面的新星:GSEA(Gene Set Enrichment Analysis)富集分析。
常规的富集分析,通过log2fc筛选差异基因,将筛选差异基因的标准聚集在单个基因的差异表达上,可能会错过真正具有生物学含义的基因群差异。于是我们可以选择进行GSEA富集分析。GSEA集富集分析有以下几个优点:
1. 全局视角:GSEA不仅关注单一基因的差异,而是关注一组基因集合的整体行为,这使得它能揭示生物学过程的整体模式,而不仅仅是局部变化。
2. 生物意义:通过预定义的基因集,GSEA提供了生物学意义的验证,比如特定通路的活动,这有助于理解基因表达变化背后的生物学背景。
3. 适应复杂数据:GSEA可以处理复杂的数据分布,如连续的基因表达数据,而不仅仅是二元的上调或下调状态。
1.2
GSEA富集分析原理图

1.3
GSEA富集分析过程
1. 计算富集分数(ES) 富集分数:S 反应基因集(比如某个通路内的基因集)成员 s 在排序基因集 L(比如根据 logFC 排序的差异基因集,默认降序,所以上调基因在顶端)的两端富集的程度。富集得分 ES 最后定义为最大的peak值。正值ES表示基因集 S 在基因集 L 的顶部富集,负值ES表示基因集 S 在基因集 L 的底部富集。
2. 估计富集分数的显著性水平:因为每个基因集的大小不同,而 ES 又和基因集大小有关,因此需要对其进行 normalization。为了检验每个基因集的 NES 是否显著,将总基因集 L 随机打乱排列一定次数,每次都计算每个基因集的 NES(ES),得到每个基因集的 NES 在随机排序情况下的理论分布,从而计算其 p 值。
3. 矫正多重假设检验 FDR 则是对 p 值进行 BH 校正之后的 p 值。
爱基百客云平台提供了GSEA分析小工具:http://124.71.149.47:5000/smalltools/detail?id=1638074560383299585。如果您在我司做过项目,直接输入差异分析表格和基因组名称即可一键式完成富集分析。
下面我们进行GSEA富集分析实操练习。
首先点击小工具GSEA富集分析。

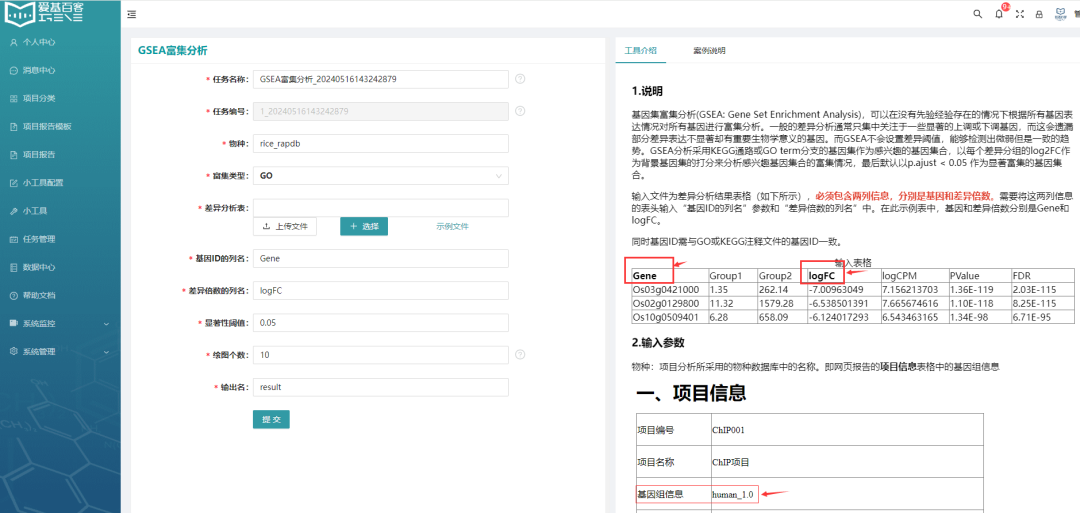
右侧的工具介绍和常见问题对富集分析小工具的主要用途,使用方法以及结果解读做了详细的说明。左侧是必要的输入文件和参数选项。任务名称和任务编号系统会自动生成。后面可用于记录查看具体的任务。小工具提供了示例文件给用户做测试分析。同时,该页面还提供了一些常用参数调节选项。您也可进行自定义,后面将详细介绍。输出名自定义,默认Enrich。
1.4
参数设置
输入文件:支持txt(制表符分隔)文本文件,以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。
差异基因表格必须包含Gene列和logFC列。如下表所示:

如果文件已经上传过,您可以直接点击选择按钮找到需要的文件勾选确定,无须再次上传。
物种选择项目中所使用的基因组名称;富集类型支持GO和KEGG;基因列和logFC列的名字根据输入表格的信息填写;显著性阈值您可根据实际情况进行筛选,默认是p.adjust < 0.05;绘图个数默认选取排名靠前的10个通路绘制,你可自行选择需要展示前多少个;输出名自定义即可。
填写好上述所有的参数后,点击提交即可。
1.5
任务查看
您可在任务管理栏中查看任务的运行情况和结果。默认情况下新任务将会在最上方展示。也可通过任务名、任务编码、日期点击查询,找到需要的任务。如下图:当状态成功时,表示任务成功结束。

点击下载按钮可直接打包下载全部结果。点击查看按钮可在线查看结果。
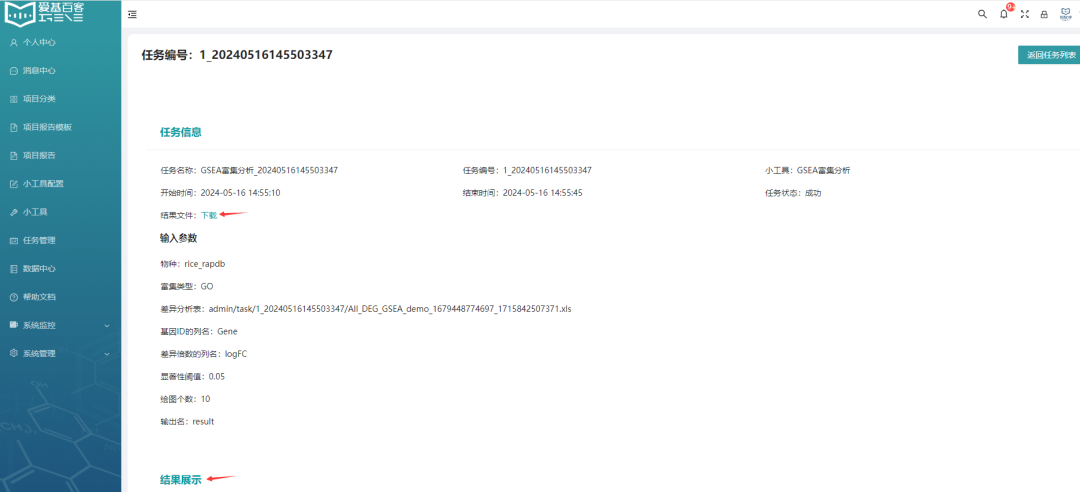
您可在在线查看结果后选择是否下载保存。另外如果结果较多时,结果展示区还可通过点击左侧或右侧的箭头进行图片切换。您可挑选自己感兴趣的结果图片进行下载保存。

1.6
结果
输出结果包含GSEA富集总表和若干通路的GSEA富集图。
1.6.1
富集结果表格

-
ID:通路名
-
Description:通路描述信息
-
setSize:该通路中包含表达数据集文中的基因数目(经过条件筛选后的值)
-
enrichmentScore:富集分数
-
NES:标准化后的富集分数
-
pvalue:是对 ES 的统计学分析,用来表征富集结果的可信度
-
p.adjust:是多重假设检验校正之后的 p-value
-
qvalues:是多重假设检验(FDR法)校正之后的 p-value,即对NES可能存在的假阳性结果的概率估计。GSEA 对显著性的定义为 p-value<5%,FDR q-val<25%
-
rank:当 ES 最大时,对应基因所在排序好的基因列表中所处的位置
-
leading_edge:tags 表示核心基因占该通路基因集的百分比;list 表示核心基因占所有基因的百分比;signal,将前 2 项统计值结合在一起计算出的富集信号强度
-
core_enrichment:核心富集基因集
1.6.2
富集可视化图解读

富集图一共分为上中下 3 部分。如A_VS_B差异分析:
-
第一部分 ES 折线图:显示了当分析沿着排序基因集按排序计算时,ES 值在计算到每个位置时的展示。最高峰处的ES得分 (垂直距离 0.0 最远)便是基因集的 ES 值。
-
第二部分 hits 图,俗称条形码图,用线条或者 hit 标记了通路基因集(基因组所有)中成员出现在基因排序列表中的位置。如果基因集里的基因 集中在所有基因的前部分,就是在A组里面富集,如果集中在后面部分,就是在B组里面富集。leading edge subset 就是(0,0)到绿色曲线峰值 ES 出现对应的这部分基因(x轴0到虚线那部分)。所谓 Leading-edge subset,就是对富集得分贡献最大的基因成员。如果ES得分都是正值(如上图所示),那么Leading-edge subset就在峰值ES的左侧,反之则在右侧(底部富集 = A/B 下调表达 = B 组高表达)。那么根据本图我们很容易看出,该通路在 A 组高表达。
-
第三部分是排序后所有基因 rank 值的分布,热图红色部分对应的基因在 A 组高表达,蓝色部分对应的基因在 B 组高表达,每个基因对应的信噪比(Signal2noise,前面选择的排序值计算方式)以灰色面积图展示。
如果常规富集没有拿到预期的结果,大家完全可以拿GSEA再分析一次,说不定有惊喜哦。
























 4562
4562

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








