富集分析是生物医学论文中非常常见的一类分析,例如GO富集分析,Pathway富集分析等。其结果一般包括以下几个要素:1,名字(GO term或者KEGG description);2,该名字中包含的基因;3,该名字中包含的基因的P值等。常见的绘图包括:气泡图,条形图。今天我们利用GOplot R包,来绘制一个弦图(chord plot)。
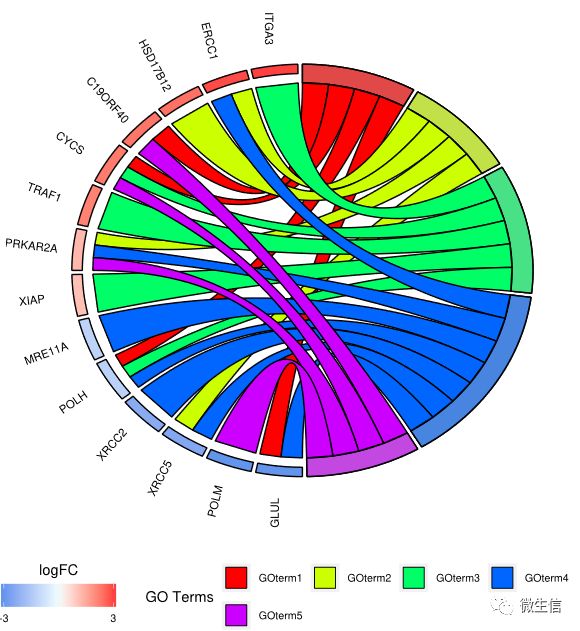
弦图简介:下面这个圆形的、错综复杂的图就是弦图,属于圈图的一种,在高大上的生物医学类文章中会经常见到。弦图可以展示多个对象间的关系。例如这里就展示了5个不同GO term(右侧)里包含的基因,及它们的倍数变化(左侧)。本质上讲,弦图是一种网络图,想象一下,如果把外侧的矩形都换成点,中间的带换成连线,就是一个圆形布局的网络图。
1,打开绘图页面
首先,使用浏览器(推荐chrome或者edge)打开弦图绘制页面。左侧为常见作图导航,中间为数据输入框和可选参数,右侧为描述和结果示例。也可以在主页搜索框中搜索chord,找到绘图页面。
https://www.bioinformatics.com.cn/plot_basic_GOplot_chord_plot_085
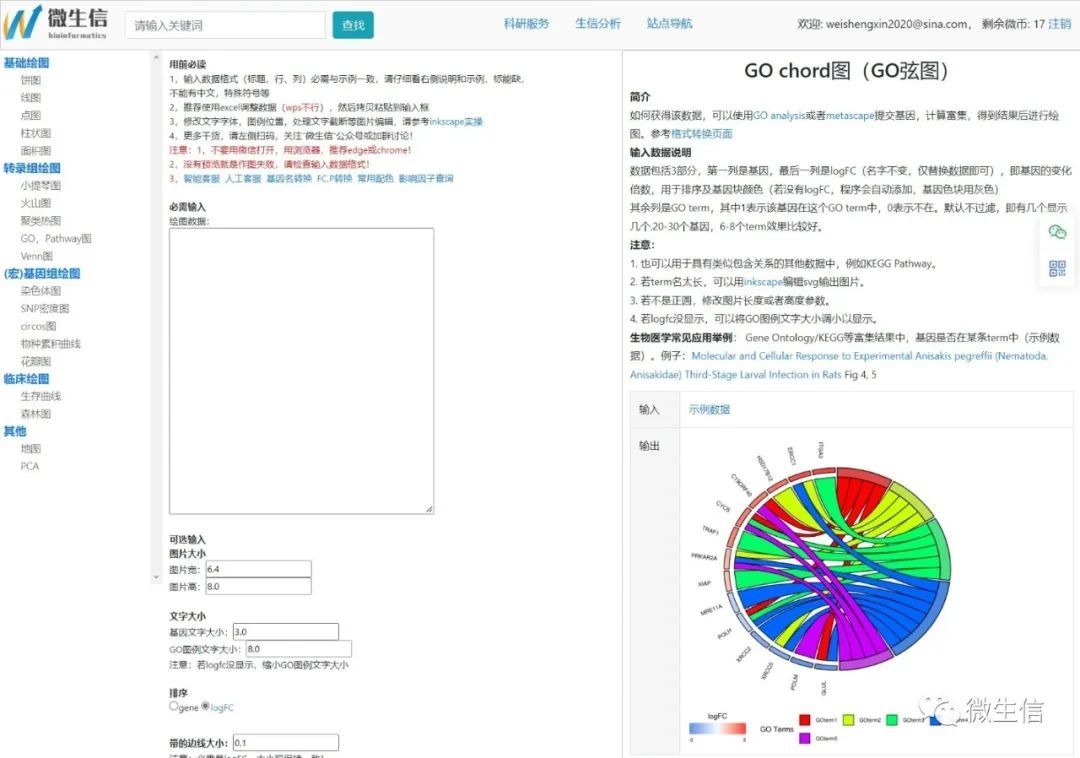
图1.可视化绘图页面
2,示例数据
点击右侧“示例数据”链接下载excel格式的示例数据。
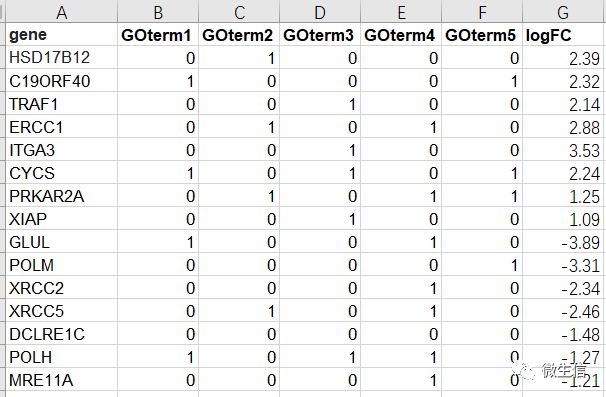
图2. 输入数据示例
示例数据(仅供参考)包括3块:
第1块(第一列)是基因名
第2块(第二列到倒数第二列)是基因在每个GO term中的从属关系,0表示不再这个GO term里,1表示在这个GO term里。
第3块(最后一列)是基因对应的倍数变化 (可选,你的数据中没有这个信息的话,就不要这一列),名字必需是logFC(大小写敏感)。
注意:需要参考示例数据,将自己的数据在excel中整理成示例数据的样式。
我们还提供了格式转换页面,可以将任何富集结果转成GO chord输入形式
https://www.bioinformatics.com.cn/GO_chord_data_format_convert_t002
输入是metascape的富集结果,第一列是名字,第二列是基因,以|分割。拷贝你的富集结果,然后输入分隔符就可以转化成绘图数据了(见图2示例)
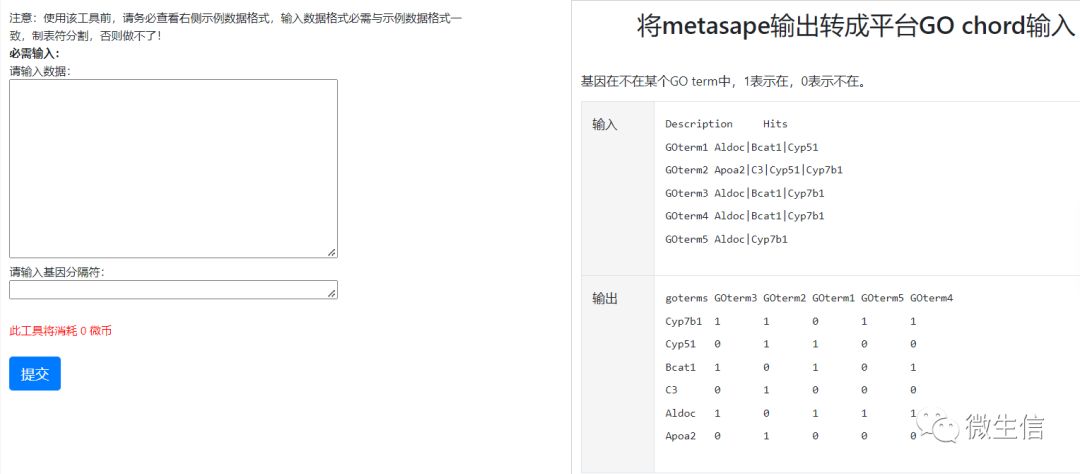
图3. 富集结果转化辅助页面
3,粘贴示例数据
直接复制示例数据中的A-G列数据,然后粘贴到输入框。
注意:不是拷贝excel文件,是拷贝excel文件里边的数据。另外粘贴到输入框后,格式乱了没关系,只要在excel中是整齐的就行。并且数据矩阵中不能有空的单元格,中文字符等。
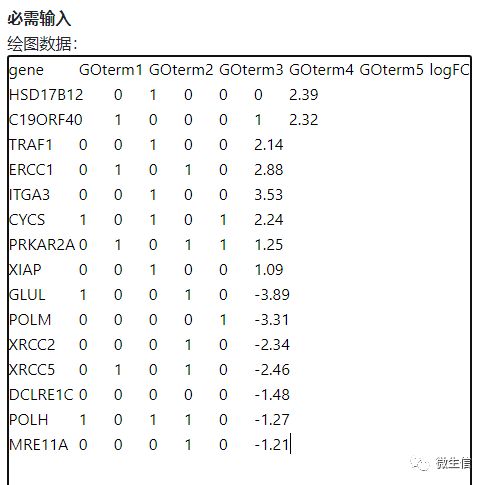
图4. 必需输入
4,修改参数,并提交
我们设置了图片尺寸,文字大小,排序,字体等参数,基本能满足日常绘图使用。如需更高级的定制,请联系我们。
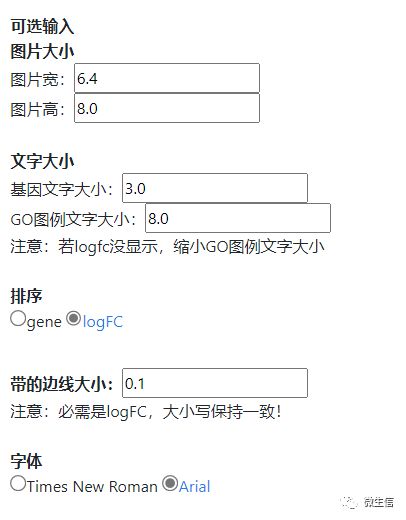
图5. 节点、文字、颜色等可调参数
5,提交出图
粘贴好输入数据,调整好参数(或者全部默认)后,点击提交按钮,约5秒后,会在页面右侧出现预览图。我们提供了4种图片格式供下载使用,两种矢量图(pdf,svg)和两种标量图(600 dpi tiff和300 dpi png)。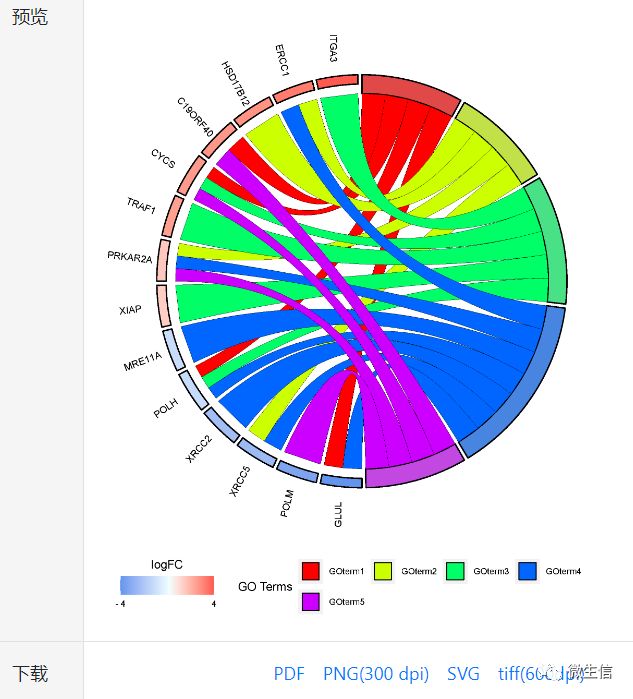
图6.预览与下载
点评:
1,由于该图利用弦来表示关联,因此条目不能过多,推荐6个以内的GO或者KEGG条目,基因50个以内。
2,由于GO term有时候非常长,导致该图经常有截断,因此可以先利用缩写来代替,或者先设置非常小的字体,然后使用AI或者inkscape调整文字位置及大小。
3,由于GO term跟圈图比例不是固定的,因此出的图并非完全正圆,若有强迫症,可以使用windows自带的“截图和草图”-“量角器”进行确证并调整图片长宽。
没有预览就是没有出图,这时请参考示例数据,检查自己输入数据的格式。
遇到文字截断,需要修改字体、调整字体大小等,使用scape软件。
微生信助力发文章,谷歌引用590+,知网引用450+











 本文介绍如何使用在线工具绘制生物医学研究中常用的GO富集分析弦图,包括准备工作、示例数据下载、数据格式调整、参数设置及最终图表导出。
本文介绍如何使用在线工具绘制生物医学研究中常用的GO富集分析弦图,包括准备工作、示例数据下载、数据格式调整、参数设置及最终图表导出。
















 308
308

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








