全网最全 KEGG 注释结果绘图,直击 SCI 绘图标注,关注我,您最好的选择!
前言
1. KEGG 原理
KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库是系统地分析基因功能、链接基因组信息和功能信息的数据库,包括代谢通路(pathway)数据库、分层分类数据库、基因数据库、基因组数据库等。KEGG的pathway数据库是应用最广泛的代谢通路公共数据库。
富集的含义:
这里pathway富集的含义与GO富集的含义相同,也是表示差异基因中注释到某个代谢通路的基因数目在所有差异基因中的比例显著大于背景基因中注释到某个代谢通路的基因数目在所有背景基因中的比例。因此,做pathway富集分析,也是涉及到前景基因和背景基因。前景基因就是你关注的要重点研究的基因集,背景基因就是所有的基因集。
**
富集显著性(P value)的计算:**
计算方法和公式与GO富集分析一样,也是利用超几何检验计算:

其中,N为所有基因中具有Pathway注释的基因数目;n为N中差异表达基因的数目;M为所有基因中注释为某特定Pathway的基因数目;m为注释为某特定Pathway的差异表达基因数目。
计算得到的P value会进一步经过多重检验校正,得到corrected-pvalue(也就是Q value)。通常我们会以Q value≤0.05为阈值,满足此条件的pathway定义为在差异表达基因中显著富集的pathway。
2. 实例解析
1. 数据读取
数据的读取我们仍然使用的是 TCGA-COAD 的数据集,表达数据的读取以及临床信息分组的获得我们上期已经提过,我们使用的是edgeR 软件包计算出来的差异表达结果,提取上调基因 2832 的 ENSEMBL 号,
###########基因列表
DEG=read.table("DEG-resdata.xls",sep="\t",check.names=F,header = T)
geneList<-DEG[DEG$sig=="Up",]$Row.names
table(DEG$sig)
##
## Down Up
## 1296 2832
读取样本分组信息,41个正常组织,478个癌症组织,如下:
######样本分组信息
group<-read.table("DEG-group.xls",sep="\t",check.names=F,header = T)
table(group$Group)
##
## NT TP
## 41 478
2. KEGG 注释结果
首先我们同样需要安装软件包并加载,这里面主程序就是 clusterProfiler 软件包,如下:
########
if(!require(clusterProfiler)){
BiocManager::install("clusterProfiler")
}
if(!require(org.Hs.eg.db)){
BiocManager::install("org.Hs.eg.db")
}
if(!require(DOSE)){
BiocManager::install("DOSE")
}
if(!require(topGO)){
BiocManager::install("topGO")
}
if(!require(pathview)){
BiocManager::install("pathview")
}
if(!require(KEGG.db)){
BiocManager::install("KEGG.db")
}
library(org.Hs.eg.db)
library(clusterProfiler)
library(DOSE)
library(topGO)
library(pathview)
library(KEGG.db)
根据数据比对我们找到1726 个KEGG数据中有的基因,同时我们获得到一个基因的三种命名方式,即"ENTREZID", “ENSEMBL”, ‘SYMBOL’,如下:
####KEGG
eg <- bitr(geneList,
fromType="ENSEMBL",
toType=c("ENTREZID","ENSEMBL",'SYMBOL'),
OrgDb="org.Hs.eg.db")
head(eg)
## ENSEMBL ENTREZID SYMBOL
## 1 ENSG00000062038 1001 CDH3
## 2 ENSG00000175832 2118 ETV4
## 3 ENSG00000167767 144501 KRT80
## 4 ENSG00000164283 11082 ESM1
## 5 ENSG00000120254 25902 MTHFD1L
## 6 ENSG00000129474 84962 AJUBA
对获得的基因进行KEGG注释,这里是人的结直肠癌的数据,故 organism = ‘hsa’, 阈值分别为 pvalueCutoff = 0.05 和 qvalueCutoff = 0.05, 注释的时候我们可以选择参数 keyType 中4种不同的选择,包括"kegg", ‘ncbi-geneid’, ‘ncib-proteinid’ and ‘uniprot’,下面我们看到利用基因列表注释到6个pathway通路,如下:
# Run KEGG enrichment analysis
kegg <- enrichKEGG(eg$ENTREZID,
organism = 'hsa',
keyType = 'kegg',
pvalueCutoff = 0.05,
pAdjustMethod = 'BH',
minGSSize = 1,
maxGSSize = 500,
qvalueCutoff = 0.05,
use_internal_data = FALSE)
head(kegg)
## ID Description GeneRatio
## hsa05034 hsa05034 Alcoholism 38/384
## hsa05322 hsa05322 Systemic lupus erythematosus 31/384
## hsa04613 hsa04613 Neutrophil extracellular trap formation 33/384
## hsa04310 hsa04310 Wnt signaling pathway 25/384
## hsa04657 hsa04657 IL-17 signaling pathway 18/384
## hsa04060 hsa04060 Cytokine-cytokine receptor interaction 34/384
## BgRatio pvalue p.adjust qvalue
## hsa05034 187/8115 8.845476e-15 2.529806e-12 2.420867e-12
## hsa05322 136/8115 1.113336e-13 1.592070e-11 1.523512e-11
## hsa04613 190/8115 5.170512e-11 4.929222e-09 4.716958e-09
## hsa04310 167/8115 2.536698e-07 1.786110e-05 1.709196e-05
## hsa04657 94/8115 3.122570e-07 1.786110e-05 1.709196e-05
## hsa04060 295/8115 1.126797e-06 5.371067e-05 5.139777e-05
## geneID
## hsa05034 2906/2904/7054/8367/8356/8364/85235/128312/8360/8366/440689/8348/8332/8335/8340/1813/2786/8331/8354/8346/8344/2792/8338/317772/8343/8336/810/3013/8359/653604/8351/8968/8970/8350/8368/6531/8342/3018
## hsa05322 2904/8367/8356/8364/85235/128312/8360/8366/440689/8348/8332/8335/8340/8331/8354/8346/8344/8338/317772/8343/8336/3013/8359/653604/8351/8968/8970/8350/8368/8342/3018
## hsa04613 5582/8367/8356/8364/85235/128312/8360/8366/440689/8348/8332/8335/8340/366/8331/8354/8346/8344/8338/317772/8343/8336/2244/3013/8359/653604/8351/8968/8970/8350/8368/8342/3018
## hsa04310 7472/4316/85409/7473/147111/54894/8840/7477/51176/85407/27121/8313/8549/8061/6424/5582/7479/7481/59352/22943/11211/89780/11197/7476/343637
## hsa04657 225689/4314/2921/4312/2919/4322/8061/2920/3576/6374/1437/27189/6372/4586/3934/6278/3605/112744
## hsa04060 3624/51330/9518/3589/2921/2919/8744/51561/268/2920/655/3576/3552/6374/1437/5473/10913/1237/284340/3557/27189/6372/10344/5008/3625/4982/6373/338376/26525/4838/3623/56300/3605/112744
## Count
## hsa05034 38
## hsa05322 31
## hsa04613 33
## hsa04310 25
## hsa04657 18
## hsa04060 34
3. 结果展示
针对 KEGG 富集结果我们这里给出了多种展示方式,根据自己的需求以及文章的设计,选择适合自己的即可。
绘图过程中我们需要安装并加载几个软件,如下:
library(stringr)
library(cowplot)
library(ggplot2)
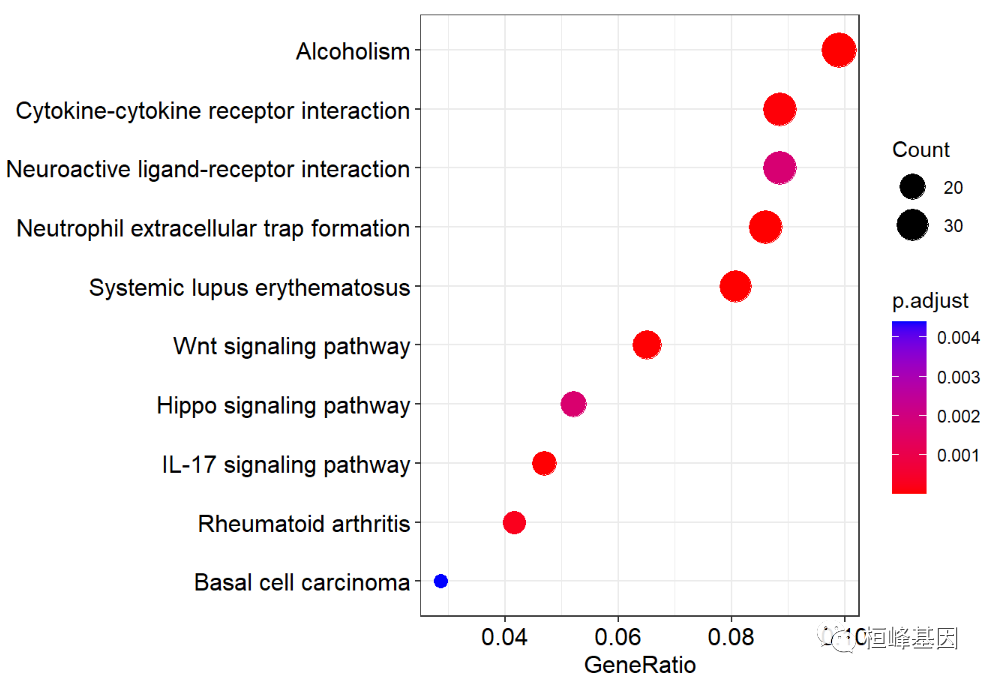
绘制气泡图,气泡图解读需要说明一下,GO富集程度通过Gene ratio、Pvalue和富集到此GO term上的基因个数来衡量。
-
横坐标是Gene ratio,数值越大表示富集程度越大。Count 位于该GO term下的差异表达基因数
-
纵坐标是富集程度较高的GO term(一般选取富集最显著的20条进行展示,不足20条则全部列出)。
-
Pvalue取值范围[0, 1],以颜色表示,越红表示Pvalue越小,说明富集越明显。
-
点的大小表示该term下差异基因的个数,点越大表示基因数越多。
dotplot(kegg,showCategory=10)

由于KEGG 的描述字体重合,故我们利用scale_y_discrete设置,避免字体彼此重合,如下:
dotplot(kegg,showCategory=10)+
scale_y_discrete(labels=function(x) stringr::str_wrap(x, width=60))
绘制柱状图,如下:
barplot(kegg,showCategory=10)+
scale_y_discrete(labels=function(x) stringr::str_wrap(x, width=60))
## Scale for 'y' is already present. Adding another scale for 'y', which
## will replace the existing scale.

绘制基因概念的网络图,Pathway 与差异基因关系网络图 Gene-Concept Network,对于基因和富集的Pathway 之间的对应关系进行展示说明:图中灰色的点代表基因,黄色的点代表富集到的Pathway ;如果一个基因位于一个Pathway 下,则将该基因与Pathway 连线;黄色节点的大小对应富集到的基因个数,top2富集到的Pathway ,如下:
cnetplot(kegg,showCategory=3,circular=TRUE,colorEdge=TRUE)

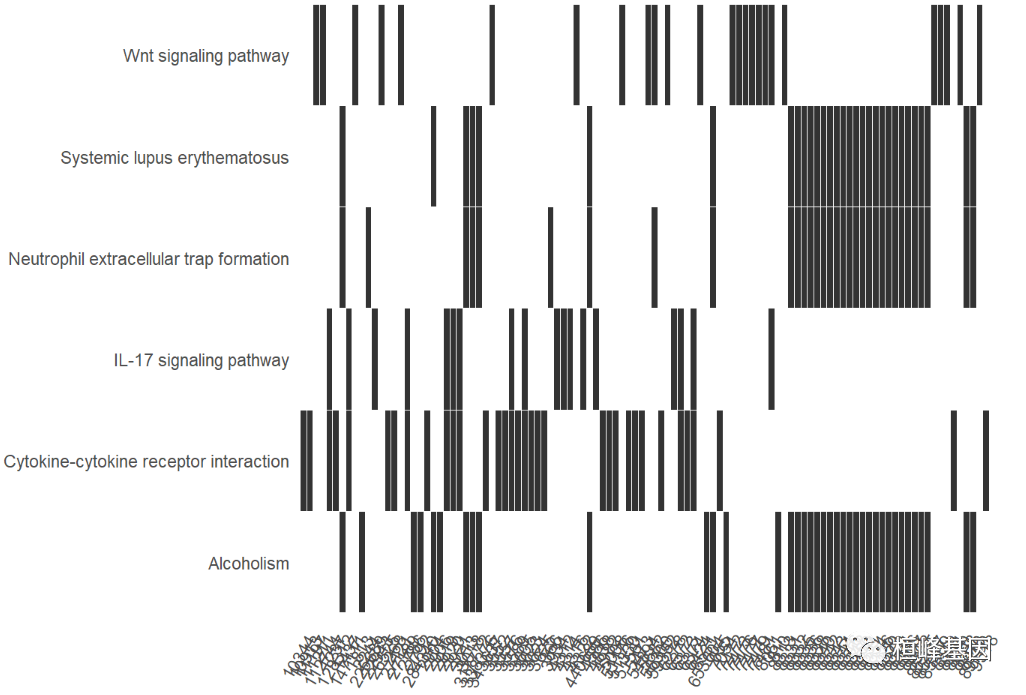
热图可以看到哪些基因富集到哪些 Pathway,如下:
heatplot(kegg,showCategory=6)+
scale_y_discrete(labels=function(x) stringr::str_wrap(x, width=60))
## Scale for 'y' is already present. Adding another scale for 'y', which
## will replace the existing scale.
软件安装并加载,如下:
if(!require(ggnewscale)){
install.packages("ggnewscale")
}
library(enrichplot)
library(ggnewscale)
对于富集到的pathways之间的基因重叠关系进行展示,如果两个pathway的差异基因存在重叠,说明这两个节点存在overlap关系,在图中用线条连接起来,每个节点是一个富集到的pathway, 默认画top30个富集到的pathways, 节点大小对应该pathway下富集到的差异基因个数,节点的颜色对应p.adjust的值,从小到大,对应蓝色到红色。用法如下:
ed = enrichDO(eg$ENTREZID, pvalueCutoff=0.05)
met <- pairwise_termsim(ed)
emapplot(met,showCategory = 15)

在pathway通路图上标记富集到的基因,代码如下 会给出一个url链接,示例:KEGG PATHWAY: Alcoholism - Homo sapiens (human)在浏览器中打开会看到如下所示的图片,如下:
browseKEGG(kegg, "hsa05034")

KEGG的富集分析与GO差不多,同样的软件包同样的函数,唯一不同的是最后一个可以看到pathway通路上的基因,非常方便!
关注公众号,每日更新,扫码进群交流不停歇,马上就出视频版,关注我,您最佳的选择!

桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
46篇原创内容
公众号
References:
-
Kanehisa M, Furumichi M, Tanabe M, Sato Y, Morishima K. KEGG: new perspectives on genomes, pathways, diseases and drugs. Nucleic Acids Res. 2017;45(D1):D353-D361. doi:10.1093/nar/gkw1092
-
Yu G, Wang L, Han Y and He Q*. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS: A Journal of Integrative Biology. 2012, 16(5):284-287.
























 5472
5472

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








