写在前面
今天周末,转眼10月份只剩一周。万万没想到,一个月下去,我还是花了不少时间在完善「GSAman」。至于为什么本来「两个小时」就干完的事情,可以干成「22天」?到底还是我对前面的版本,不太满意。当然,我想今天应该要告一段落,起码在新功能的特性的增加上,可以基本告一段落。暂时我没想到新的非常 exciting 或者用户也没提出让我觉得确实非常值得尽快更新的功能。于是,今天对 GSAman 已有的功能特性做一个汇总,同时也做一个 GSAman 相关推文目录贴,方便大伙了解。
主要特性汇总
1. 超快的装载速度
相比于 IGV 原版本,GSAman在装载基因结构注释信息文件时,采用完全索引方式,第一次装载后,后续任何一次打开 GSAman,都直接利用已有索引,速度极快
2. 更好看且直观的显示模式
使用橙色表示当前基因组正链上的转录本,使用蓝色表示当前基因组反链上的转录本,使用深灰色表示无链性的转录本。
针对 CDS,使用黑色框线标识,方便显示极小CDS;同时对外显子左右便捷使用红色线条标注,方便辨识极小内含子;此外,支持 1-bp 外显子,尽管这类可能性不大。

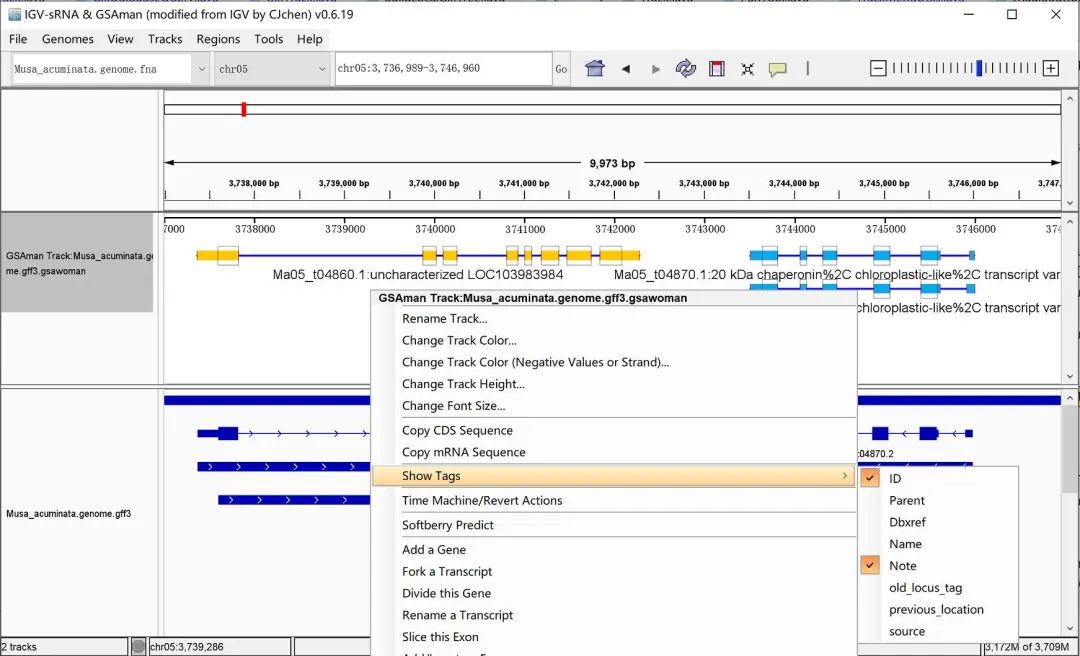
3. 支持丰富的信息展示
官方版本只能展示转录本ID或者基因ID等,但事实上,很多时候我们希望同时看到转录本ID和基因ID,更甚至各类丰富的信息,如基因功能注释信息,此处为「Note」标签。
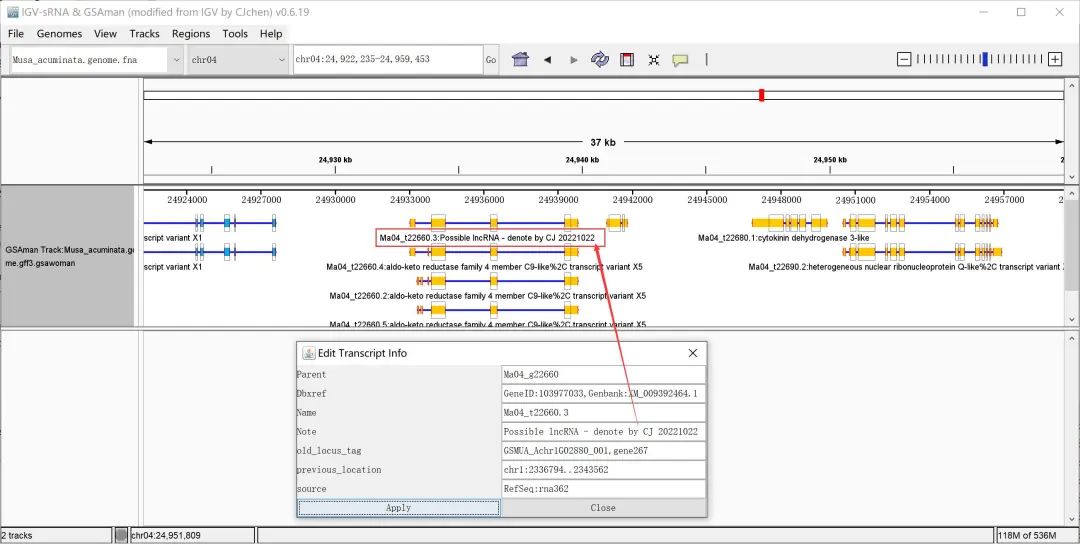
4. 支持实时编辑修改基因结构注释信息
通过鼠标右键点击转录本,在弹出菜单中点击「Edit Transcript Info.」即可修改转录本注释信息,比如增加一些个人对该转录本的评述信息。或者调整转录本归属的基因ID信息。
5. 直接支持本地BLAT检索
GSAman 安装器直接附带了 BLAT 可执行程序(支持Windows和MacOS)。相比于IGV原始版本多年未解决且可能未计划解决的基因组浏览器中序列查找痛点,GSAman增加了相应选项和功能。用户可在主菜单「View」->「Preference」->「Advanced」勾选「Local BLAT」选项,从而启用本地 BLAT 功能(注意:无需自行安装BLAT,GSAman已经全部准备好了)。









 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 2763
2763

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








