研究背景
随着科技的进步和对生物系统深入理解的不断深化,人们越来越关注生物系统中复杂的拓扑结构和其在信息处理中的重要作用。生物体内存在着诸如大脑神经网络和染色质组织中的高超结构,这些结构依赖于各种拓扑转换来动态调整节点之间的连接,从而实现功能上的适应性和效率。然而,如何在合成的分子尺度上构建能够模仿这些复杂拓扑转换并具有高度功能的人工结构,是一个迫切需要解决的挑战。
在现有的合成生物学领域中,DNA纳米技术的发展提供了一个强大的工具箱,能够精确地构建各种拓扑异构体纳米结构。DNA折纸技术,作为一种典型的DNA纳米技术,已被广泛用于构建从一维带状物到三维具有预定形状的物体的复杂结构。然而,目前的挑战在于如何利用这些技术来实现动态的拓扑转换,从而使得这些人工结构能够像生物系统一样,在信息传递和处理中展现出高度的灵活性和效率。
总结展望
为了解决这一问题,华东师范大学裴昊以及上海交通大学樊春海院士合作在Nature Chemistry期刊上发表了题为“Encoding signal propagation on topology-programmed DNA origami”的最新论文。本研究致力于开发一种系统的分子实现拓扑操作的方法。研究团队通过“粘接-切割”过程,基于多重杂交和链置换反应,设计并构建了一种拓扑编程的DNA折纸系统。这些折纸结构能够通过拓扑操作全局地改变其构形,例如从二维折纸到三维折纸再到二维折纸的连续转换。研究团队利用整数拓扑不变量如亏格、边界组分数量和可定向性来表征这些结构的拓扑特性的变化,并展示了如何通过这些操作实现对结构的动态调整。
具体而言,通过在2D折纸的不同部位粘接DNA链的端部,研究团队成功实现了包含可定向或非定向表面的3D折纸结构。这些结构不仅展示了复杂的拓扑变换,还作为动态支架,用于探索空间和时间上的信号传播。通过空间排列具有反应性的DNA发夹,研究团队演示了在3D折纸的曲面上不同长度、方向和曲率的传输路径上的信号传播。此外,研究团队还利用拓扑变换诱导的链置换级联在接近位置进行分子控制,进一步构建了多功能的可重编程双轨电路,允许在折纸拓扑变换时重新连接局部电路元件,从而启动不同的信号传播路径。  研究亮点
研究亮点
(1)本文首次实现了基于DNA折纸的拓扑编程系统,通过"粘接-切割"过程进行拓扑操作,实现了全局结构的连续变化。
(2)实验通过多重杂交和链置换反应,成功实现了DNA折纸结构的拓扑连续转换,包括从二维折纸到三维折纸再到二维折纸的转变。作者使用整数拓扑不变量如亏格、边界组分数量和可定向性来表征这些拓扑结构的变化,验证了拓扑连续转换的有效性。
(3)此外,作者展示了通过空间排列反应性DNA发夹,在三维折纸的曲面上实现了信号传播路径的可编程控制,跨越不同长度、方向和曲率的传输路径。
(4)进一步地,利用拓扑变换诱导的链置换级联在接近位置进行分子控制,构建了多功能可重编程双轨电路,允许在折纸拓扑变换时重新连接局部电路元件,以实现不同的信号传播路径。这些结果表明,作者的方法为制造能够在可编程控制下执行复杂拓扑转换的动态DNA折纸纳米结构提供了通用途径。
图文解读
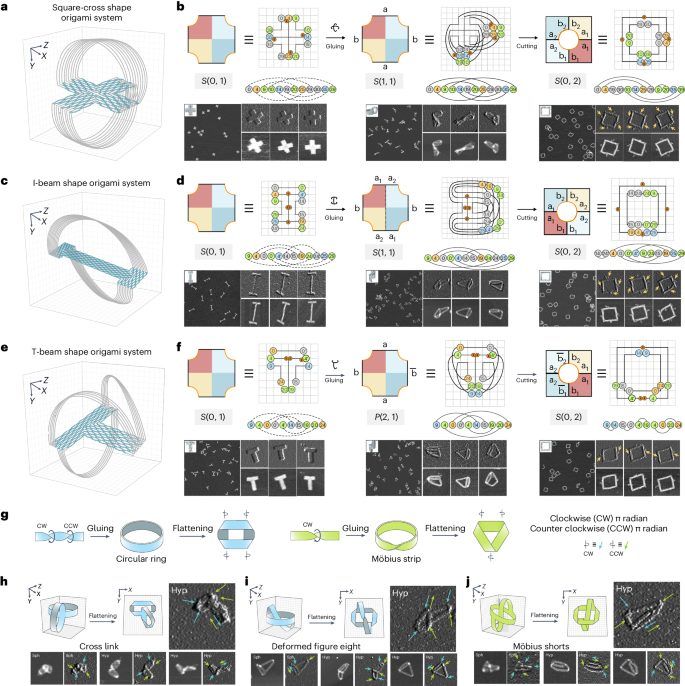 图1:DNA折纸系统的拓扑变换。
图1:DNA折纸系统的拓扑变换。
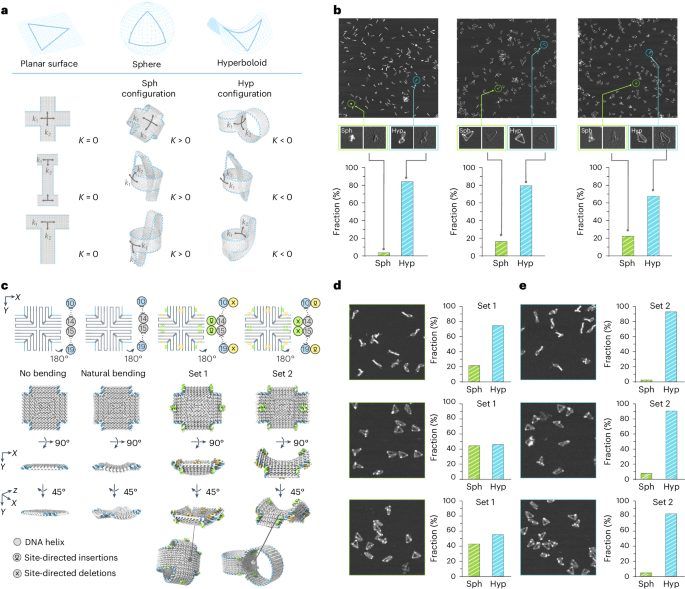 图2. 可重构拓扑DNA折纸的设计和可视化。
图2. 可重构拓扑DNA折纸的设计和可视化。
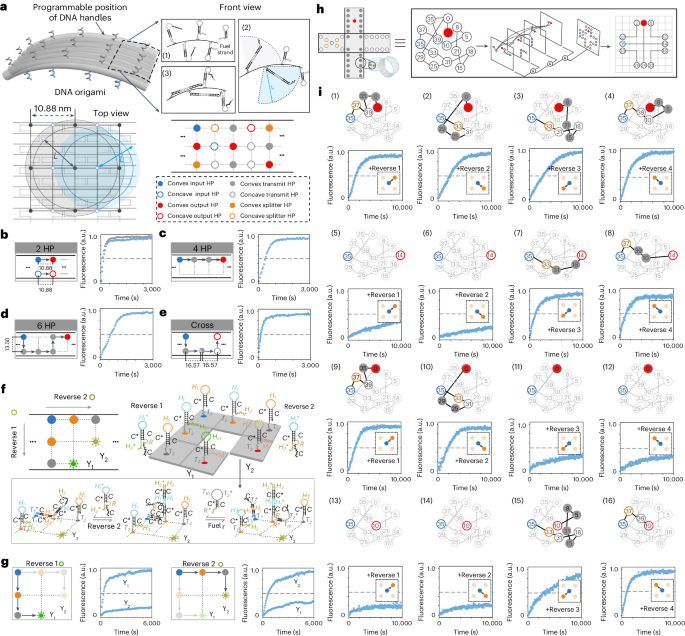 图3. 具有椭球或双曲面配置的三维折纸。
图3. 具有椭球或双曲面配置的三维折纸。
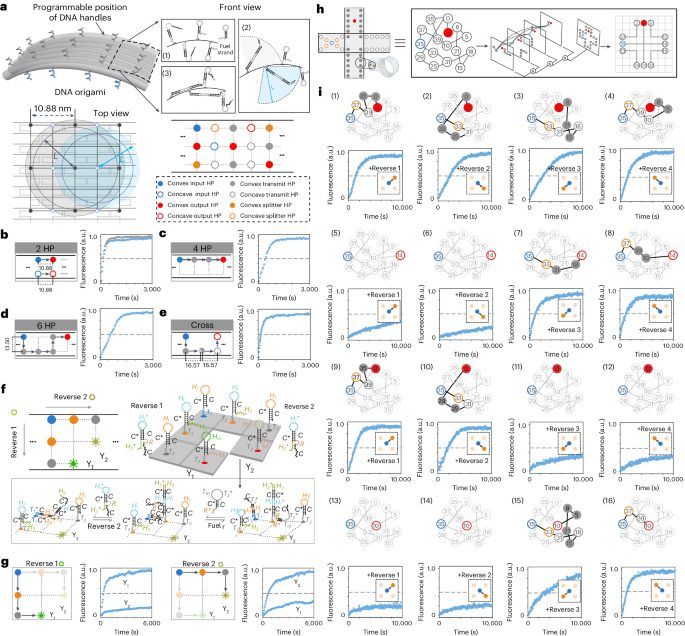 图4:通过曲面上的分子线,实现了信号传播。
图4:通过曲面上的分子线,实现了信号传播。
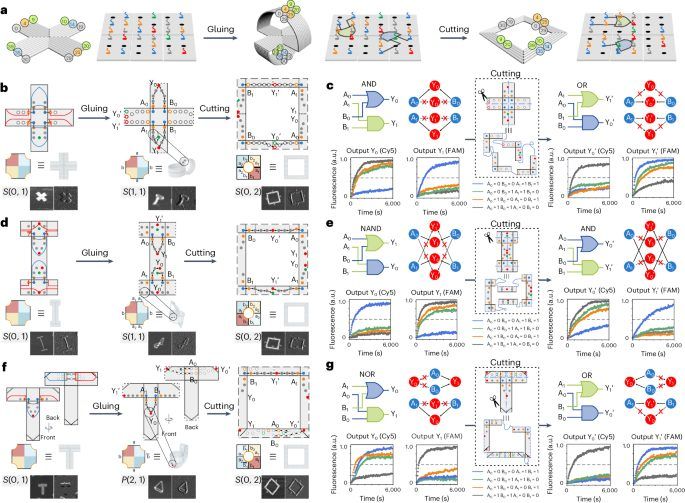 图5:可重构DNA折纸上的多用途双轨门。
图5:可重构DNA折纸上的多用途双轨门。
结论展望
以上研究揭示了利用DNA纳米技术实现复杂拓扑操作的新途径,这对于仿生学和分子计算领域具有重要的价值。通过多重链置换和杂交步骤,研究团队成功地在分子水平上实施了“粘接-切割”过程,从而能够精确控制DNA折纸结构的大小、形状和拓扑特性。这一研究不仅拓展了DNA纳米技术在构建复杂拓扑结构方面的应用,还展示了在控制精准拓扑转换过程中的高度可编程性和可配置性。
此外,通过演示拓扑编程的DNA折纸在分子电路元素排列中的应用,研究为设计和实现多功能逻辑计算提供了新思路。这些成果促进了分子系统的复杂动态行为研究,进一步推动了纳米技术在生物学和信息科学交叉领域的前沿探索,为未来发展具有高度动态和可控性的分子结构提供了理论和实验基础。
文献信息
Ji, W., Xiong, X., Cao, M. et al. Encoding signal propagation on topology-programmed DNA origami. Nat. Chem. (2024). https://doi.org/10.1038/s41557-024-01565-2
























 5万+
5万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








